一种基于句嵌入Infersent模型的蛋白质-蛋白质相互作用预测方法与流程
 2021-01-08 11:01:35|
2021-01-08 11:01:35| 277|
277| 起点商标网
起点商标网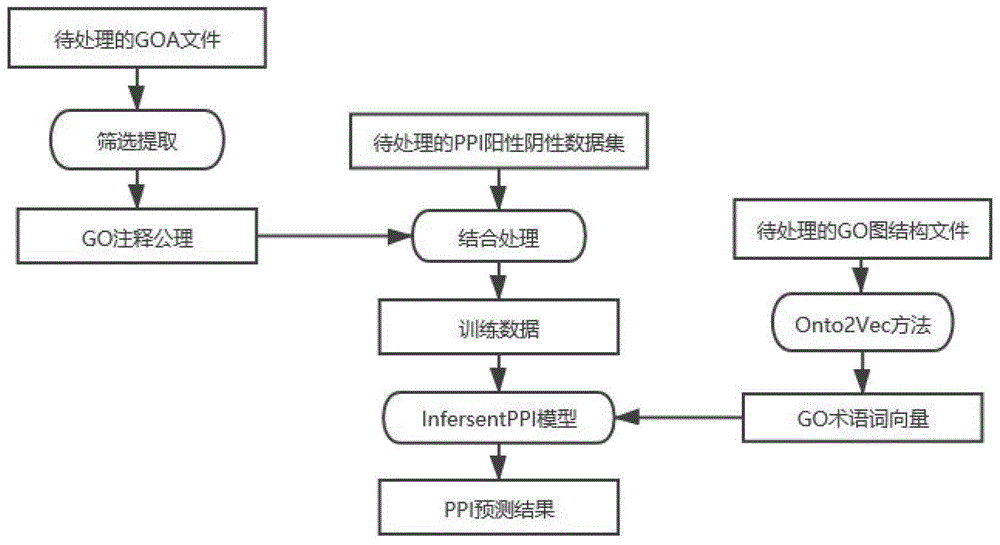
本发明涉及生物信息与自然语言处理领域,尤其涉及一种基于基因本体、句嵌入模型在蛋白质-蛋白质相互作用(ppi)预测领域中的应用。
背景技术:
:蛋白质-蛋白质相互作用(ppi)是许多生物信息学应用程序(例如蛋白质功能和药物发现)的一项基本指标。因此,准确预测蛋白质之间的相互作用将有助于我们理解潜在的分子机制,并显著促进药物的发现。通过基因本体(go)信息可以较为准确的预测出ppi。先前的基因本体信息预测ppi的大多数研究都是利用信息内容(ic)。最近,一些研究利用自然语言处理领域的词嵌入技术去学习代表go术语和蛋白质的向量,以此来预测ppi。基因本体是生物学功能注释的一个标准词汇术语,是一种统一的术语,用于描述跨物种的同源基因及基因产物的功能。本发明利用有监督句嵌入技术捕获go结构与go注释信息以预测ppi。将基因本体与强大的自然语言处理技术结合在一起,即使不使用蛋白质序列信息,我们的方法也提供了一条通用的计算流程来预测蛋白质与蛋白质的相互作用。技术实现要素:本发明的目的在于提供一种基于句嵌入infersent模型的蛋白质-蛋白质相互作用预测方法,其是基于自然语言处理模型infersent结合基因本体(go)预测蛋白质-蛋白质相互作用(ppi)。该方法中go注释公理的每条记录有相应权重;结合go注释公理与go结构公理,在基于句嵌入infersent的模型上训练ppi阳性阴性数据集,最终得到预测ppi的模型。为了达到上述目的,本发明通过以下技术方案实现:一种基于句嵌入infersent的模型预测蛋白质-蛋白质相互作用的方法,包含以下步骤:s1、go的本体被构造成一个图,其中go术语作为图中的节点,go术语之间的关系称为边。使用现有的onto2vec技术,在go图结构文件中提取生成go结构公理,训练go结构公理,得到go术语词向量;s2、筛选提取注释公理:在基因本体论注释(goa)文件中筛选提取有相应权重的每条go注释记录,生成go注释公理;s3、结合步骤s1中的所述go注释公理,将ppi阳性阴性数据集的蛋白质逐行替换为注释它的go术语,得到最终的训练数据;s4、将infersent模型改造成infersentppi模型,结合步骤s2中的所述go术语词向量,在infersentppi模型上对步骤s3中的所述训练数据进行迭代训练,最终得到预测ppi的模型,输出ppi预测结果。优选地,所述步骤s1进一步包含以下步骤:s1.1、提取出go.owl文件中的go图结构记录,每条go图结构记录由多个go唯一标识码与其关系词组成,go图结构记录组织成文件,得到go结构公理文件;s1.2、将步骤s1.1中的所述go结构公理文件逐行输入word2vec的skip-gram模型;s1.3、在skip-gram模型中进行训练,如下:给定一个序列的训练单词x1,x2,.....,x3,skip-gram模型的目的是最大化下列公式:其中c是训练上下文窗口的大小,t是训练词集合的大小,wi是序列中的第i个训练词;s1.4、训练结束得到go术语的词向量组织成文件输出;优选地,所述步骤s2进一步包含以下步骤:s2.1、根据待处理基因本体论注释(goa)文件的evidencecode字段内容,对goa的每条记录进行筛选,删除evidencecode字段内容为‘iea’或’nd’的记录,得到筛选后的goa文件,提取出筛选后的goa文件的每一行记录的uniprotkb唯一标识码与go唯一标识码,得到go注释记录文件,go注释记录文件中重复的记录不删除,重复的次数代表这条注释记录的有效引用的数量,可作为对应注释记录的权重;s2.2、提取步骤s1.2中的所述go注释记录文件的相同uniprotkb唯一标识码以及对应的所有go唯一标识码,将其集中在同一行,组织成文件,得到go注释公理文件;优选地,所述步骤s3进一步包含以下步骤:s3.1、提取出蛋白质-蛋白质相互作用(ppi)阳性阴性数据集每一行记录的一对蛋白质,映射为两个uniprotkb唯一标识码,无法映射为uniprotkb唯一标识码的蛋白质将其所在的蛋白质对进行删除,根据数据集的性质生成对应蛋白质对的属性标签’positive’或’negative’,蛋白质对与属性标签组织成ppi记录文件,该ppi记录文件中每一行的内容是由两个uniprotkb唯一标识码与属性标签组成;s3.2、利用步骤s1中的所述基因本体注释公理,对步骤s3.1中的所述ppi记录文件的蛋白质逐行替换为注释它的go唯一标识码,得到训练模型的ppi语料库;s3.3、步骤s1中的所述ppi语料库,随机选取80%、10%、10%作为训练集、验证集、测试集,作为最终的训练数据。优选地,所述步骤s4进一步包含以下步骤:s4.1、基于infersent模型进行改造,其中infersent模型的句子编码器设置为卷积神经网络,infersent模型的分类器设置为二分类,二分类的标签为’positive’与’negative’,得到infersentppi模型;s4.2、结合步骤s2中的所述go术语的词向量,在步骤s4.1中的所述infersentppi模型中对步骤s3中的所述训练数据进行迭代训练;优选地,所述步骤s4.2中的迭代训练包含以下步骤:s4.2-1、训练数据的训练集按行提取的两个集合的go唯一标识码作为句子a与句子b分别输入两个句子编码器,句子编码器使用的词向量为go术语的词向量,句子编码器使用卷积神经网络,生成的句向量u与句向量v就是蛋白质向量u与蛋白质向量v;s4.2-2、利用步骤s4.1中的所述句向量u与句向量v,计算u和v的首尾相连得到(u,v)、计算u和v相乘得到u*v、计算u和v相减得到|u-v|,最后将得到的(u,v,u*v,|u-v|)结果送入一个2分类的分类器,分类器由多个全连接层和一个softmax层组成,最终得到步骤s4.2-1中的所述句子a和句子b的标签’positive’与’negative’的概率分布预测值;s4.2-3、使训练集的标签与步骤s4.2-2中的所述标签’positive’与’negative’的概率分布预测值的误差其最小化;s4.2-4、重复步骤s4.2-1到s4.2-3,直到所有训练集的数据迭代完一次;s4.2-5、预测ppi的公式如下:infersentppi(a,b)=p(positive)>p(negative)?positive:negatives4.2-6、在验证集上进行预测,若验证集结果比上一次验证集结果差则停止训练,不保存模型,若验证集结果比上一次验证集结果好,则保存模型,并调整学习率,当学习率低于设置的最小学习率时停止训练,当学习率高于参数设置的最小学习率时重复步骤s4.2-1到s4.2-4继续下一轮迭代训练,迭代次数达到参数设置的最大迭代次数时,停止训练;s4.3、迭代训练结束,得到了效果最好的预测ppi的模型;s4.4、步骤s4.3中的所述预测ppi的模型在测试集上进行预测,将测试集的预测结果组织成文件输出;优选地,步骤s4.1中,所述二分类’positive’是代表ppi阳性,所述二分类’negative’是代表ppi阴性,步骤s4.3中,所述预测ppi是指被基因本体注释过的蛋白质的ppi预测。与现有技术相比,本发明的有益效果在于:本发明提供的基于句嵌入infersent的模型预测蛋白质-蛋白质相互作用的方法,借助自然语言处理模型infersent,结合基因本体论,有效地提升了ppi预测的准确率与auc。附图说明图1是本发明的工作总流程图,分为4个模块:onto2vec、筛选提取注释公理、结合处理和infersentppi;图2是本发明的onto2vec生成go向量的具体实施方法;图3是本发明的筛选提取注释公理的流程示意图;图4是本发明的infersentppi模型的具体实施方法;图5是本发明的infersentppi模型的句子编码器的具体实施方法;具体实施方式为使本发明实施例的目的、技术方案和优点更加清楚,下面将结合本发明实施例中的附图,对本发明实施例中的技术方案进行清楚、完整地描述,显然,所描述的实施例仅仅是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。如图1-图5所示,本发明提供了一种基于句嵌入infersent的模型预测蛋白质-蛋白质相互作用的方法(下述以ppi阳性阴性数据为例进行详细说明),该方法包括如下步骤:步骤s1、go的本体被构造成一个图,其中go术语作为图中的节点,go术语之间的关系(也称为对象属性)称为边,go常规信息在go.owl文件中可得到。使用现有的onto2vec技术,在go图结构文件go.owl中提取生成go结构公理,训练go结构公理,得到go术语词向量,其中go结构公理为go术语(代表每个方面的根术语除外)与另一个go术语具有子类关系的描述组成;步骤s2、筛选提取注释公理:在基因本体论注释(goa)文件中筛选提取有相应权重的每条go注释记录,生成go注释公理;步骤s3、结合步骤s1中的所述go注释公理,将ppi阳性阴性数据集的蛋白质逐行替换为注释它的go术语,得到最终的训练数据;步骤s4、将infersent模型改造成infersentppi模型,结合步骤s2中的所述go术语词向量,在infersentppi模型上对步骤s3中的所述训练数据进行迭代训练,最终得到预测ppi的模型,输出ppi预测结果。如图2所示,所述步骤s1进一步包含以下步骤:s1.1、提取出go.owl文件中的go图结构记录,每条go图结构记录由多个go唯一标识码与go术语之间的关系词(例如subclassof,disjointwith)组成,go图结构记录组织成文件,得到go结构公理文件,具体示例如表1所示:表1是go结构公理文件的内容示例s1.2、将步骤s1.1中的所述go结构公理文件逐行输入word2vec的skip-gram模型;s1.3、在skip-gram模型中进行训练,如下:给定一个序列的训练单词x1,x2,...,xt,skip-gram模型的目的是最大化下列公式:其中c是训练上下文窗口的大小,t是训练词集合的大小,wi是序列中的第i个训练词;s1.4、训练结束得到go术语的词向量组织成文件输出;如图3所示,所述步骤s2进一步包含以下步骤:s2.1、根据待处理基因本体论注释(goa)文件的evidencecode字段内容,对goa的每条记录进行筛选。evidencecode是go注释的有效证据代码,删除evidencecode字段内容为‘iea’或’nd’的记录,得到筛选后的goa文件。当无法获得有关被注释的基因或基因产物的分子功能、生物学过程或细胞成分的信息时,nd证据代码用于注释。iea支持的注释最终基于同源性和/或其他实验或序列信息,但通常无法追溯到实验来源。提取出筛选后的goa文件的每一行记录的uniprotkb唯一标识码与go唯一标识码,得到go注释记录文件,go注释记录文件中重复的记录不删除,重复的次数代表这条注释记录的有效引用的数量,可作为对应注释记录的权重;具体示例如表2表3所示:表2是goa文件的内容示例表3是go注释记录文件的内容示例uniprotkbidrelationgoida2p2r3hasfunctiongo:0006047d6vtk4hasfunctiongo:0000750d6vtk4hasfunctiongo:0000750s2.2、提取步骤s1.2中的所述go注释记录文件的相同uniprotkb唯一标识码以及对应的所有go唯一标识码,将其集中在同一行,组织成文件,得到go注释公理文件;具体示例如表4所示:表4是go注释公理文件的内容示例uniprotkbidgoida2p2r3go:0006002;go:0006047d6vtk4go:0000750;go:0000750所述步骤s3进一步包含以下步骤:s3.1、提取出蛋白质-蛋白质相互作用(ppi)阳性阴性数据集每一行记录的一对蛋白质,映射为两个uniprotkb唯一标识码,无法映射为uniprotkb唯一标识码的蛋白质将其所在的蛋白质对进行删除,根据数据集的性质生成对应蛋白质对的属性标签’positive’或’negative’,’positive’指的是ppi阳性、’negative’指的是ppi阴性。蛋白质对与属性标签组织成ppi记录文件,该ppi记录文件中每一行的内容是由两个uniprotkb唯一标识码与属性标签组成;具体示例如表5所示:表5是ppi记录文件的内容示例proteinaproteinbtagp16649p14922positivep07269p22035positivep53248p32366negativeq08558p31412negativeq06169p41807negatives3.2、利用步骤s1中的所述基因本体注释公理,对步骤s3.1中的所述ppi记录文件的蛋白质逐行替换为注释它的go唯一标识码,得到训练模型的ppi语料库;具体示例如表6所示:表6是ppi语料库的内容示例s3.3、步骤s1中的所述ppi语料库,随机选取80%、10%、10%作为训练集、验证集、测试集,作为最终的训练数据。所述步骤s4进一步包含以下步骤:s4.1、基于infersent模型进行改造,其中infersent模型的句子编码器设置为卷积神经网络,infersent模型的分类器设置为二分类,二分类的标签为’positive’与’negative’,得到infersentppi模型;s4.2、结合步骤s2中的所述go术语的词向量,在步骤s4.1中的所述infersentppi模型中对步骤s3中的所述训练数据进行迭代训练;如图4所示,所述步骤s4.2中的迭代训练包含以下步骤:s4.2-1、训练数据的训练集按行提取的两个集合的go唯一标识码作为句子a与句子b分别输入两个句子编码器,句子编码器使用的词向量为go术语的词向量,句子编码器使用卷积神经网络,如图5所示,生成的句向量u与句向量v就是蛋白质向量u与蛋白质向量v;s4.2-2、利用步骤s4.1中的所述句向量u与句向量v,计算u和v的首尾相连得到(u,v)、计算u和v相乘得到u*v、计算u和v相减得到|u-v|,最后将得到的(u,v,u*v,|u-v|)结果送入一个2分类的分类器,分类器由多个全连接层和一个softmax层组成,最终得到步骤s4.2-1中的所述句子a和句子b的标签’positive’与’negative’的概率分布预测值;s4.2-3、使训练集的标签与步骤s4.2-2中的所述标签’positive’与’negative’的概率分布预测值的误差其最小化;s4.2-4、重复步骤s4.2-1到s4.2-3,直到所有训练集的数据迭代完一次;s4.2-5、预测ppi的公式如下:infersentppi(a,b)=p(positive)>p(negative)?positive:negative例如:infersentppi的一次输入为句子a和句子b,及蛋白质a和蛋白质b的uniprotkb唯一标识码:句子a:p16649;句子b:p14922;然后根据步骤s3.2将蛋白质逐行替换为注释它的go唯一标识码,得到测试数据是单词集gos1,单词集gos2:gos1:{go_0000329,go_0005739,go_0005739,go_0006623,go_0022857,go_0055085}gos2:{go_0005783,go_0006633,go_0006892,go_0009922,go_0009922,go_0019367,go_0030148,go_0030148,go_0030176,go_0030497,go_0032511,go_0034625,go_0034626,go_0042761,go_0042761}最后根据步骤s4.2中的公式计算文本a和b的p(positive),p(negative)为0.724和0.276,计算得到infersentppi(a,b)=positive.s4.2-6、在验证集上进行预测,若验证集结果比上一次验证集结果差则停止训练,不保存模型,若验证集结果比上一次验证集结果好,则保存模型,并调整学习率,当学习率低于设置的最小学习率时停止训练,当学习率高于参数设置的最小学习率时重复步骤s4.2-1到s4.2-4继续下一轮迭代训练,迭代次数达到参数设置的最大迭代次数时,停止训练;s4.3、迭代训练结束,得到了效果最好的预测ppi的模型;s4.4、步骤s4.3中的所述预测ppi的模型在测试集上进行预测,将测试集的预测结果组织成文件输出,参数batch_size=2训练的模型,在测试集上的预测效果最好;步骤s4.1中,所述二分类’positive’是代表ppi阳性,所述二分类’negative’是代表ppi阴性,步骤s4.3中,所述预测ppi是指被基因本体注释过的蛋白质的ppi预测。综上所述,本发明提供的基于句嵌入infersent的模型预测蛋白质-蛋白质相互作用的方法,借助自然语言处理模型infersent,结合基因本体论,有效地提升了ppi预测的准确率与auc。本发明不仅能够应用于蛋白质,对于其他被本体注释的实例同样可以按此方法实施。此外,自然语言处理模型infersent的句子编码器也是可以替换的,不会影响整体模型的实施。使用者可以根据需求选择合适的句子编码器。本领域普通技术人员可以理解:附图只是一个实施例的示意图,附图中的模块或流程不一定是实施本发明所必须的。尽管本发明的内容已经通过上述优选实施例作了详细介绍,但应当认识到上述的描述不应被认为是对本发明的限制。在本领域技术人员阅读了上述内容后,对于本发明的多种修改和替代都将是显而易见的。因此,本发明的保护范围应由所附的权利要求来限定。当前第1页1 2 3
起点商标作为专业知识产权交易平台,可以帮助大家解决很多问题,如果大家想要了解更多知产交易信息请点击 【在线咨询】或添加微信 【19522093243】 与客服一对一沟通,为大家解决相关问题。
与客服一对一沟通,为大家解决相关问题。
此文章来源于网络,如有侵权,请联系删除
 热门咨询
热门咨询

tips


 商标分类
商标分类  商标转让
商标转让 



