盐角草SeSMT1蛋白及其编码基因及应用的制作方法
 2021-02-02 18:02:01|
2021-02-02 18:02:01| 497|
497| 起点商标网
起点商标网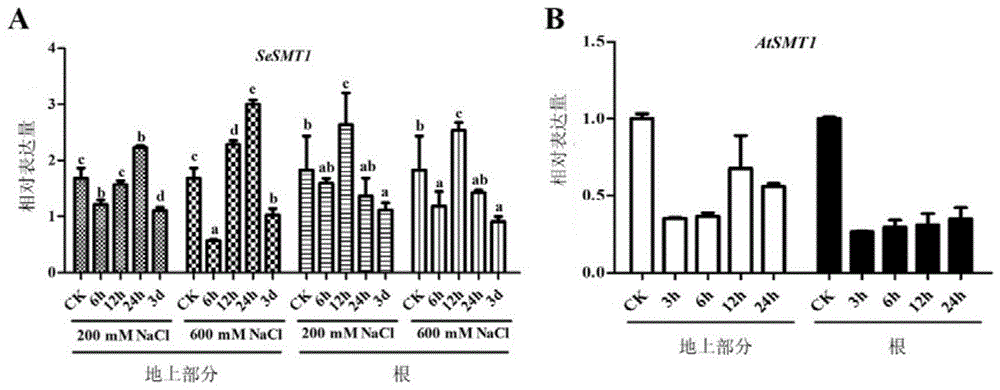
本发明属于生物技术领域,尤其涉及一种盐角草sesmt1蛋白及其编码基因及应用。
背景技术:
土壤盐渍化是农业生态环境退化的重要原因之一,也是限制农作物产量的主要环境因素(sharmaetal.,2016)。据联合国教科文组织(unesco)和粮农组织(fao)不完全统计,全世界每年有一千万公顷的土地变成盐碱地。研究植物响应盐胁迫的机制、创建耐盐作物新品种、充分利用盐碱地资源是实现农业可持续发展的重要战略。
氮素是植物生长发育过程中的必须大量元素,既是合成蛋白质的基础物质,也是生理代谢过程中的催化物质(林郑和等,2011)。土壤中有效氮含量不足会导致农作物严重减产,而盐渍化土壤中有效氮含量较低,因此研究提高植物在盐渍环境中的氮素利用效率对于培育氮高效作物具有指导意义。
不同于甜土植物,盐生植物在高盐环境下仍能维持正常生长,同时保证生物量和结实量(boyko,1967)。植物响应外界盐胁迫的机制主要包括:通过膜蛋白与转运蛋白调节离子选择性吸收和外排及离子区隔化;通过细胞骨架动态重塑维持正常的细胞结构、物质运输与信息传递;通过转录、翻译与翻译后修饰调节蛋白质动态变化与相互作用;通过调控基础代谢与次生代谢水平保证细胞结构与代谢正常;利用抗氧化系统解毒和积累渗透保护物等(赵琪等,2012)。
植物质膜是细胞感知外界盐信号的关键结构,盐胁迫可以引起植物质膜中的脂类物质的组分和含量发生变化,同时改变质膜的结构特性,即发生质膜重塑(chalbietal.,2014)。质膜重塑使质膜物理结构发生相应改变,影响膜的侧向流动速率、膜上效应分子的功能以及膜蛋白正常功能的实现(meeretal.,2008;zauberetal.,2014),而植物对氮素的吸收依赖于质膜上的转运蛋白。因此,植物的质膜重塑模式与其耐盐性和氮素吸收利用效率紧密相关。
质膜的主要脂质组成包括磷脂、甾醇和脂肪酸。其中,甾醇作为生物膜系统的重要组分,能够调控质膜的流动性和通透性,也是形成脂筏的重要组分。同时,甾醇也是油菜素内酯等激素的前体,在植物生长发育和响应胁迫中发挥重要作用(何小钊等,2013)。
植物甾醇与动物甾醇结构相似,主要区别在于其24位碳原子含有甲基或乙基侧链基团,即24-甲基甾醇(菜油甾醇)或24-乙基甾醇(谷甾醇、豆甾醇),这主要是由依赖于腺苷甲硫氨酸(s-adenosyl-l-methionine,sam)的c-24甾醇甲基转移酶(sterolmethyltransferase,smt)(ec2.1.1.41)在环阿屯醇途径和羊毛甾醇途径发生催化作用而形成的(valitovaetal.,2016)。smt包括smt1和smt2两个家族,其中smt1家族成员是植物甾醇合成通路上的重要分支点,能够催化环阿屯醇生成24-亚甲基-4-甲基-7-烯-胆甾烷醇,使24位碳加上第一个烷基,从而开始合成植物甾醇,并与合成胆固醇的支路分开。
目前,人们已经成功地从多种植物中分离到smt1的cdna,包括大豆(gmsmt1,shietal.,1996),烟草(ntsmt1,bouvier-navéetal.,1998),拟南芥(atsmt1,dieneretal.,2000),狗牙根(cdsmt1,chenetal.,2018)和南非醉茄(wssmt1,paletal.,2019)等。这些smt1基因在植物生长发育中发挥着重要功能,而在抗逆方面的研究缺乏。
盐角草(salicorniaeuropaeal.)是藜科真盐生植物,能够耐受高达1000mmnacl,是研究植物耐盐机制的优良材料之一;同时也是富含蛋白质的饲料作物、油料作物和观赏植物(venturaandsagi,2012)。
技术实现要素:
本发明一个目的是提供一种蛋白。
本发明提供的蛋白,为sesmt1a蛋白或sesmt1b蛋白,如下(1)或(2)或(3):
(1)由序列表中序列2所示的氨基酸序列组成的蛋白质;
(2)由序列表中序列4所示的氨基酸序列组成的蛋白质;
(3)将序列表中序列2或序列4所示的氨基酸序列经过一个或几个氨基酸残基的取代和/或缺失和/或添加且来源于盐角草具有相同功能的由(1)衍生的蛋白质。
编码上述蛋白的核酸分子也是本发明保护的范围。
上述核酸分子是如下1)-6)中任一种的dna分子:
1)编码区为序列表中序列1所示的dna分子;
2)编码区为序列表中序列7第17-1074位所示的dna分子;
3)编码区为序列表中序列3所示的dna分子;
4)编码区为序列表中序列8第17-1074位所示的dna分子;
5)在严格条件下与1)-4)中任一限定的dna序列杂交且编码具有相同功能蛋白质的dna分子;
6)与1)-4)中任一限定的dna序列至少具有70%、至少具有75%、至少具有80%、至少具有85%、至少具有90%、至少具有95%、至少具有96%、至少具有97%、至少具有98%或至少具有99%同源性且来源于盐角草编码具有相同功能蛋白质的dna分子。
含有上述核酸分子的重组载体、表达盒或重组菌也是本发明保护的范围。
在本发明的实施例中,重组载体为pgwb651-35s::sesmt1a和pgwb502ω-35s::sesmt1a或pgwb651-35s::sesmt1b和pgwb502ω-35s::sesmt1b;
pgwb651-35s::sesmt1a融合载体为将序列3第17-1071所示的dna片段替换pgwb651载体的attr1和attr2位点间的片段得到的,由camv35s启动子驱动sesmt1a表达。
pgwb502ω-35s::sesmt1a融合载体为将序列7第17-1074位所示的dna片段替换pgwb502ω载体的attr1和attr2位点间的片段得到的,由camv35s启动子驱动sesmt1a表达。
pgwb651-35s::sesmt1b融合载体为将序列3第17-1071所示的dna片段替换pgwb651载体的attr1和attr2位点间的片段得到的,由camv35s启动子驱动sesmt1b表达。
pgwb502ω-35s::sesmt1b融合载体为将序列8第17-1074位所示的dna片段替换pgwb502ω载体的attr1和attr2位点间的片段得到的,由camv35s启动子驱动sesmt1b表达。
上述蛋白、上述核酸分子或上述的重组载体、表达盒或重组菌在调控植物耐逆性中的应用也是本发明保护的范围;
或,上述蛋白、上述核酸分子或上述的重组载体、表达盒或重组菌在提高植物在盐渍环境中利用氮素的效率中的应用也是本发明保护的范围;
或,上述蛋白、上述核酸分子或上述的重组载体、表达盒或重组菌在培育高耐逆性植物中的应用也是本发明保护的范围。
atsmt1蛋白或其编码核酸分子或含有所述核酸分子的重组载体、表达盒或重组菌在调控植物耐逆性中的应用也是本发明保护的范围;
或,atsmt1蛋白或其编码核酸分子或含有所述核酸分子的重组载体、表达盒或重组菌在提高植物在盐渍环境中利用氮素的效率中的应用也是本发明保护的范围;
或,atsmt1蛋白或其编码核酸分子或含有所述核酸分子的重组载体、表达盒或重组菌在培育高耐逆性植物中的应用也是本发明保护的范围;
所述atsmt1蛋白的氨基酸序列为如下:
(1)序列表中序列9第27-1037位编码的氨基酸残基组成的序列;
(2)将(1)所示的氨基酸序列经过一个或几个氨基酸残基的取代和/或缺失和/或添加且来源于拟南芥具有相同功能的由(1)衍生的蛋白质。
所述atsmt1蛋白编码核酸分子为如下任一种:
1)编码区为序列表中序列9第27-1037位所示的dna分子;
2)编码区为序列表中序列9第17-1044位所示的dna分子;
3)在严格条件下与1)或2)限定的dna序列杂交且编码具有相同功能蛋白质的dna分子;
4)与1)或2)限定的dna序列至少具有70%、至少具有75%、至少具有80%、至少具有85%、至少具有90%、至少具有95%、至少具有96%、至少具有97%、至少具有98%或至少具有99%同源性且来源于盐角草编码具有相同功能蛋白质的dna分子。
上述应用中,所述耐逆性为耐盐性或耐低氮性。低氮具体为n浓度在1mm以下。
上述应用中,所述耐逆性为耐盐性或耐低氮性。
本发明另一个目的是提供一种培育耐逆性提高的转基因植物的方法。
本发明提供的方法,为如下1)或2):
1)所述的方法包括如下步骤:提高目的植物中上述蛋白的含量和/或活性,得到转基因植物;
2)所述的方法包括如下步骤:提高目的植物中编码上述蛋白的核酸分子的表达,得到转基因植物;
所述转基因植物的耐受性高于所述目的植物。
上述方法中,所述提高目的植物中上述蛋白的含量和/或活性,或,提高目的植物中编码上述蛋白的核酸分子的表达,均通过上述核酸分子导入目的植物实现。
上述方法中,所述耐逆性为耐盐性或耐低氮性。
本工作首次从真盐生植物盐角草中克隆到了编码c-24甾醇甲基转移酶1的基因(命名为sesmt1),发现该基因存在两种序列,分别标示为sesmt1a和sesmt1b。二者cds全长均为1041bp,编码346个氨基酸。该氨基酸序列具有smt蛋白的两个保守结构域。进化分析预测显示,sesmt1与菠菜sosmt1同源性最高。蛋白亚细胞定位显示,sesmt1定位于内质网。在盐胁迫处理24h时,盐角草sesmt1基因在转录水平的表达量明显升高,与拟南芥atsmt1表达量变化趋势相反。将sesmt1a和sesmt1b分别转入拟南芥,对转基因拟南芥的抗盐性和耐低氮能力分析发现,与野生型相比,在盐胁迫下,过表达植株地上部分、侧根数、叶绿素a/叶绿素b比值提高;在低氮条件下,地上部分、侧根数、叶绿素含量增多;在低氮加盐条件下过表达植株地上部分鲜重显著增加,sesmt1b过表达株系侧根相比其他株系也显著增多。表明该序列编码的蛋白在盐角草中发挥的功能强于另一种,并且比拟南芥同源基因在盐渍环境中利用氮素的效率更高。这些结果表明,sesmt1基因过表达能提高耐盐和低氮的能力,为培育抗盐及氮高效作物提供了理论基础和基因资源。此外,鉴于植物甾醇在降血压血脂、提高免疫力、预防或减轻癌症和阿兹海默症等方面的作用,对盐角草甾醇合成途径关键基因的克隆及功能解析也可为利用盐生植物反应器大规模生产植物甾醇提供新的思路。
在以往拟南芥等大多数植物的研究中,smt1基因往往只有一种序列的报道。而此次盐生植物盐角草中克隆得到了smt1基因的两种序列,并且发现二者在功能上存在差异。目前研究中报道的催化24-乙基甾醇的生成的smt2被认为是植物中smt1在进化过程中变异产生的酶,与此类似,smt1基因的两种序列很有可能也是植物在物种演化过程中为适应环境的变化而逐步形成的。这种基因存在多种序列的现象可能是植物在适应环境变化中进化出的机制,能够调控基因表达和最终蛋白生成量。盐角草属于盐沼植物,生长于沿海滩涂潮上带以及内陆盐沼等极端盐渍化环境,而多拷贝的存在为基因发挥功能适应环境变化提供了保障,这也提示了植物从水生向陆生进化过程中可能发生的变化。
综上,盐角草sesmt1可能通过侧根的发育吸收更多养分补给地上部分,并通过多种序列存在的方式提高转录和翻译水平最终积累蛋白量,使得植物本身能够同时响应低氮和盐胁迫,从而在盐渍土壤上实现氮的高效利用。盐角草sesmt1基因及功能解析有望在作物抗逆遗传育种中发挥作用,为培育抗盐及氮高效作物提供基因资源和理论支持;此外,作为植物甾醇和胆固醇合成通路分支点上的关键酶,该基因的表达能够通过基因工程调控植物甾醇的合成与积累,为利用生物反应器生产提炼甾醇提供指导。
附图说明
图1为盐角草和拟南芥smt1在nacl处理下的表达模式。
图2为sesmt1蛋白的亚细胞定位研究。
图3为smt1在各拟南芥转基因株系的转录本表达情况。
图4为拟南芥野生型和smt1转基因株系对nacl和低氮的耐受能力。
具体实施方式
下述实施例中所使用的实验方法如无特殊说明,均为常规方法。
下述实施例中所用的材料、试剂等,如无特殊说明,均可从商业途径得到。
实施例1、sesmt1基因的克隆
一、sesmt1基因全长cds克隆及生物信息学分析
利用trizol试剂(takala公司,9109)小量提取盐角草(盐角草种子购自江苏省大丰市晶隆海洋产业发展有限公司)地上部分总rna,并利用全式金公司的反转录酶获得第一链cdna。
分别设计正向引物sesmt1-f(5'-atgtcaaaatctggagcatttg-3'),和反向引物sesmt1-r(5'-ttaattgctatctgaatcaggctt-3')。
以盐角草cdna为模板,用easypfudnapolymerase(全式金,北京)通过两轮pcr扩增全长cdna。具体反应条件如下:第一轮pcr反应95℃,5min;4cycles:95℃,30sec,46℃,30sec,72℃,1min30sec;5cycles:95℃,30sec,47℃,30sec,72℃,1min30sec;5cycles:95℃,30sec,48℃,30sec,72℃,1min30sec;18cycles:95℃,30sec,49℃,30sec,72℃,1min30sec;72℃,10min。取第一轮pcr产物作为模板进行第二轮pcr,反应条件与第一轮相同。
将得到的pcr产物连入zt4simple-blunt载体(庄盟生物,北京),得到sesmt1-pzt4载体,进行测序确认。
测序结果表明,扩增得到两种序列,全长均为1041bp,均编码了346个氨基酸,二者碱基序列相似性为99.23%,氨基酸序列相似性为98.84%。将其分别标示为sesmt1a和sesmt1b,所编码的蛋白分别标示为sesmt1a蛋白和sesmt1b蛋白。
sesmt1a的核苷酸序列为序列表中序列1;其编码的蛋白sesmt1a的氨基酸序列为序列表中序列2;含有sesmt1a的载体命名为sesmt1a-pzt4载体;
sesmt1b的核苷酸序列为序列表中序列3;其编码的蛋白sesmt1b的氨基酸序列为序列表中序列4;含有sesmt1b的载体命名为sesmt1b-pzt4载体。
二、盐处理下基因表达分析
盐处理:将盐角草种子播种于7cm×7cm的花盆中(营养土:蛭石,1:1),萌发一周后开始用1/2hoagland营养液浇灌,每周一次。萌发四周后用含有0、200和600mmnacl的1/2hoagland营养液浇灌,分别取处理0、6h、12h、24h、3d时间的地上部分和根材料,用于基因表达检测。
使用trizol试剂提取总rna,反转录试剂盒(全式金,北京)反转录后,利用thunderbird
荧光定量qrt-pcr中所用引物序列如下:
盐角草内标tubulin引物:
setubulin-qpcr-f:5'-cagtgcctttgagccatcttc-3'
setubulin-qpcr-r:5'-ctgaatggttcgcttggtctt-3'
盐角草sesmt1基因引物:
sesmt1-qpcr-f:5'-ctggtgttggcggaaagatcgagaa-3'
sesmt1-qpcr-r:5'-gtgctcatggcgtttgatgctctca-3'
荧光定量qrt-pcr检测结果如图1所示,数据为平均值±se,n=3,误差线上不同字母表示相同部位表达量在p<0.05时有显著差异;a.盐角草sesmt1基因在nacl处理下的表达模式,b.拟南芥atsmt1基因在nacl处理下的表达模式,原始数据来源于arabidopsisefp数据库;盐角草在最适条件(200mmnacl)和胁迫条件(600mmnacl)处理下smt1基因在地上部分和根中表达量均呈现先下降后上升最终显著下降的趋势(图1a);拟南芥在胁迫条件(150mmnacl)下,atsmt1在地上部分和根中表达量下降(图1b)。结果显示在24h时,最适盐条件和盐胁迫下盐角草中sesmt1的表达量与对照相比显著上升,而盐胁迫下拟南芥中atsmt1表达量下降明显。这表明不同于拟南芥atsmt1受盐诱导后的下调表达模式,24h时盐角草sesmt1受盐诱导上调表达。
三、sesmt1蛋白亚细胞定位
1、蛋白亚细胞定位载体构建
选用pentrytmdirectionaltopo(invitrogen,k240020)入门载体,设计引物noti-sesmt1-651-entry-f和asci-sesmt1-651-entry-r。
盐角草sesmt1基因引物:
noti-sesmt1-651-entry-f:
5'-ataagaatgcggccgcccccttcaccatgtcaaaatctggagcatttg-3'
asci-sesmt1-651-entry-r:
5'-aggcgcgcccacccttattgctatctgaatcaggctt-3'
分别以上述一种中的sesmt1a-pzt4或sesmt1b-pzt4质粒作为模板,noti-sesmt1-651-entry-f和asci-sesmt1-651-entry-r作为引物,使用easypfudnapolymerase(全式金,北京)进行pcr扩增,得到1080bppcr产物。
sesmt1a-pzt4质粒的扩增产物的核苷酸序列为序列5,其中序列5第27-1064位为去除终止密码子的sesmt1a核苷酸序列,序列5第17-26为上游同源臂,第1065-1071为下游同源臂;
sesmt1b-pzt4质粒的扩增产物的核苷酸序列为序列6,其中序列6第27-1064位为去除终止密码子的sesmt1b核苷酸序列,序列6第17-26为上游同源臂,第1065-1071为下游同源臂;
将回收的pcr产物与入门载体pentrytmdirectionaltopo通过t4连接酶(全式金公司,fl101-01)连接,构建pentrytmd-topo-sesmt1载体,进行测序确认。将pentrytmd-topo-sesmt1质粒与gfp载体pgwb651(参考文献“nakamuras,manos,tanakay,etal.(2010)gatewaybinaryvectorswiththebialaphosresistancegene,bar,asaselectionmarkerforplanttransformation.bioscience,biotechnology,andbiochemistry,74:1315-1319.”)通过同源重组酶gatewaytmlrclonasetmⅱenzymemix(invitrogen,11791-020)相连接,构建了pgwb651-35s::sesmt1a融合载体和pgwb651-35s::sesmt1b融合载体。
对融合表达载体进行酶切验证,采用ecorⅴ和smaⅰ酶切,得到2518bp和9147bp产物,采用xbaⅰ和salⅰ酶切得到2582bp和9083bp产物。2对酶切结果均验证了基因片段正确插入载体。
pgwb651-35s::sesmt1a融合载体为将序列5第17-1071所示的dna片段替换pgwb651载体的attr1和attr2位点间的片段得到的,由camv35s启动子驱动sesmt1a表达。
pgwb651-35s::sesmt1b融合载体为将序列6第17-1071所示的dna片段替换pgwb651载体的attr1和attr2位点间的片段得到的,由camv35s启动子驱动sesmt1b表达。
2、蛋白亚细胞定位
将pgwb651-35s::sesmt1a融合载体或pgwb651-35s::sesmt1b融合载体转入c58农杆菌(北京华越洋生物公司,huayueyang1728s)中,得到重组农杆菌c58/pgwb651-35s::sesmt1a或pgwb651-35s::sesmt1b。
将内质网marker(rfp-hdel,参考文献“hus,yeh,cuiy,jiangl.(2020)atsec62iscriticalforplantdevelopmentandisinvolvediner-phagyinarabidopsisthaliana.journalofintegrativeplantbiology,62:181-200.”)转入c58农杆菌(北京华越洋生物公司,huayueyang1728s)中,得到对照农杆菌c58/marker(rfp-hdel)。
重组农杆菌和对照农杆菌分别在含有20μmas(acetosyringone,乙酰丁香酮),10mmmes,100μmkan(kanamycin,卡那霉素)和100μmrif(rifampicin,利福平)的lb液体培养基中振荡过夜培养(28℃,180rpm)至od600=2.0。离心收集菌体后,用含有200μmas,10mmmgcl2,10mmmes的溶液重悬至od600=2.0,静置6h。
将重悬的两种菌液按1:1的比例混合。用注射器将菌液注射到本生烟草(记载在如下文献中:xianyangchen,hexigedulengbao,jieguo,weitaojia,fangtai,linglingnie,pingjiang,juanjuanfeng,sulianlv,yinxinli.(2014)na+/h+exchanger1participatesintobaccodiseasedefenceagainstphytophthoraparasiticavar.nicotianaebyaffectingvacuolarphandprimingtheantioxidativesystem.journalofexperimentalbotany,65:6107-6122”.材料在文献中的名称为nicotianabenthamiana)叶片中,烟草正常培养2d后,用激光扫描共聚焦显微镜观察蛋白亚细胞定位。
结果如图2所示,上一行图为sesmt1a的亚细胞定位;下一行图为sesmt1b的亚细胞定位。标尺均为10μm。盐角草sesmt1a和sesmt1b与gfp融合蛋白所发出的绿色荧光均与内质网marker(rfp-hdel)所发出的红色荧光重叠,表明sesmt1主要定位在内质网上。这与植物甾醇在内质网合成后再运送至质膜上的研究报道一致。
实施例2、sesmt1在植物抗逆性中的研究
一、基因过表达载体构建
选用pentrytmdirectionaltopo入门载体,设计引物sesmt1-502-entry-f/r。盐角草sesmt1基因引物:
盐角草sesmt1基因引物:
noti-sesmt1-502-entry-f:
5'-ataagaatgcggccgcccccttcaccatgtcaaaatctggagcatttg-3'
asci-sesmt1-502-entry-r:
5'-aggcgcgcccacccttttaattgctatctgaatcaggctt-3'
分别以sesmt1a-pzt4质粒和sesmt1b-pzt4质粒作为模板,noti-sesmt1-502-entry-f和asci-sesmt1-502-entry-r为引物,使用easypfudnapolymerase(全式金,北京)进行pcr扩增,得到1083bppcr产物。
sesmt1a-pzt4质粒的扩增产物的核苷酸序列为序列7,其中序列7第27-1067位为sesmt1a核苷酸序列,序列7第17-26为上游同源臂,第1068-1074为下游同源臂;
sesmt1b-pzt4质粒的扩增产物的核苷酸序列为序列8,其中序列8第27-1067位为sesmt1b核苷酸序列,序列8第17-26为上游同源臂,第1068-1074为下游同源臂;
将回收的pcr产物分别与入门载体pentrytmdirectionaltopo(invitrogen,k240020)通过t4连接酶(全式金公司,fl101-01)连接,构建pentrytmd-topo-sesmt1载体,进行测序确认。
然后采用gatewaytmlrclonasetmiienzymemix(invitrogen,11791-020)将pentrtm/d-topo-sesmt1中间载体质粒与pgwb502ω目的载体(addgene,74844)通过同源重组技术相连接,构建了pgwb502ω-35s::sesmt1融合表达载体。
对该融合表达载体进行酶切验证。采用xbaⅰ和smaⅰ酶切,得到2616bp和8793bp产物,采用speⅰ和hindⅲ酶切得到1090bp和10319bp产物,采用speⅰ和xbaⅰ酶切得到434bp和10975bp产物。3对酶切结果均验证了基因片段正确插入载体。
pgwb502ω-35s::sesmt1a融合载体为将序列7第17-1074位所示的dna片段替换pgwb502ω载体的attr1和attr2位点间的片段得到的,由camv35s启动子驱动sesmt1a表达。
pgwb502ω-35s::sesmt1b融合载体为将序列8第17-1074位所示的dna片段替换pgwb502ω载体的attr1和attr2位点间的片段得到的,由camv35s启动子驱动sesmt1b表达。
pgwb502ω-35s::atsmt1融合载体为将序列9第17-1044位所示的dna片段(含有同源臂的拟南芥atsmt1片段,其中atsmt1的编码核酸为序列9第27-1037位)替换pgwb502ω载体的attr1和attr2位点间的片段得到的,由camv35s启动子驱动atsmt1基因表达。
二、转基因拟南芥的获得及筛选
1、转基因拟南芥的获得
用构建的植物表达载体pgwb502ω-35s::sesmt1a和pgwb502ω-35s::sesmt1b分别转化农杆菌c58菌株,得到重组菌c58/pgwb502ω-35s::sesmt1a和重组菌c58/pgwb502ω-35s::sesmt1b。
将重组菌c58/pgwb502ω-35s::sesmt1a和重组菌c58/pgwb502ω-35s::sesmt1b分别转入野生型拟南芥(columbia-0,wt,该材料记载在文献“sulianlv*,pingjiang*,linglingnie,xianyangchen,fangtai,duoliyawang,pengxiangfan,juanjuanfeng,hexigedulengbao,jinhuiwang,yinxinli.(2015)h+-pyrophosphatasefromsalicorniaeuropaeaconferstolerancetosimultaneouslyoccurringsaltstressandnitrogendeficiencyinarabidopsisandwheat.plantcell&environment,38(11):2433-2449.”)中,获得t0代转sesmt1a拟南芥和t0代转sesmt1b拟南芥。
上述转入方法参照zhang等人(2006)的方法(参考文献“zhangxr,henriquesr,linss,etal.(2006)agrobacterium-mediatedtransformationofarabidopsisthalianausingthefloraldipmethod.natureprotocols,1:641-646.”),利用农杆菌介导的浸花法转化野生型拟南芥。
2、转基因拟南芥纯合系的筛选
将t0代转sesmt1a拟南芥和t0代转sesmt1b拟南芥种子分别经表面灭菌后均匀地撒播于含25mg/l潮霉素(hygromycin,hyg)的1/2ms固体培养基上,首先暗处于4℃下放置2天,然后置于25℃光照下培养7天。挑选阳性苗转入培养土中生长至种子成熟。分单株收取t1代种子。
参照t0代种子,对t1代种子进行表面消毒和播种。种子萌发7d后对后代分离比进行统计,选择分离比为3:1的株系,将阳性苗转移至营养土中生长,直至种子成熟,分单株收取t2代种子。
参照t0代种子,对t2代种子进行表面消毒和播种。种子萌发7d后,选择后代不分离的株系(即为纯合系),对阳性苗进行转移和培养,直至收取t3代种子。
采用同样的方法将pgwb502ω-35s::atsmt1转入野生型拟南芥中,培育得到t3代转atsmt1拟南芥。
3、转基因拟南芥纯合株系sesmt1基因表达量测定
对野生型拟南芥、t3代转sesmt1a拟南芥(sesmt1a-oelines)、t3代转sesmt1b拟南芥(sesmt1b-oelines)和t3代转atsmt1拟南芥(atsmt1-oelines)的种子表面灭菌后,播种于1/2ms培养基上,在4℃下暗培养2天,于25℃光照下培养两周后,各取10株(约100ng),提取总rna经dnase消化,反转录得到的cdna作为模板,利用thunderbird
以拟南芥actin基因作为内参,通过2-δδct的方法对基因表达进行相对定量。
荧光定量rt-pcr中所用引物序列如下:
拟南芥内标actin基因引物:
atactin-qpcr-f:5'-atatgcctatctacagggtt-3'
atactin-qpcr-r:5'-atacaatttcccgttctgctgt-3'
盐角草sesmt1基因特异引物(可用于检测sesmt1a和sesmt1b):
sesmt1-qpcr-f:5'-ctggtgttggcggaaagatcgagaa-3'
sesmt1-qpcr-r:5'-gtgctcatggcgtttgatgctctca-3'
拟南芥atsmt1基因特异引物:
atsmt1-qpcr-f:5'-gagagagtattaagcgacacga-3'
atsmt1-qpcr-r:5'-ggtattcgttattgttgagccc-3'
结果如图3所示,ck,野生型拟南芥;sesmt1a-oelines,t3代转sesmt1a拟南芥;sesmt1b-oelines,t3代转sesmt1b拟南芥;atsmt1a-oelines,t3代转atsmt1拟南芥;*、**和***表示在同一处理条件下,与野生型拟南芥相比,该值分别在p<0.05、p<0.01和p<0.001水平上达到显著差异;可以看出,t3代转sesmt1a拟南芥、t3代转sesmt1b拟南芥和t3代转atsmt1拟南芥株系中smt1的转录本水平均比野生型显著提高。这说明盐角草sesmt1基因和拟南芥中同源基因atsmt1已经分别转入拟南芥,并能稳定表达。
二、转基因拟南芥的抗盐性检测
用70%乙醇和1%次氯酸钠(购自北京化工厂)对野生型拟南芥(wt)、t3代转sesmt1a拟南芥(ss1a-1、ss1a-2)、t3代转sesmt1b拟南芥(ss1b-1、ss1b-2)和t3代转atsmt1拟南芥(as1-1、as1-2)种子进行表面消毒,灭菌水清洗3-5次后播种到1/2ms培养基上。在4℃条件下暗处理2d后,转至人工气候室(温度24-25℃,相对湿度60-70%,光照条件16h光照/8h黑暗)培养。种子萌发5d后,挑取生长一致的小苗分别转到1/2ms培养基(配方参考文献“murashiget,skoogfa.(1962)arevisedmediumforrapidgrowthandbioassayswithtobaccotissueculture.physiologiaplantarum,15:473-497.”;正常条件ck)、含有150mmnacl的1/2ms培养基(将nacl添加到1/2ms培养基中,且使其浓度为150mm;nacl)、含有100μmkno3的1/2ms低氮加盐培养基(将kno3添加到1/2ms培养基中,且使kno3的浓度为100μm;ln)和含有100μmkno3和150mmnacl的1/2ms低氮加盐培养基(将nacl和kno3添加到1/2ms培养基中,且使nacl浓度为150mm,使kno3的浓度为100μm;ln+nacl)上,然后在光照下(光照条件16h光照/8h黑暗)进行竖直盐胁迫培养。盐胁迫培养7天后,测定植株地上部分鲜重、侧根数、主根长和叶绿素含量。
叶绿素含量测定方法:将植株称鲜重后,浸泡于95%的乙醇中,3天后,分别测定浸提液在波长665、649和470nm处的吸光度,并进一步计算叶绿素含量和叶绿素a/叶绿素b比值(参考文献:“lichtenthalerhk.(1987)chlorophyllsandcarotenoids:pigmentsofphotosyntheticbiomembranes.methodinenzymology,148:350-382.”)。
表型结果如图4所示,a.正常生长条件(ck)和100μmkno3(ln)、150mmnacl(nacl)、100μmkno3+150mmnacl(ln+nacl)处理下拟南芥野生型和smt1转基因株系的表型。wt,野生型;as1-1、as1-2,t3代转atsmt1拟南芥株系;ss1a-1、ss1a-2,t3代转sesmt1a拟南芥株系;ss1b-1、ss1b-2,t3代转sesmt1b拟南芥株系;b.地上部分鲜重;c.侧根数;d.根长;e.叶绿素;f.叶绿素a/叶绿素b比值。标尺为1cm。数据为平均值±se(n=18),*和**表示在同一处理条件下,与野生型相比,该值分别在p<0.05和p<0.01水平上达到显著差异。可以看出,在正常生长条件下,各个株系转基因株系侧根多于野生型。在低氮(100μmkno3,ln)处理7天时,拟南芥侧根生长被促进。在盐胁迫(150mmnacl;nacl)处理7天时,拟南芥的生长被抑制。
经生理指标统计表明,低氮处理7天时,t3代转sesmt1a拟南芥和t3代转sesmt1b拟南芥的地上部分鲜重、侧根数和叶绿素a/叶绿素b比值均高于野生型,其中sesmt1b以及t3代转atsmt1拟南芥株系的表型更显著;盐处理7天时,过表达株系地上部分鲜重、侧根数和叶绿素含量均高于野生型;低氮加盐处理7天时,过表达株系地上部分鲜重均显著高于野生型,并且t3代转sesmt1b拟南芥株系的侧根数也显著高于其他株系(图4b-f)。
这些结果说明sesmt1基因的过表达提高了拟南芥苗期的抗盐性和耐低氮的能力,并且该基因的两种序列中sesmt1b在植物耐盐和低氮方面的功能更强大。同时该结果也首次验证了atsmt1过表达能够提高拟南芥在盐胁迫和低氮生境中的耐受能力。
sequencelisting
<110>中国科学院植物研究所
<120>盐角草sesmt1蛋白及其编码基因及应用
<160>9
<170>patentinversion3.5
<210>1
<211>1041
<212>dna
<213>artificialsequence
<400>1
atgtcaaaatctggagcatttgatctagccactggtgttggcggaaagatcgagaaaaat60
gatgtactttctgctgttgacaagtatgagaaataccatggctactatggaggtgaagag120
gaagagagaaaatctaactacactgacatggtgaacaagtattatgatttggttactagt180
ttctatgagtttggttggggagagtccttccatttcgctcacaggtggaatggagaatct240
cttcgtgagagcatcaaacgccatgagcacttccttgctttgcaactaggactaaagcct300
gggaacaaggtattggatgtggggtgtggtattggaggaccactgagggaaatttctaga360
ttcagctcatcatctgtgacagggttgaacaataatgagtatcaaattacaaggggaaag420
gaacttaaccggattgctggggttgacaagacatgtaattttgttaaagctgatttcatg480
aaaatgccatttcctgacaattcatatgatgccatatatgccattgaggcaacatgtcat540
gccccagatgctgttggatgctataaagaaatttaccgagtcttaaagcccggacaacac600
tttgctgcatatgaatggtgcatgacagatgcctttgatgccaaaaaccaagagcaccaa660
aaaatcaaggcagaaattgagcttggtgatgggcttccggatatcagatcaacaaaacag720
tgccttgatgctctgaagaaagcaggttttgagataatttgggagagagatcttgcatct780
gactcaccagttccctggtacttgcctttggatacaaatcatttctctctgagtagcttc840
cgtctaactgctgttggacgttttttcacacgaaacctggtcaaaacattagaatatgtt900
ggacttgctccgtcagggagcctaagagttcagtctttccttgaaaaagctgccgaagga960
ctcgttgacggtggaaggagagagatcttcactcctatgtattttttcctggctcggaag1020
cctgattcagatagcaattaa1041
<210>2
<211>346
<212>prt
<213>artificialsequence
<400>2
metserlysserglyalapheaspleualathrglyvalglyglylys
151015
ileglulysasnaspvalleuseralavalasplystyrglulystyr
202530
hisglytyrtyrglyglyglugluglugluarglysserasntyrthr
354045
aspmetvalasnlystyrtyraspleuvalthrserphetyrgluphe
505560
glytrpglygluserphehisphealahisargtrpasnglygluser
65707580
leuarggluserilelysarghisgluhispheleualaleuglnleu
859095
glyleulysproglyasnlysvalleuaspvalglycysglyilegly
100105110
glyproleuarggluileserargpheserserserservalthrgly
115120125
leuasnasnasnglutyrglnilethrargglylysgluleuasnarg
130135140
ilealaglyvalasplysthrcysasnphevallysalaaspphemet
145150155160
lysmetpropheproaspasnsertyraspalailetyralaileglu
165170175
alathrcyshisalaproaspalavalglycystyrlysgluiletyr
180185190
argvalleulysproglyglnhisphealaalatyrglutrpcysmet
195200205
thraspalapheaspalalysasnglngluhisglnlysilelysala
210215220
gluilegluleuglyaspglyleuproaspileargserthrlysgln
225230235240
cysleuaspalaleulyslysalaglyphegluileiletrpgluarg
245250255
aspleualaseraspserprovalprotrptyrleuproleuaspthr
260265270
asnhispheserleuserserpheargleuthralavalglyargphe
275280285
phethrargasnleuvallysthrleuglutyrvalglyleualapro
290295300
serglyserleuargvalglnserpheleuglulysalaalaglugly
305310315320
leuvalaspglyglyargarggluilephethrpromettyrphephe
325330335
leualaarglysproaspseraspserasn
340345
<210>3
<211>1041
<212>dna
<213>artificialsequence
<400>3
atgtcaaaatctggagcatttgatctagccactggtgttggcggaaagatcgagaaaaat60
gatgtactctctgctgttgacaagtatgagaaataccatggctactatggaggtgaagag120
gaagagagaaaatctaactacactgacatggtgaacaagtattatgatctagttactagt180
ttctatgagtttggttggggagagtccttccatttcgctcacaggtggaatggagaatct240
cttcgtgagagcatcaaacgccatgagcacttccttgctttgcaactaggactaaagcct300
gggaacaaggtattggatgtggggtgtggtattggaggaccactgagggaaatttctaga360
ttcagctcatcatctgtgacagggttgaacaataatgactatcaaattacaaggggaaag420
gaacttaaccggattgctggggttgacaagacatgtaattttgttaaagctgatttcatg480
aaaatgccatttcctgacaattcatatgatgccatatatgccattgaggcaacatgtcat540
gccccagatgctgttggatgctataaagaaatttaccgagtcttaaagcccggacaacac600
tttgctgcatatgaatggtgcatgacagatgcatttgattccaaaaaccaagaccaccaa660
aaaatcaaggcagaaattgagcttggtgatgggcttccggatatcagatcaacaaaacag720
tgccttgatgctctgaagaaagcaggttttgagataatttgggagagagatcttgcatct780
gactcaccagttccctggtacttgcctttggatacaaatcatttctctctgagtagcttc840
cgtctaactgctgttggacgttttttgacacgaaacctggtcaaaacattagaatatgtt900
ggacttgctccgtcagggagcctaagagttcagtctttccttgaaaaagctgccgaagga960
ctcgttgacggtggaaggagagagatcttcactcctatgtattttttcctggctcggaag1020
cctgattcagatagcaattaa1041
<210>4
<211>346
<212>prt
<213>artificialsequence
<400>4
metserlysserglyalapheaspleualathrglyvalglyglylys
151015
ileglulysasnaspvalleuseralavalasplystyrglulystyr
202530
hisglytyrtyrglyglyglugluglugluarglysserasntyrthr
354045
aspmetvalasnlystyrtyraspleuvalthrserphetyrgluphe
505560
glytrpglygluserphehisphealahisargtrpasnglygluser
65707580
leuarggluserilelysarghisgluhispheleualaleuglnleu
859095
glyleulysproglyasnlysvalleuaspvalglycysglyilegly
100105110
glyproleuarggluileserargpheserserserservalthrgly
115120125
leuasnasnasnasptyrglnilethrargglylysgluleuasnarg
130135140
ilealaglyvalasplysthrcysasnphevallysalaaspphemet
145150155160
lysmetpropheproaspasnsertyraspalailetyralaileglu
165170175
alathrcyshisalaproaspalavalglycystyrlysgluiletyr
180185190
argvalleulysproglyglnhisphealaalatyrglutrpcysmet
195200205
thraspalapheaspserlysasnglnasphisglnlysilelysala
210215220
gluilegluleuglyaspglyleuproaspileargserthrlysgln
225230235240
cysleuaspalaleulyslysalaglyphegluileiletrpgluarg
245250255
aspleualaseraspserprovalprotrptyrleuproleuaspthr
260265270
asnhispheserleuserserpheargleuthralavalglyargphe
275280285
leuthrargasnleuvallysthrleuglutyrvalglyleualapro
290295300
serglyserleuargvalglnserpheleuglulysalaalaglugly
305310315320
leuvalaspglyglyargarggluilephethrpromettyrphephe
325330335
leualaarglysproaspseraspserasn
340345
<210>5
<211>1080
<212>dna
<213>artificialsequence
<400>5
ataagaatgcggccgcccccttcaccatgtcaaaatctggagcatttgatctagccactg60
gtgttggcggaaagatcgagaaaaatgatgtactttctgctgttgacaagtatgagaaat120
accatggctactatggaggtgaagaggaagagagaaaatctaactacactgacatggtga180
acaagtattatgatttggttactagtttctatgagtttggttggggagagtccttccatt240
tcgctcacaggtggaatggagaatctcttcgtgagagcatcaaacgccatgagcacttcc300
ttgctttgcaactaggactaaagcctgggaacaaggtattggatgtggggtgtggtattg360
gaggaccactgagggaaatttctagattcagctcatcatctgtgacagggttgaacaata420
atgagtatcaaattacaaggggaaaggaacttaaccggattgctggggttgacaagacat480
gtaattttgttaaagctgatttcatgaaaatgccatttcctgacaattcatatgatgcca540
tatatgccattgaggcaacatgtcatgccccagatgctgttggatgctataaagaaattt600
accgagtcttaaagcccggacaacactttgctgcatatgaatggtgcatgacagatgcct660
ttgatgccaaaaaccaagagcaccaaaaaatcaaggcagaaattgagcttggtgatgggc720
ttccggatatcagatcaacaaaacagtgccttgatgctctgaagaaagcaggttttgaga780
taatttgggagagagatcttgcatctgactcaccagttccctggtacttgcctttggata840
caaatcatttctctctgagtagcttccgtctaactgctgttggacgttttttcacacgaa900
acctggtcaaaacattagaatatgttggacttgctccgtcagggagcctaagagttcagt960
ctttccttgaaaaagctgccgaaggactcgttgacggtggaaggagagagatcttcactc1020
ctatgtattttttcctggctcggaagcctgattcagatagcaataagggtgggcgcgcct1080
<210>6
<211>1080
<212>dna
<213>artificialsequence
<400>6
ataagaatgcggccgcccccttcaccatgtcaaaatctggagcatttgatctagccactg60
gtgttggcggaaagatcgagaaaaatgatgtactctctgctgttgacaagtatgagaaat120
accatggctactatggaggtgaagaggaagagagaaaatctaactacactgacatggtga180
acaagtattatgatctagttactagtttctatgagtttggttggggagagtccttccatt240
tcgctcacaggtggaatggagaatctcttcgtgagagcatcaaacgccatgagcacttcc300
ttgctttgcaactaggactaaagcctgggaacaaggtattggatgtggggtgtggtattg360
gaggaccactgagggaaatttctagattcagctcatcatctgtgacagggttgaacaata420
atgactatcaaattacaaggggaaaggaacttaaccggattgctggggttgacaagacat480
gtaattttgttaaagctgatttcatgaaaatgccatttcctgacaattcatatgatgcca540
tatatgccattgaggcaacatgtcatgccccagatgctgttggatgctataaagaaattt600
accgagtcttaaagcccggacaacactttgctgcatatgaatggtgcatgacagatgcat660
ttgattccaaaaaccaagaccaccaaaaaatcaaggcagaaattgagcttggtgatgggc720
ttccggatatcagatcaacaaaacagtgccttgatgctctgaagaaagcaggttttgaga780
taatttgggagagagatcttgcatctgactcaccagttccctggtacttgcctttggata840
caaatcatttctctctgagtagcttccgtctaactgctgttggacgttttttgacacgaa900
acctggtcaaaacattagaatatgttggacttgctccgtcagggagcctaagagttcagt960
ctttccttgaaaaagctgccgaaggactcgttgacggtggaaggagagagatcttcactc1020
ctatgtattttttcctggctcggaagcctgattcagatagcaataagggtgggcgcgcct1080
<210>7
<211>1083
<212>dna
<213>artificialsequence
<400>7
ataagaatgcggccgcccccttcaccatgtcaaaatctggagcatttgatctagccactg60
gtgttggcggaaagatcgagaaaaatgatgtactttctgctgttgacaagtatgagaaat120
accatggctactatggaggtgaagaggaagagagaaaatctaactacactgacatggtga180
acaagtattatgatttggttactagtttctatgagtttggttggggagagtccttccatt240
tcgctcacaggtggaatggagaatctcttcgtgagagcatcaaacgccatgagcacttcc300
ttgctttgcaactaggactaaagcctgggaacaaggtattggatgtggggtgtggtattg360
gaggaccactgagggaaatttctagattcagctcatcatctgtgacagggttgaacaata420
atgagtatcaaattacaaggggaaaggaacttaaccggattgctggggttgacaagacat480
gtaattttgttaaagctgatttcatgaaaatgccatttcctgacaattcatatgatgcca540
tatatgccattgaggcaacatgtcatgccccagatgctgttggatgctataaagaaattt600
accgagtcttaaagcccggacaacactttgctgcatatgaatggtgcatgacagatgcct660
ttgatgccaaaaaccaagagcaccaaaaaatcaaggcagaaattgagcttggtgatgggc720
ttccggatatcagatcaacaaaacagtgccttgatgctctgaagaaagcaggttttgaga780
taatttgggagagagatcttgcatctgactcaccagttccctggtacttgcctttggata840
caaatcatttctctctgagtagcttccgtctaactgctgttggacgttttttcacacgaa900
acctggtcaaaacattagaatatgttggacttgctccgtcagggagcctaagagttcagt960
ctttccttgaaaaagctgccgaaggactcgttgacggtggaaggagagagatcttcactc1020
ctatgtattttttcctggctcggaagcctgattcagatagcaattaaaagggtgggcgcg1080
cct1083
<210>8
<211>1083
<212>dna
<213>artificialsequence
<400>8
ataagaatgcggccgcccccttcaccatgtcaaaatctggagcatttgatctagccactg60
gtgttggcggaaagatcgagaaaaatgatgtactctctgctgttgacaagtatgagaaat120
accatggctactatggaggtgaagaggaagagagaaaatctaactacactgacatggtga180
acaagtattatgatctagttactagtttctatgagtttggttggggagagtccttccatt240
tcgctcacaggtggaatggagaatctcttcgtgagagcatcaaacgccatgagcacttcc300
ttgctttgcaactaggactaaagcctgggaacaaggtattggatgtggggtgtggtattg360
gaggaccactgagggaaatttctagattcagctcatcatctgtgacagggttgaacaata420
atgactatcaaattacaaggggaaaggaacttaaccggattgctggggttgacaagacat480
gtaattttgttaaagctgatttcatgaaaatgccatttcctgacaattcatatgatgcca540
tatatgccattgaggcaacatgtcatgccccagatgctgttggatgctataaagaaattt600
accgagtcttaaagcccggacaacactttgctgcatatgaatggtgcatgacagatgcat660
ttgattccaaaaaccaagaccaccaaaaaatcaaggcagaaattgagcttggtgatgggc720
ttccggatatcagatcaacaaaacagtgccttgatgctctgaagaaagcaggttttgaga780
taatttgggagagagatcttgcatctgactcaccagttccctggtacttgcctttggata840
caaatcatttctctctgagtagcttccgtctaactgctgttggacgttttttgacacgaa900
acctggtcaaaacattagaatatgttggacttgctccgtcagggagcctaagagttcagt960
ctttccttgaaaaagctgccgaaggactcgttgacggtggaaggagagagatcttcactc1020
ctatgtattttttcctggctcggaagcctgattcagatagcaattaaaagggtgggcgcg1080
cct1083
<210>9
<211>1053
<212>dna
<213>artificialsequence
<400>9
ataagaatgcggccgcccccttcaccatggatctcgcgtcgaatcttggtgggaagatcg60
ataaatccgatgttctcaccgccgtcgaaaagtatgagcaatatcatgtcttccatggag120
gaaacgaggaagagagaaaagccaattatactgacatggttaataagtactatgaccttg180
ctactagcttttatgagtacggatggggagaatccttccattttgcacaaagatggaaag240
gagaatcgcttcgagagagtattaagcgacacgagcactttcttgctcttcagcttggca300
tccaaccaggacaaaaggtactggatgtaggatgtggaattggtggaccgttgagggaaa360
ttgcaagattcagcaattcagttgttaccgggctcaacaataacgaataccagatcacca420
gaggcaaggaactaaaccgacttgcaggtgtcgacaagacatgtaactttgtcaaggctg480
acttcatgaagatgccattcccggaaaacagtttcgatgcagtttatgcaatagaagcaa540
cttgccatgcacctgatgcgtatggatgctacaaagagatctacagagtgctaaagcctg600
ggcaatgttttgctgcctatgagtggtgtatgactgatgcatttgaccctgataacgccg660
aacatcagaaaataaagggagagatagagattggagatggtcttcctgacattaggctga720
ctacaaaatgcctcgaagctctgaagcaggccggttttgaagtgatatgggaaaaggatc780
tggccaaggactcgccggtcccatggtacttacctcttgacaaaaatcatttctcactca840
gtagcttccgccttacagctgttggacgattcataaccaaaaacatggtcaagatccttg900
aatacataagacttgcacctcaaggaagccaaagggtctcaaatttcctggagcaggctg960
cggaaggattagtcgacggtggaaggagagagattttcacgccaatgtatttcttcttgg1020
cccggaagccagagtaaaagggtgggcgcgcct1053
起点商标作为专业知识产权交易平台,可以帮助大家解决很多问题,如果大家想要了解更多知产交易信息请点击 【在线咨询】或添加微信 【19522093243】 与客服一对一沟通,为大家解决相关问题。
与客服一对一沟通,为大家解决相关问题。
此文章来源于网络,如有侵权,请联系删除
 热门咨询
热门咨询



 商标分类
商标分类  商标转让
商标转让 



