一种遗传性原发不孕的致病突变及其检测试剂的制作方法
 2021-02-02 18:02:26|
2021-02-02 18:02:26| 389|
389| 起点商标网
起点商标网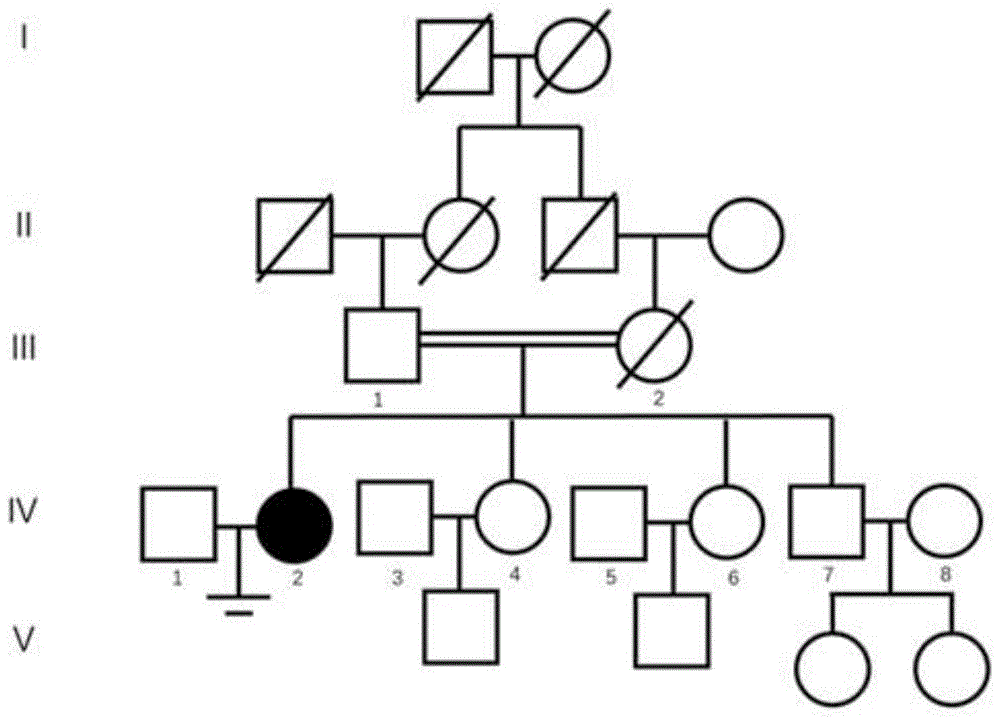
本发明属于生物医药领域,涉及一种遗传性原发不孕的致病突变及其检测试剂。
背景技术:
不孕不育已成为现代社会常见病,who数据表明不孕不育现已成为仅次于肿瘤和心脑血管疾病的第三大疾病。国家计生委发布的数据显示,中国育龄夫妇的不孕不育率近年已攀升至12%-15%,其中女性患者人数已超2500万,因此探究女性不孕症病因已显得尤为迫切。辅助生殖技术是目前广泛采用的不孕症治疗手段,但是临床上仍然有部分不孕症病例既没有找到病因也没有找到有效的医治方法,有相当部分的女性患者不断经历着反复的art周期失败,她们的共同特点为早期胚胎发育障碍。这一临床表型背后的遗传学因素及机制一直未知。因此,发现、鉴定可能的遗传因素对临床诊治尤为重要。
早期胚胎发育障碍以早期胚胎停滞在卵裂期不能发育至囊胚为特征的生殖障碍,其致病机制研究多借助于动物模型,人类的致病基因及遗传模式鲜有报道。研究早期胚胎发育障碍的一个重要目的是对其引起的女性原发性不孕症进行分子诊断。鉴于人类胚胎发育障碍所致不孕相关研究尚有空缺,发现胚胎发育障碍全新的致病基因及位点的研究方法显得尤为重要。高通量二代测序技术能够高效的针对大量基因的遗传信息进行捕获,是寻找复杂遗传性疾病致病基因及其突变最有效的方法,具有传统技术所不可及的优势。
nlrfamilypyrindomaincontaining5基因位于19号染色体长臂19q13.43位置,该基因含有15个外显子,其编码的nlrp5蛋白是一种含有1200个氨基酸的卵母细胞特异性表达蛋白。目前有研究报道nlrp5是重要的母源性蛋白,对于小鼠及人类早期胚胎发育、生命个体起始至关重要,但其致病突变位点仍有待进一步确认。
遗传所致的原发性不孕严重危害了国民的生殖健康。挖掘其新致病突变有利于进一步探索遗传性原发性不孕的分子遗传学病因,是充分利用我国的生殖遗传病资源,造福不孕不育患者的现实需要,是基因组时代最重要的研究方向之一。
本专利旨在探索原发性不孕的遗传学病因,从而帮助了解发病机制、辅助临床诊断、产前诊断和转基因治疗。
技术实现要素:
本发明的目的是针对上述障碍,提供一种遗传性原发不孕的致病突变。
本发明的另一目的是提供该致病突变的应用。
本发明的又一目的是提供该致病突变的检测试剂。
本发明的目的可通过如下技术方案实现:
一种导致遗传性原发不孕的突变的nlrp5基因,所述的突变为纯合的移码突变nlrp5c.2945deltp.leu982fs;野生型nlrp5基因在ensemble数据库中的基因编号为:ensg00000171487,nlrp5基因的突变在物理位置在第11外显子,为56552446的碱基t缺失,后续序列造成移码突变,其他部分与野生型相同。
本发明所述的突变的nlrp5基因编码的突变的nlrp5蛋白,其特征在于野生型nlrp5蛋白在ensemble数据库中的基因转录本编号为enst00000390649.3,突变的nlrp5蛋白在第982号氨基酸移码突变,在翻译2个新的氨基酸后遇到终止密码子,蛋白质终止翻译,蛋白质序列截短,且突变后本来编码富含亮氨酸重复序列lrr的外显子不再编码蛋白,其他部分与野生型相同。
本发明所述的突变的nlrp5基因或本发明所述的突变的nlrp5蛋白作为检测靶点在制备女性原发不孕的辅助诊断试剂或检测设备中的应用。
作为本发明的一种优选,所述的辅助诊断试剂选自引物或引物对、探针、核酸芯片或抗体中的一种或多种。
作为本发明的一种优选,所述的检测设备为含有检测突变的nlrp5基因的基因芯片的检测平台。
检测本发明所述的突变的nlrp5基因或本发明所述的突变的nlrp5蛋白的试剂在制备女性原发不孕的辅助诊断试剂或检测设备中的应用。
作为本发明的一种优选,所述的试剂选自引物或引物对、探针、核酸芯片或抗体中的一种或多种。
一种女性原发性不孕辅助诊断试剂盒,所述的试剂盒包括:检测nlrp5基因物理位置chr1956552446位核苷酸位点的试剂;或检测nlrp5蛋白第982位氨基酸位点的试剂。
作为本发明的一种优选,所述的试剂优选引物或引物对、探针、抗体、或核酸芯片中的一种或多种。
作为本发明的一种优选,所述的试剂为基于深度测序为平台的基因芯片杂交探针;检测chr1956552446位核苷酸位点的杂交探针序列如seqidn0.31所示。
有益效果:
目前已有动物实验表明nlrp5母源蛋白对早期胚胎起着至关重要的作用。因此,针对nlrp5基因与女性原发性不孕及早期胚胎障碍的相关研究显得尤为必要。本专利选择nlrp5的c.2945deltp.leu982fs突变作为原发性不孕的候选位点,是由申请人在多年从事临床医疗、遗传学研究的基础上所筛选出的。
一个基因可以对应多个不同的转录本,且不同转录本所编码的rna及蛋白有所不同。本专利中申请人根据长期从事遗传学研究的经验,筛选出nlrp5基因的最优转录本,并根据不同的转录本设计相应的探针,从而使筛查效益达到最高,最终获得导致早期胚胎发育障碍的致病突变nlrp5c.2945deltp.leu982fs。
原发性不孕的临床表现具有一定的异质性,目前已知一些致病基因如padi6,tle6,但仍存在许多未知的致病基因。本发明提供了重要母源致病基因nlrp5的新致病位点,为该疾病的诊断提供了新的分子生物学基础。
附图说明
图1:家系图
图2:早期胚胎发育障碍患者与正常对照的胚胎照片
图3:nlrp5突变测序图
图4:物种间保守性分析
图5:野生型及携带有突变的nlrp5蛋白三维结构
图6:小鼠胚胎发育图
图7:小鼠胚胎免疫荧光染色
图8:细胞实验免疫共沉淀结果
具体实施方式
实施例1
对一个原发性女性不孕家系的nlrp5基因突变进行检测。
实验方法:
1.该家系临床资源的采集及遗传资源库的建立:
该家系中先证者为患者(ⅳ-2),女性,两年前就诊于南京市妇幼保健院,诊断为原发性不孕症;其妹妹及弟弟生育能力正常;其父母近亲婚配,但母亲已过世。(家系图见图1)。临床资料主要包括个人病史、家族史、生育史等。收集该家系中患者(ⅳ-2)的临床资料及父亲与两位可生育妹妹的血液样本。并用血液基因组dna提取试剂盒(qiagen,hilden,germany)对患者(ⅳ-4)及其父亲(ⅲ-1)、妹妹(ⅳ-4、ⅳ-6)的血液基因组dna进行提取。
2.借助于高通量二代测序挖掘该家系的致病突变:
2.1设计并订制捕获芯片:
2.1.1nlrp5基因及转录本序列信息:
该基因捕获芯片为全外显子组捕获芯片,可对目前所有已知基因进行检测,其中包括全部目前已知的已有研究的早期胚胎阻滞相关的致病基因。我们所参照的nlrp5基因在ensemble数据库中的基因编号为:ensg00000171487(注:该编号来自ensemble数据库,www.ensembl.org,可输入基因编码检索基因详细信息及基因序列)。
2.1.2转录本的选择:
针对不同基因选择特定的转录本,每个基因均含有多个转录本,在选择转录本时,我们的原则是:首先考虑拥有nlrp5编码蛋白的转录本,若一个基因有多个转录本均编码蛋白,则首选含氨基酸数最多的蛋白相对应的转录本,若多个转录本氨基酸含量相同,则进一步选择含碱基数最多的转录本。据以上原则,我们所筛选出的nlrp5基因转录本编号为:enst00000390649.3(注:该编号来自ensemble数据库,www.ensembl.org,可输入转录本编码检索转录本详细信息及转录本序列)。
2.1.2杂交探针的设计:
杂交探针的设计标准为:(1)探针覆盖所有候选基因的目标区域,即外显子区域以及外显子和内含子拼接处(外显子上下游各100个bp);(2)去除重复序列:对于在基因组出现的高度重复序列以及在人类基因组中出现2-5倍的较低频率的重复片段,我们予以去除,避免捕获其他同源基因,增加假阳性,从而降低检测效率。申请人将所有候选基因的目标区域与人类基因组dna序列进行比对,共移除了2.5%的重复序列;(3)在探针设计过程中,我们对临近的外显子进行了特定的整合,其相邻探针整合标准为:当相邻外显子的综合目标区域(即前个外显子的上游100bp起至后一个外显子的下游100bp止)总和小于600bp,即将其整合为一个探针,以求通过一对探针完成多对外显子区域的捕获;(4)当所设计探针序列小于250bp时,在其两端各包含上下游100bp的内含子的基础上,各继续增加相同bp数的内含子,使探针大小达到250bp。根据以上设计原则,我们针对nlrp5基因所设计的探针序列如seqidn0.1—seqidno.31所示。
2.2目标区域捕获及深度测序:
首先将基因组dna片段化,并在dna末端标记“a”,与illuminape接头一寡核苷酸混合物相连接,连接产物经pcr富集,获得dna文库。然后将dna文库与已知致病基因捕获芯片杂交、洗脱、纯化,获得编码序列。最后创建配对末端,在hiseqtm2000(illumina,inc.,sandiego,ca,usa)平台上对目标序列进行测序。
2.3对测序数据进行生物信息学分析,筛选出候选致病基因:
2.3.1采用mosaik软件(http://bioinformatics.bc.edu/marthlab/mosaik)处理工llumina原始测序数据(配对末端数据),产生.bam类型文件。将.bam文件输入gatk,利用gatk检测单核普酸变异体(singlenucleotidevariant)和小的插入或缺失(insertion/deletions),同时进行质量评估,便于下游的生物信息学分析,最后产生.vcf类型文件。
2.3.2将患者的测序结果在包括
dbsnp144(http://hgdownload.cse.ucsc.edu/goldenpath/hgl9/database/snpl44.txt.gz.),hapma计划(ftp://ftp.ncbi.nlm.nih.gov/hapmap),1000genomeproject(ftp://ftp.l000genomes.ebi.ac.uk/voll/ftp),炎黄数据库((http://yh.genomics.org.cnj)以及exomevariantserver(http://evs.gs.washington.edu/evs/)在内的五个单核普酸多态性(snp)数据库中筛查,滤过所有已知的snp位点;
2.3.3将患者的测序结果所对应的基因序列进行比较和分析,优先分析插入/缺失突变、无义突变和错义突变,结果可分为三类,包括已知突变、已知基因的新突变和新基因的突变。
2.4经sanger测序验证,鉴定致病基因:
pcr法分别针对筛选出的突变位点及邻近dna序列在相应家系中进行扩增,所用引物序列采用primer3(http://frodo.wi.mit.edu/)引物设计软件设计。所用pcr的反应体系(20μl体系)为:5*buffer4μl,25mmmgcl22μl,dnalμl,正向引物f1μl,反向引物r1μl,10mmdntp0.4μl,taq酶0.1μl,ddh2o10.5μl。pcr反应程序:98℃5min,35个循环(98℃10s,60℃15s,72℃1min),72℃7min,4℃5min。2%琼脂糖凝胶电泳检测,与紫外线切胶仪下切取pcr产物凝胶并纯化。对所有的pcr产物分别以正、反向引物测序,并对测序结果进行进一步分析,使用ncbi在线对比工具blast(http://blast.ncbi.nlm.nih.gov/),排除假阳性结果,并筛选出在家族中共分离的突变位点。其中检测chr1956552373-56552493位核苷酸位点的引物序列为seqidn0.31。
实验结果:
1.家系临床资料:
生殖医学临床专家对该家系中的先证者(ⅳ-2)进行了详细全面的临床检查后,对该患者做出原发性不孕的临床诊断,并建议进行ivf治疗,具体获卵情况及胚胎培养情况如表1及图2。
表1早期胚胎发育障碍患者(患者ⅳ-2)内分泌检查结果及获卵情况
2.该家系遗传检测结果:
通过对家族成员进行了全外显子组测序及生物信息学分析后,我们发现该患者携带可疑的纯合缺失突变nlrp5c.2945deltp.l982fs。测序结果见图3。患者存在的纯合缺失突变nlrp5c.2945deltp.l982fs位于19号染色体,物理位置为chr19-56552446位缺失1个碱基t;rna水平:nlrp5基因编码rna第2945位碱基缺失;蛋白水平:nlrp5基因编码蛋白第982位氨基酸移码突变并使终止密码子提前,该基因相关的缺失突变从未在原发性不孕患者中发现。
根据我们所设计的筛选流程,借助我们所设计的基因芯片及深度测序技术,我们成功证实所检测到的,nlrp5c.2945deltp.l982fs为该疾病的新致病位点。
实施例2:
针对实施例1中所检测出的致病突变nlrp5c.2945deltp.l982fs进行生物学分析。
实验结果:
1.保守性分析:
采用ncbihomologene数据库(http://www.ncbi.nlm.nih.gov/homologene)对所筛选到的nlrp5p.l982fs突变在人、小鼠、猩猩、羊、狗、马多个物种中进行保守性评估及预测,发现该位点在进化过程中保守,侧面印证了改位点的突变可能会引起较为严重的病理疾病,结果见图3。
2.蛋白晶体结构改变研究:
采用swissmodel(http://swissmodel.expasy.org/)预测软件对nlrp5野生型蛋白及携带有c.2945deltp.leu982fs突变的突变蛋白结构分别进行预测,评估突变所引起的蛋白结构改变。蛋白晶体结构预测结果显示,c.2945deltp.l982fs突变使982所在的lrr单元的疏水区暴露,影响蛋白的折叠或引起聚集,且lrr具有识别ligand功能,leu982提前终止突变可能破坏与配体ligand的识别,结果见图4。
3.小鼠实验结果:
我们从以下几个方面:比较nlrp5wt和nlrp5mumrna注射的组小鼠胚胎的异同:
1)小鼠胚胎发育率:我们对小鼠合子进行nlrp5wt和nlrp5mumrna注射后体外培养比较胚胎发育率,发现nlrp5wt和nlrp5mumrna注射组的小鼠胚胎发育至2细胞阶段并未出现明显差异,但是后续4细胞、桑葚胚及囊胚阶段nlrp5mumrna注射组均明显出现了发育率降低,许多胚胎阻滞在2细胞阶段不在继续发育,这一结果与患者的胚胎发育结果相似见图5。
2)小鼠胚胎中nlrp5蛋白定位的改变:我们比较了nlrp5wt和nlrp5mumrna注射后,小鼠2细胞胚胎中的定位,发现nlrp5mumrna注射后,突变的nlrp5蛋白不再特异定位于皮质下,而是呈现胞质散在分布,这一结果也与结构预测部分内容相吻合,见图6。
4.细胞实验结果:
突变对nlrp5与其他scmc成员结合的影响:
我们分别分别使用myc-nlrp5wt或myc-nlrp5mu与v5-khdc3l、flag-tle6、和ha-ooep高表达质粒转染293t细胞系,通过免疫共沉淀实验发现该突变使nlrp5与scmc组分tle6、ooep、khdc3l相互作用减弱(图8)。该结果提示nlrp5突变可能导致scmc形成障碍。
综上,我们通过遗传学及动物实验学的实验结论从多方面证实了nlrp5c.2945deltp.l982fs突变对于胚胎发育的特异性损坏,阐明了其与原发性不孕的关联。本发明所述的nlrp5突变位点或突变的nlrp5蛋白,可在制备原发性不孕检测试剂或检测设备中应用。
序列表
<110>南京医科大学
<120>一种遗传性原发不孕的致病突变及其检测试剂
<160>39
<170>siposequencelisting1.0
<210>1
<211>120
<212>dna
<213>人工序列(artificialsequence)
<400>1
gttcagttactttccatggcgatgttatcatgaaggttgcaggaggacttgaacttggag60
ctgctgctctgctctcagcatcaccacgtgcgtcgacagcctcttagagttacctatgag120
<210>2
<211>120
<212>dna
<213>人工序列(artificialsequence)
<400>2
tgatgttagttgtatctacttgagaatttgctgcaagatcctcttttaagtcttgtcact60
ctttccacaggtcctacttgctctatattaccaaagaatccacttttcccccaaaacctg120
<210>3
<211>120
<212>dna
<213>人工序列(artificialsequence)
<400>3
agctctcagccttgtatcaagatggaaggagacaaatcgctcaccttttccagctacggg60
ctgcaatggtgtctctatgagctagacaaggaagaatttcagacattcaaggaattacta120
<210>4
<211>120
<212>dna
<213>人工序列(artificialsequence)
<400>4
aagaagaaatcttcagaatcgaccacatgctctattccacagtttgaaatcgagaatgcc60
aacgtggaatgtctggcactcctcttgcatgagtattatggagcatcgctggcctgggct120
<210>5
<211>120
<212>dna
<213>人工序列(artificialsequence)
<400>5
acgtccattagcatctttgaaaacatgaacctgcgaaccctctcggagaaggcacgggat60
gacatgaaaagtaagcgagacttgggacaagtctagggcagggaggggaggtgatttcag120
<210>6
<211>120
<212>dna
<213>人工序列(artificialsequence)
<400>6
cagtctccctttttctttgtcttccaggacattcaccagaagatcctgaagcaacgatga60
ctgaccaaggaccaagcaaggaaaaagtgccaggttagaggggtggagttgggggaaata120
<210>7
<211>120
<212>dna
<213>人工序列(artificialsequence)
<400>7
ttgtgtttattcttctcccttctcttttgcaggaatttcacaagctgtgcaacaagatag60
tgccacagctgcagagacaaaagaacaaggtgaatgaaatagatctattcatttgttgcc120
<210>8
<211>120
<212>dna
<213>人工序列(artificialsequence)
<400>8
caagtatgttggaattcattcttcttttgcagaaatttcacaagctatggaacaagaagg60
tgccacagcagcagagacagaagaacaaggtgaggaaaatagatgtattccttggttgcc120
<210>9
<211>120
<212>dna
<213>人工序列(artificialsequence)
<400>9
acacaagtatgttggaattcattcttttgcagaaatttcacaagctatggaacaagaagg60
tgccacagcagcagagacagaagaacaaggtgaggaaaatagatgtattccttggttgcc120
<210>10
<211>120
<212>dna
<213>人工序列(artificialsequence)
<400>10
tggtctgtttcctgattttcattctaccctctctgactccaggacatggaggtgacacat60
gggactacaagagtcacgtgatgaccaaattcgctgaggaggaggatgtacgtcgtagtt120
<210>11
<211>120
<212>dna
<213>人工序列(artificialsequence)
<400>11
acagccactgtgaggagtactacaccttcttccacctcagtctccaggacttctgtgccg60
ccttgtactacgtgttagagggcctggaaatcgagccagctctctgccctctgtacgttg120
<210>12
<211>120
<212>dna
<213>人工序列(artificialsequence)
<400>12
agaagacaaagaggtccatggagcttaaacaggcaggcttccatatccactcgctttgga60
tgaagcgtttcttgtttggcctcgtgagcgaagacgtaaggaggccactggaggtcctgc120
<210>13
<211>120
<212>dna
<213>人工序列(artificialsequence)
<400>13
tgggctgtcccgttcccctgggggtgaagcagaagcttctgcactgggtctctctgttgg60
gtcagcagcctaatgccaccaccccaggagacaccctggacgccttccactgtcttttcg120
<210>14
<211>120
<212>dna
<213>人工序列(artificialsequence)
<400>14
agactcaagacaaagagtttgttcgcttggcattaaacagcttccaagaagtgtggcttc60
cgattaaccagaacctggacttgatagcatcttccttctgcctccagcactgtccgtatt120
<210>15
<211>120
<212>dna
<213>人工序列(artificialsequence)
<400>15
tgcggaaaattcgggtggatgtcaaagggatcttcccaagagatgagtccgctgaggcat60
gtcctgtggtccctctatggtgagtaccccaggcagttttatcctatgccgtgtgctgag120
<210>16
<211>120
<212>dna
<213>人工序列(artificialsequence)
<400>16
ttgaaaacactgctgctgactggccggaaatgcaaacgttggctggtgcttttgattcag60
accggtggggcttccggcctcgcacggtggttctgcacggaaagtcaggaattgggaaat120
<210>17
<211>120
<212>dna
<213>人工序列(artificialsequence)
<400>17
cggctctagccagaaggatcgtgctgtgctgggcgcaaggtggactctaccagggaatgt60
tctcctacgtcttcttcctccccgttagagagatgcagcggaagaaggagagcagtgtca120
<210>18
<211>120
<212>dna
<213>人工序列(artificialsequence)
<400>18
cagagttcatctccagggagtggccagactcccaggctccggtgacggagatcatgtccc60
gaccagaaaggctgttgttcatcattgacggtttcgatgacctgggctctgtcctcaaca120
<210>19
<211>120
<212>dna
<213>人工序列(artificialsequence)
<400>19
atgacacaaagctctgcaaagactgggctgagaagcagcctccgttcaccctcatacgca60
gtctgctgaggaaggtcctgctccctgagtccttcctgatcgtcaccgtcagagacgtgg120
<210>20
<211>120
<212>dna
<213>人工序列(artificialsequence)
<400>20
gcacagagaagctcaagtcagaggtcgtgtctccccgttacctgttagttagaggaatct60
ccggggaacaaagaatccacttgctccttgagcgcgggattggtgagcatcagaagacac120
<210>21
<211>120
<212>dna
<213>人工序列(artificialsequence)
<400>21
aagggttgcgtgcgatcatgaacaaccgtgagctgctcgaccagtgccaggtgcccgccg60
tgggctctctcatctgcgtggccctgcagctgcaggacgtggtgggggagagcgtcgccc120
<210>22
<211>120
<212>dna
<213>人工序列(artificialsequence)
<400>22
ccttcaaccaaacgctcacaggcctgcacgccgcttttgtgtttcatcagctcacccctc60
gaggcgtggtccggcgctgtctcaatctggaggaaagagttgtcctgaagcgcttctgcc120
<210>23
<211>120
<212>dna
<213>人工序列(artificialsequence)
<400>23
gtatggctgtggagggagtgtggaataggaagtcagtgtttgacggtgacgacctcatgg60
ttcaaggactcggggagtctgagctccgtgctctgtttcacatgaacatccttctcccag120
<210>24
<211>120
<212>dna
<213>人工序列(artificialsequence)
<400>24
ccatgagcccatgtttctatcccccctgacataggatgcgggataagaccctcattgagg60
agcagtgggaagatttctgctccatgcttggcacccacccacacctgcggcagctggacc120
<210>25
<211>120
<212>dna
<213>人工序列(artificialsequence)
<400>25
tgggcagcagcatcctgacagagcgggccatgaagaccctgtgtgccaagctgaggcatc60
ccacctgcaagatacagaccctgatgtaaggctgcccgccccctacgagagaatcccttc120
<210>26
<211>120
<212>dna
<213>人工序列(artificialsequence)
<400>26
accagtgtcgaatgtgtctccccttccccattgcaggtttagaaatgcacagattacccc60
tggtgtgcagcacctctggagaatcgtcatggccaaccgtaacctaagatccctcaactt120
<210>27
<211>120
<212>dna
<213>人工序列(artificialsequence)
<400>27
gggaggcacccacctgaaggaagaggatgtaaggatggcgtgtgaagccttaaaacaccc60
aaaatgtttgttggagtctttgaggtacgtctctggtagagcttttgccttgtttttctt120
<210>28
<211>120
<212>dna
<213>人工序列(artificialsequence)
<400>28
gagctgggattgcttcatgctgcctgtctctgcaggctggattgctgtggattgacccat60
gcctgttacctgaagatctcccaaatccttacgacctcccccagcctgaaatctctgagc120
<210>29
<211>120
<212>dna
<213>人工序列(artificialsequence)
<400>29
ctggcaggaaacaaggtgacagaccagggagtaatgcctctcagtgatgccttgagagtc60
tcccagtgcgccctgcagaagctgatgtgagtgccacttcctttccaccaggattatcgt120
<210>30
<211>120
<212>dna
<213>人工序列(artificialsequence)
<400>30
tcatcatgtcctctctggggctctcttcttgcagactggaggactgtggcatcacagcca60
cgggttgccagagtctggcctcagccctcgtcagcaaccggagcttgacacacctgtgcc120
<210>31
<211>120
<212>dna
<213>人工序列(artificialsequence)
<400>31
tatccaacaacagcctggggaacgaaggtgtaaatctactgtgtcgatccatgaggcttc60
cccactgtagtctgcagaggctgatgtgagtctggcttgctcccctgcaaggacttccta120
<210>32
<211>120
<212>dna
<213>人工序列(artificialsequence)
<400>32
tcacgatcttctttcccatgtgtgtgccccacaggctgaatcagtgccacctggacacgg60
ctggctgtggttttcttgcacttgcgcttatgggtaactcatggctgacgcacctgagcc120
<210>33
<211>120
<212>dna
<213>人工序列(artificialsequence)
<400>33
ttagcatgaaccctgtggaagacaatggcgtgaagcttctgtgcgaggtcatgagagaac60
catcttgtcatctccaggacctggagtgagtttcccatgggcgttgggtcaactctatca120
<210>34
<211>120
<212>dna
<213>人工序列(artificialsequence)
<400>34
gaggcagactctctctattccccgcctcttgcaggttggtaaagtgtcatctcaccgccg60
cgtgctgtgagagtctgtcctgtgtgatctcgaggagcagacacctgaagagcctggatc120
<210>35
<211>120
<212>dna
<213>人工序列(artificialsequence)
<400>35
tcacggacaatgccctgggtgacggtggggttgctgcgctgtgcgagggactgaagcaaa60
agaacagtgttctggcgagactcgggtaacttcctggggcgcctctttgcgggccgggct120
<210>36
<211>120
<212>dna
<213>人工序列(artificialsequence)
<400>36
ttggggttattttctgggtgtcctgaccctgcaggttgaaggcatgtggactgacttctg60
attgctgtgaggcactctccttggccctttcctgcaaccggcatctgaccagtctaaacc120
<210>37
<211>120
<212>dna
<213>人工序列(artificialsequence)
<400>37
tggtgcagaataacttcagtcccaaaggaatgatgaagctgtgttcggcctttgcctgtc60
ccacgtctaacttacagataattgggtaagtcgccagcaattgtcttctgagatacagac120
<210>38
<211>120
<212>dna
<213>人工序列(artificialsequence)
<400>38
ggagaagagtcgaaagcaacatctcagtaacgagtcctctctctgcctccccaggctgtg60
gaaatggcagtaccctgtgcaaataaggaagctgctggaggaagtgcagctactcaagcc120
<210>39
<211>120
<212>dna
<213>人工序列(artificialsequence)
<400>39
ccgagtcgtaattgacggtagttggcattcttttgatgaagatgaccggtactggtggaa60
aaactgaagatacggaaacctgccccactcacacccatctgatggaggaactttaaacgc120
起点商标作为专业知识产权交易平台,可以帮助大家解决很多问题,如果大家想要了解更多知产交易信息请点击 【在线咨询】或添加微信 【19522093243】 与客服一对一沟通,为大家解决相关问题。
与客服一对一沟通,为大家解决相关问题。
此文章来源于网络,如有侵权,请联系删除
 热门咨询
热门咨询



 商标分类
商标分类  商标转让
商标转让 



