核酸检测方法与流程
 2021-02-02 18:02:14|
2021-02-02 18:02:14| 780|
780| 起点商标网
起点商标网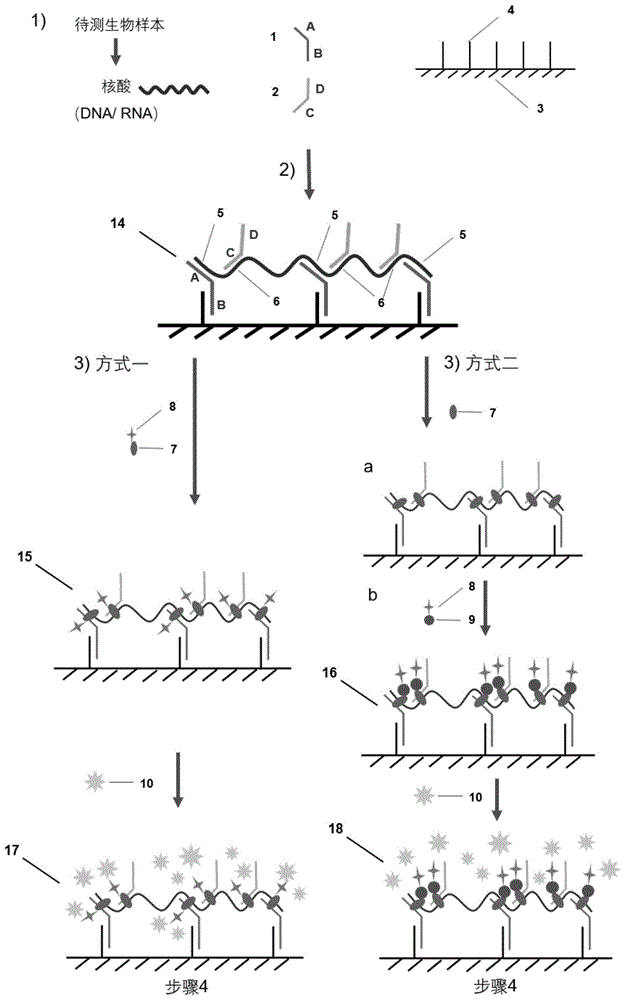
本发明涉及分子生物学领域,具体涉及一种核酸检测方法。
背景技术:
目前检测核酸的主流方法是pcr技术、基因芯片和测序技术。其中,基因芯片和测序技术用于大量基因的检测,对仪器、操作人员以及数据分析的要求比较高,且需要其他的实验来验证,普通实验室无法独立完成。pcr技术适合一种或多种核酸的检测,是实验室常用的技术手段,也被广泛地应用于临床上,对多种疾病、细菌感染和病毒感染进行快速诊断。由于pcr技术需要使用引物对核酸序列的其中一个片段进行扩增,所以要求扩增的片段是完整的。若样本中的核酸序列断裂、降解或发生变异,则无法与引物结合,从而导致假阴性结果。尤其是rna,在一般的环境和运输保存中极易降解,使得pcr容易产生假阴性的结果。另外,很多样本如粪便中含有pcr酶抑制物,会严重影响pcr的扩增效率。因此,pcr技术具有其固有的不确定性。
目前基于杂交捕获进行核酸检测技术有bdna和hc2两种。
bdna(brancheddna,bdna)技术根据固相杂交的原理,采用人工合成的可结合多个酶标记物的分支dna,在目标核酸序列不扩增的情况下,放大检测信号,从而提高灵敏度,克服了pcr技术中的不确定因素。bdna方法已经被美国食品药品监督管理局(fda)批准用于艾滋病毒(hiv)和丙肝病毒(hcv)检测,但是该方法涉及到酶标记探针的合成,价格昂贵,而且耗时长,大约需要16小时。
hc2是美国马里兰州的分子诊断公司digene(后被qiagen公司收购)推出的第二代杂交捕获技术,通过将rna探针与单束dna杂交,随后利用elisa化学发光检测rna和dna形成的杂交物。基于hc2的人乳头瘤病毒(hpv)检测是目前唯一经美国食品药品监督管理局(fda)、欧洲ce和中国食品药品监督管理局(sfda)共同认证的hpv检测技术,准确率高,是公认的hpvdna检测的金标准。但是,该方法需要合成长链rna探针,需要两种抗体,步骤复杂,检测成本昂贵。
技术实现要素:
本发明的目的在于克服现有技术的不足,提供一种高灵敏、高通量、操作简便快捷、低成本的核酸检测方法。
本发明的第二个目的是提供第二种核酸检测方法。
本发明的技术方案概述如下:
一种核酸检测方法,包括如下步骤:
1)合成目标核酸或从待测生物样本中获取含目标核酸的待测物
从genbank检索到目标核酸,从中选出特异性核苷酸序列,并将一部分序列作为捕获功能引物区5,另一部分序列作为标记功能引物区6;
根据所述捕获功能引物区5的序列,设计合成捕获功能区引物1,捕获功能区引物包括a段和b段,所述a段的序列与捕获功能引物区5的序列互补;
根据所述标记功能引物区6的序列,设计合成标记功能区引物2,标记功能区引物包括c段和d段,所述c段的序列与标记功能引物区6的序列互补;
将氨基修饰的通用引物4通过交联吸附法与固相支持物3结合,形成包被有通用引物的固相支持物;所述通用引物的序列与所述捕获功能区引物1的b段的序列互补;
2)将所述待测物或目标核酸与所述捕获功能区引物1和标记功能区引物2一起放入杂交缓冲液中,混合均匀,滴加到所述包被有通用引物的固相支持物上,孵育,弃去液体,用杂交洗液洗涤,弃去液体,获得捕获有目标核酸的固相支持物14;
3)采用下述两种方式之一种进行:
方式一:
将带有酶8标记的特异性抗体7加入到抗体稀释液中,混合均匀,滴加到步骤2)获得的捕获有目标核酸的固相支持物14上,孵育,弃去液体,用抗体洗液洗涤,弃去液体,得到第一种固相支持物15;
将含有酶的底物10的检测液滴加到的第一种固相支持物15上,所述酶的底物与酶进行化学发光反应或进行显色反应,得到第一产物17,用酶标仪检测第一产物信号,根据信号强弱判断待检测核酸的含量。
方式二:
a将特异性抗体7加入到抗体稀释液,混合均匀,滴加到步骤2)获得的捕获有目标核酸的固相支持物14,孵育,弃去液体,用抗体洗液洗涤,弃去液体;
b将带有酶8标记的二抗9加入到抗体稀释液中,混合均匀,滴加到步骤a获得的固相支持物上,孵育,弃去液体,用抗体洗液洗涤,弃去液体,得到第二种固相支持物16;
将含有酶的底物10的检测液滴加到第二种固相支持物16上,所述酶的底物与酶进行化学发光反应或进行显色反应,得到第二产物18,用酶标仪检测第二产物信号,根据信号强弱判断待检测核酸的含量。
第二种核酸检测方法,包括如下步骤:
1)合成目标核酸或从待测生物样本中获取含目标核酸的待测物;
从genbank检索到目标核酸,从中选出特异性核苷酸序列,并将一部分序列作为捕获功能引物区5,另一部分序列作为标记功能引物区6;
根据所述捕获功能引物区5的序列,设计合成捕获功能区引物1,捕获功能区引物包括a段和b段,所述a段的序列与捕获功能引物区5的序列互补;
根据所述标记功能引物区6的序列,设计合成标记功能区引物2,标记功能区引物包括c段和d段,所述c段的序列与标记功能引物区6的序列互补;
将氨基修饰的通用引物4通过交联吸附法与固相支持物3结合,形成包被有通用引物的固相支持物;所述通用引物的序列与所述捕获功能区引物1的b段的序列互补;
2)设计合成核酸支链信号放大系统,所述核酸支链信号放大系统包括初级信号放大链11,次级信号放大链12以及检测引物13;初级信号放大链11序列包括e段和f段;次级信号放大链12序列包括g段和h段;所述e段序列为与所述标记功能区引物2的d段序列互补;所述f段的序列能与2-20条次级信号放大链12的g段序列互补;所述h段序列与2-20条检测引物13序列互补;
3)采用下述两种方式之一种进行:
方式一:
将所述待测物或目标核酸与所述的捕获功能区引物1、标记功能区引物2、初级信号放大链11、次级信号放大链12以及检测引物13一起放入杂交缓冲液,混合均匀,滴加到所述的包被有通用引物的固相支持物上,孵育,弃去液体,用杂交洗液洗涤,弃去液体,获得含核酸支链信号放大系统的捕获有目标核酸的固相支持物19;
方式二:
a将所述待测物或目标核酸与所述捕获功能区引物1和标记功能区引物2一起放入杂交缓冲液中,混合均匀,滴加到所述包被有通用引物的固相支持物上,孵育,弃去液体,用杂交洗液洗涤,弃去液体;
b将所述初级信号放大链11加入杂交缓冲液中,混合均匀,滴加到步骤a获得的固相支持物上,孵育,弃去液体,用杂交洗液洗涤,弃去液体;
c将所述次级信号放大链12加入到杂交缓冲液中,混合均匀,滴加到步骤b获得的固相支持物上,孵育,弃去液体,用杂交洗液洗涤,弃去液体;
d将所述检测引物13加入到杂交缓冲液中,混合均匀,滴加到步骤c获得的固相支持物上,孵育,弃去液体,用杂交洗液洗涤,弃去液体,获得含核酸支链信号放大系统的捕获有目标核酸的固相支持物19;
4)采用下述两种方式之一种进行:
方式一:
将带有酶8标记的特异性抗体7加入到抗体稀释液中,混合均匀,滴加到步骤2)获得的含核酸支链信号放大系统的捕获有目标核酸的固相支持物19上,孵育,弃去液体,用抗体洗液洗涤,弃去液体,获得第三种固相支持物20;
将含有酶的底物10的检测液滴加到第三种固相支持物上,所述酶的底物与酶进行化学发光反应或进行显色反应,得到第三产物22,用酶标仪检测第三产物信号,根据信号强弱判断待检测核酸的含量;
方式二:
a将特异性抗7加入到抗体稀释液,混合均匀,滴加到步骤3)获得的含核酸支链信号放大系统的捕获有目标核酸的固相支持物19上,孵育,弃去液体,用抗体洗液洗涤,弃去液体;
b将带有酶8标记的二抗9加入到抗体稀释液中,混合均匀,滴加到步骤a获得的固相支持物上,孵育,弃去液体,用抗体洗液洗涤,弃去液体,获得第四种固相支持物21;
将含有酶的底物的检测液滴加到第四种固相支持物21上,所述酶的底物与酶进行化学发光反应或进行显色反应,得到第四产物23,用酶标仪检测第四产物信号,根据信号强弱判断待检测核酸的含量。
酶优选:碱性磷酸酶或辣根氧化酶。
待测生物样本选自血液、粘液、尿液、粪便、细胞或组织,也可以选用人工合成。
固相支持物为聚苯乙烯微孔板、尼龙膜、纤维素膜和聚偏二氟乙烯膜中的一种;
氨基修饰的通用引物通过交联吸附法与固相支持物结合的步骤优选为:将氨基修饰的通用引物与化学交联剂一起加入到pbs缓冲液,反应后,加入乙醇得到沉淀,离心后弃去液体,将离心得到的沉淀用pbs溶解,再加入含氨基的蛋白或多肽,混合均匀,滴加到固相支持物上,孵育,弃去液体,用pbs缓冲液洗涤,弃去液体。
本发明的有益效果:
(1)本发明的方法灵敏度高:最低可以检测单个分子的dna和rna或低于1pmmirna,相对以往的杂交捕获核酸检测方法,灵敏度大大提高。
(2)特异性高:本方法对核酸样本的质量要求低,无需扩增目标序列,仅扩增检测信号,因而避开了pcr技术固有的假阴性高的缺点。实验证明本方法可区分出1个碱基突变的mirna,具有超高的特异性。
(3)抗干扰性强:本方法检测过程中不涉及核酸相关的酶反应,对核酸纯净度要求低,克服pcr反应易受样品纯度、片段完整性影响的缺点。
(4)快速:传统的杂交捕获方法一般需要1-2天,而使用本方法检测核酸,可省略核酸提取纯化步骤,整个检测过程小于5小时。
(5)高通量:本方法仅需在最终步骤信号检测时使用酶标仪,之前的所有步骤均可在水浴或恒温箱中进行,可以同时进行多个样本测试。由于可以连续不断的进行检测,具有高通量的特点。
(6)操作简单:本方法不涉及各种复杂的pcr操作,操作人员仅需要掌握简单的移液枪操作,以及简单的酶标仪使用。
(7)成本低:本方法中,除了捕获功能区引物和标记功能区引物需要针对目标核酸序列单独设计外,其他所有材料都是通用,易于获得。经核算,每个样本检测的成本约为荧光定量pcr法的一半。
附图说明
图1是本发明的第一种核酸检测方法流程图;
图2是本发明实施例1中不采取核酸支链信号放大系统的cyp1a1mrna表达的检测结果;
图3是本发明实施例2中不采取核酸支链信号放大系统的mirna21的检测结果;
图4是本发明实施例3中采取核酸支链信号放大系统的cyp1a1mrna表达的检测结果;
图5是本发明实施例4中采取核酸支链信号放大系统的apcdna检测结果;
图6是本发明实施例5中采取核酸支链信号放大系统的covid-19的n片段的检测结果;
图7a是本发明实施例6中采取核酸支链信号放大系统的mirna21的检测结果;
图7b是本发明实施例6中采取核酸支链信号放大系统的mirna21检测方法的特异性验证;
图8是本发明的第二种核酸检测方法流程图。
具体实施方式
下面通过具体实施例对本发明作进一步的说明,以使本领域普通技术人员参照说明书文字能够实施。应当理解,此处所描述的具体实施例仅用于解释本发明,不用于限定本发明。试剂配置:
1)depc处理水(购自索莱宝);
2)核酸裂解液:100mmtris-hcl,200mmnacl,10mmedta,0.5%tritonx-100,1.5mg/ml蛋白酶k;余量为depc处理水;
3)杂交缓冲液:100mmtris-hcl,10mmedta,150mmsodiumcitrate,2%sds,40%formamide,5%bsa,0.05mg/ml鲑鱼精dna;余量是depc处理水;
4)杂交洗液:300mmnacl,30mmsodiumcitrate,0.2%sds,余量是depc处理水;
5)特异性抗体为s9.6(购自sigma),通过hrp偶联试剂盒(购自abcam)获得带有辣根氧化酶标记的特异性抗体。
6)带有辣根氧化酶标记的二抗购自sigma
7)抗体稀释液:含0.05%tween20,5%bsa的1xpbs缓冲液(ph7.4),;
8)抗体洗液:含0.05%tween20的1xpbs缓冲液(ph7.4)
9)检测液:solutiona(0.2%luminol,30mmnaoh,余量是水)和solutionb(3%h2o2,0.03%potassiumferricyanide,余量是水),用时两者按体积比例1:1混合。
实施例1
一种核酸检测方法,包括如下步骤:见图1。
(本实施例以检测小鼠cyp1a1的mrna表达为例)
1)取10mg小鼠新鲜肝脏组织于1.5ml无酶管中,加入100μlrnaisoplus(购自takara)混合,加入4粒磁珠,通过磁力组织破碎仪使得组织破碎裂解,冰上静置5min后,加入20μl三氯甲烷混匀,置于冰上静置5min,12000g离心15min,将上层水相转入新1.5ml无酶管中,加入等体积异丙醇混匀,-20℃静置30min,12000g离心10min,获得rna沉淀,加入75%乙醇上下颠倒润洗沉淀,冰上静置5min后12000g离心5min,弃去液体,打开离心管盖,在室温下静置5分钟,加入30μldepc处理水,溶解所得的rna沉淀,得到含目标核酸cyp1a1mrna的待测物;
从ncbi的genbank中检索获取小鼠cyp1a1的mrna序列(accessionnumber:nm_001136059.2),综合考虑引物长度、tm值、重复序列比率、gc含量、特异性、目标区域覆盖深度等信息,从中选出特异性核苷酸序列,将cyp1a1的mrna的特异性核苷酸序列的一部分作为捕获功能引物区5-1,另一部分序列作为标记功能引物区6-1;
根据所述捕获功能引物区5-1的序列,设计并采用天然核苷酸合成捕获功能区引物1-1-1到1-1-7(共7条),分别用seqidno.1-seqidno.7所示;
捕获功能区引物1-1-1到1-1-7,各自包括a段和b段,其中a段和b段隔有5个碱基为t的天然核苷酸,a段的序列与捕获功能引物区5-1的序列互补,b段是从3’端向5’端数的19个碱基(所有b段的序列都相同);
根据标记功能引物区6-1的序列,设计并采用天然核苷酸合成标记功能区引物2-1-1到2-1-13(共设计13条),分别用seqidno.8-seqidno.20所示;
标记功能区引物2-1-1到2-1-13,各自包括c段和d段,所述c段的序列与标记功能引物区6-1的序列互补,其中c段和d段隔有5个碱基为t的天然核苷酸,从3’端向5’端数的20个碱基为d段,所有的d段的序列都相同;
包被有通用引物的固相支持物的制备:
选择96孔聚苯乙烯微孔板(购自corning)作为固相支持物3;
将1od的氨基修饰的通用引物4(通用引物4用5’-nh2-acaacttctccttaagtag-3’所示,其中的核苷酸序列如seqidno.21所示,由上海生工公司合成)与2mg交联剂disuccinimydyltartrate(购自thermofisher公司)溶于0.5ml1xpbs缓冲液(ph7.4),室温反应30min后,加入1ml乙醇,12000g离心10min,弃去上清,保留沉淀,加入10ml1xpbs缓冲液(ph7.4),溶解沉淀,再加入polylysine(购自sigma公司)至终浓度为1mg/ml,混合均匀,将混合物加入96孔聚苯乙烯微孔板的孔内,每孔100μl,37℃孵育10h后,弃去液体,用1xpbs缓冲液(ph7.4)洗涤三次,甩干,弃去液体,将所得的包被有通用引物的96孔聚苯乙烯微孔板保存于4℃,通用引物的序列与所述捕获功能区引物1-1-1到1-1-7的b段的序列互补;
2)将cyp1a1mrna的7条捕获功能区引物和13条标记功能区引物混合,再用depc处理水稀释,得到每个引物终浓度为10fmole/μl的引物混合物;
在1.5ml无酶管中,加入10μl引物混合物,10μl100pg/μl的待测物(以10μldepc处理水为空白对照)和80μl杂交缓冲液,混合均匀,在85℃加热5min后,迅速放入冰上冷却3min,取出80μl,滴加到包被有通用引物的96孔聚苯乙烯微孔板的孔内,用密封膜将孔覆盖封紧,200g室温离心10s,放入65℃恒温箱内静置孵育,2小时后取出,冷却到室温,弃去液体,用杂交洗液洗三次,每次200μl/孔,甩干,获得捕获有目标核酸的固相支持物14-1;
3)将带有辣根氧化酶8标记的特异性抗体7加入到抗体稀释液中稀释500倍,混合均匀,滴加到步骤2)获得的捕获有目标核酸的固相支持物14-1的孔内,每孔100μl,37℃孵育30min,弃去液体,用抗体洗液洗三次,每次200μl/孔,甩干,得到第一种固相支持物15-1;
将solutiona和solutionb等体积混合配制检测液(其中luminol和h2o2为酶的底物10),滴加到第一种固相支持物15-1上,每孔加入200μl,所述酶的底物与酶进行化学发光反应,得到第一产物17-1,用酶标仪检测化学发光强度,每隔3min读一次,共21min,取最大读数为最终信号值。根据化学发光信号强弱判断目标核酸的含量。也可将检测液更换为tmb单组分显色液,通过显色反应获取核酸信号。
结果如图2所示,本方法可在1ng小鼠肝脏rna中检测到小鼠cyp1a1mrna。
用碱性磷酸酶替代本实施例的辣根氧化酶,其它同本实施例,也可以对小鼠肝脏rna中小鼠cyp1a1mrna进行检测。
将待测样本小鼠肝脏更换为小鼠肝脏细胞,其他同本实施例,也可对小鼠肝脏细胞rna中小鼠肝脏cyp1a1mrna进行检测。
实施例2
一种核酸检测方法,包括如下步骤:
(本实施例以检测人mirna21为例)
1)本实施例中目标核酸mirna21(seqidno.22)由上海生工提供合成;
从ncbi的genbank中获取人mirna21序列(accessionnumber:nr_029493.1),综合考虑引物长度、tm值、重复序列比率、gc含量、特异性、目标区域覆盖深度等信息,将mirna21的特异性核苷酸序列的一部分作为捕获功能引物区5-2,另一部分序列作为标记功能引物区6-2;
根据所述捕获功能引物区5-2的序列,设计并采用非天然核苷酸合成捕获功能区引物1-2,用seqidno.23所示,捕获功能区引物1-2包括a段和b段,所述a段的序列与捕获功能引物区5-2的序列互补,其中a段和b段隔有5个碱基为t的天然核苷酸,从3’端向5’
端数的19个碱基为b段。
根据所述标记功能引物区6-2的序列,设计并采用非天然核苷酸合成标记功能区引物2-2,如seqidno.24所示,标记功能区引物2-2包括c段和d段,所述c段的序列与标记功能引物区6-2的序列互补,其中c段和d段隔有5个碱基为t的天然核苷酸,从3’端向5’
端数的20个碱基为d段;
包被有通用引物的固相支持物的制备参照实施例1的包被有通用引物的固相支持物的制备;
2)将mirna21的1个捕获功能区引物和1个标记功能区引物混合,再用depc处理水稀释,得到每个引物终浓度为10fmole/μl的引物混合物,在1.5ml无酶管中,加入10μl引物混合物,10μl10pmmirna21(以10μldepc处理水为空白对照)和80μl杂交缓冲液,混合均匀,在85℃加热5min后,迅速放入冰上冷却3min,取出80μl,滴加到包被有通用引物的96孔聚苯乙烯微孔板的孔内,用密封膜将孔覆盖封紧,放入40℃恒温箱内静置孵育,2小时后取出,冷却到室温,弃去液体,用杂交洗液洗三次,每次200μl/孔,甩干,获得捕获有目标核酸的固相支持物14-2;
3)a将特异性抗体7加入到抗体稀释液,稀释500倍,混合均匀,滴加到步骤2)获得的捕获有目标核酸的固相支持物14-2上,每孔100μl,37℃孵育30min,弃去液体,用抗体洗液洗涤三次,每次200μl/孔,弃去液体;
b将带有辣根氧化酶8标记的二抗9加入到抗体稀释液中,稀释5000倍,混合均匀,滴加到步骤a获得的固相支持物上,每孔100μl,37℃孵育30min,弃去液体,用抗体洗液三次洗涤,每次200μl/孔,弃去液体,获得第二种固相支持物16-2;
将solutiona和solutionb等体积混合配制检测液(其中luminol和h2o2为酶的底物10),滴加到第二种固相支持物16-2上,每孔加入200μl,所述酶的底物与酶进行化学发光反应,得到第二产物18-2,用酶标仪读取化学发光强度,每隔3min读一次,共21min,取最大读数为最终信号值。也可将检测液更换为tmb单组分显色液,通过显色反应获取核酸信号。
结果如图3所示,本方法在不进行信号放大的情况下,最低可以检测小于1pm的mirna。
除直接合成mirna21的方式外,也可从血液、粘液、尿液、粪便等生物样本中提取mirna,其他同本实施例,对mirna21进行检测。
实施例3
一种核酸检测方法,见图8,包括如下步骤:
(本实施例以检测小鼠cyp1a1的mrna表达为例)
1)同实施例1步骤1);
2)设计并采用天然核苷酸合成小鼠cyp1a1的mrna的核酸支链信号放大系统,所述核酸支链信号放大系统包括初级信号放大链11-3(seqidno.25),次级信号放大链12-3(seqidno.26)以及检测引物13-3(seqidno.27);
初级信号放大链11-3序列包括e段和f段,从3’端向5’端数的20个碱基为e段;次级信号放大链12-3序列包括g段和h段,从3’端向5’端数的20个碱基为g段;所述e段序列为与所述标记功能区引物2-1-1到2-1-13的d段序列互补;所述f段的序列能与4条次级信号放大链12-3的g段序列互补;所述h段序列与4条检测引物(13-3)序列互补;
3)将实施例1所述的cyp1a1mrna的7条捕获功能区引物和13条标记功能区引物混合,再用depc处理水稀释,得到每个引物终浓度为10fmole/μl的引物混合物;
在1.5ml无酶管中,加入10μl引物混合物,10μl10pg/μl待测物(以10μldepc处理水为空白对照)和80μl杂交缓冲液,混合均匀,在85℃加热5min后,迅速放入冰上冷却3min,取出80μl,滴加到包被有通用引物的96孔聚苯乙烯微孔板的孔内,用密封膜将孔覆盖封紧,200g室温离心10s,放入65℃恒温箱内静置孵育,2小时后取出,冷却到室温,弃去液体,用杂交洗液洗三次,每次200μl/孔,甩干,获得获得捕获有目标核酸的固相支持物(cyp1a1mrna的96孔聚苯乙烯微孔板)14-1;
分别将小鼠cyp1a1的mrna的初级信号放大链11-3、次级信号放大链12-3、检测引物13-3用depc处理水配制为100fmole/μl的溶液;
在上述获得的捕获有目标核酸cyp1a1mrna的96孔聚苯乙烯微孔板14-1的每孔内先加入10μl100fmole/μl初级信号放大链和90μl杂交缓冲液,65℃孵育25min,弃去液体,用杂交洗液洗三次,每次200μl/孔,甩干;
再加入10μl100fmole/μl次级信号放大链和90μl杂交缓冲液,65℃孵育25min,弃去液体,用杂交洗液洗三次,每次200μl/孔,甩干;
最后加入10μl100fmole/μl检测引物和90μl杂交缓冲液,65℃孵育25min,弃去液体,用杂交洗液洗三次,每次200μl/孔,甩干,获得含核酸支链信号放大系统的捕获有目标核酸的固相支持物19-3;
4)将带有辣根氧化酶8标记的特异性抗体7加入到抗体稀释液中,稀释500倍,混合均匀,滴加到步骤3)获得的含核酸支链信号放大系统的捕获有目标核酸的固相支持物19-3上,每孔100μl,37℃孵育30min,弃去液体,用抗体洗液洗涤三次,每次200μl/孔,弃去液体,获得第三种固相支持物20-3;
将solutiona和solutionb等体积混合配制检测液(其中luminol和h2o2为酶的底物10),滴加到第三种固相支持物20-3上,每孔加入200μl,所述酶的底物与酶进行化学发光反应,得到第三产物22-3,用酶标仪读取化学发光强度,每隔3min读一次,共21min,取最大读数为最终信号值。也可将检测液更换为tmb单组分显色液,通过显色反应获取核酸信号。
结果如图4所示,本方法采用核酸支链信号放大系统可在0.1ng小鼠肝脏rna中检测到小鼠cyp1a1mrna,且所获取的核酸信号值高于实施例1中未采取核酸支链信号放大系统时获取的核酸信号值。
用碱性磷酸酶替代本实施例的辣根氧化酶,其它同本实施例,也可以对小鼠肝脏rna中小鼠cyp1a1mrna进行检测。
将待测样本小鼠肝脏更换为小鼠肝脏细胞,其他同本实施例,也可对小鼠肝脏细胞rna中小鼠肝脏cyp1a1mrna进行检测。
实施例4
一种核酸检测方法,包括如下步骤:
(本实施例以检测人肠组织apc的dna为例)
1)取20mg人的正常肠组织于预冷好的玻璃匀浆器中,加入200μl核酸裂解液,研磨充分,55℃孵育2hr后,14000rpm,4℃离心10min,将上清转移至1.5ml无酶管中,取40μl于新的1.5ml无酶管中,加入20μl1mnaoh水溶液,63℃加热30min后,加入20μl1mhcl中和至ph=6(也可以是ph=6-9的任意值),得到含目标核酸hapc(人类apc)的待测物;
从ncbigenbank检索到待检测核酸人类apc的基因序列(accessionnumber:nc_000005.10),综合考虑引物长度、tm值、重复序列比率、gc含量、特异性、目标区域覆盖深度等信息,从中选出特异性核苷酸序列,将apc的特异性dna序列一部分序列作为捕获功能引物区5-4,另一部分序列作为标记功能引物区6-4;
根据所述捕获功能引物区5-4的序列,设计并采用天然核苷酸合成捕获功能区引物1-4-1到1-4-6,共6条,分别如seqidno.28-seqidno.33所示;
捕获功能区引物1-4-1到1-4-6各自包括a段和b段,其中a段和b段隔有5个碱基为t的天然核苷酸,从3’端向5’端数的19个碱基为b段,(b段的序列都相同)所述a段的序列与捕获功能引物区5-4的不同序列互补;
根据所述标记功能引物区6-4的序列,设计并采用天然核苷酸合成标记功能区引物2-4-1到2-4-16,共16条,分别用seqidno.34-seqidno.49所示;
标记功能区引物2-4-1到2-4-16各自包括c段和d段,所述c段的序列与标记功能引物区6-4的序列互补,其中c段和d段隔有5个碱基为t的天然核苷酸,从3’端向5’端数的20个碱基为d段;所有的d段的序列都相同;
包被有通用引物的固相支持物的制备同实施例1的包被有通用引物的固相支持物的制备;
2)设计并采用天然核苷酸合成人类apcdna的核酸支链信号放大系统,所述核酸支链信号放大系统包括初级信号放大链11-4(seqidno.50),次级信号放大链12-4(seqidno.51)以及检测引物13-5(seqidno.52);初级信号放大链11-4序列包括e段和f段,从3’端向5’端数的20个碱基为e段;次级信号放大链12-4序列包括g段和h段,从3’端向5’端数的20个碱基为g段;所述e段序列与所述标记功能区引物2-4的d段序列互补;所述f段的序列能与4条次级信号放大链12-4的g段序列互补;所述h段序列与4条检测引物(13-4)序列互补;
3)将apcdna的6个捕获功能区引物和16个标记功能区引物混合,再用depc处理水稀释,得到每个引物的终浓度为10fmole/μl的引物混合物,取40μl含0.1μgdna的步骤1)所得的待测物(以40μl用1mhcl中和至ph=6的核酸裂解液为空白对照),加入10μl引物混合物(10fmole/μl),再加入50μl杂交缓冲液,混匀后离心,取80μl,滴加到包被有通用引物的96孔聚苯乙烯微孔板的孔内,用密封膜将孔覆盖封紧,将96孔聚苯乙烯微孔板放入65℃恒温箱中,孵育2h后取出,冷却到室温,弃去液体,用杂交洗液洗三次,每次200μl/孔,甩干,获得捕获有目标核酸的固相支持物(捕获有目标核酸apcdna的96孔聚苯乙烯微孔板)14-4;
分别将人类apcdna的初级信号放大链11-4、次级信号放大链12-4、检测引物13-4用depc处理水配制为100fmole/μl的溶液,在上述获得的捕获有目标核酸apcdna的96孔聚苯乙烯微孔板14-4的孔内先加入10μl100fmole/μl初级信号放大链和90μl杂交缓冲液,65℃孵育25min,弃去液体,用杂交洗液洗三次,每次200μl/孔,甩干;再加入10μl100fmole/μl次级信号放大链和90μl杂交缓冲液,65℃孵育25min,弃去液体,用杂交洗液洗三次,每次200μl/孔,甩干;最后加入10μl100fmole/μl检测引物和90μl杂交缓冲液,65℃孵育25min,弃去液体,用杂交洗液洗三次,每次200μl/孔,甩干,获得含核酸支链信号放大系统的捕获有目标核酸的固相支持物19-4;
4)a将特异性抗体7加入到抗体稀释液,稀释500倍,混合均匀,滴加到步骤3)获得的含核酸支链信号放大系统的捕获有目标核酸的固相支持物(19-4)上,每孔100μl,37℃孵育30min,弃去液体,用抗体洗液洗涤三次,每次200μl/孔,弃去液体;
b将带有辣根氧化酶8标记的二抗9加入到抗体稀释液中,稀释5000倍,混合均匀,滴加到步骤a获得的固相支持物上,每孔100μl,37℃孵育30min,弃去液体,用抗体洗液洗涤三次,每次200μl/孔,弃去液体,获得第四种固相支持物21-4;
将solutiona和solutionb等体积混合配制检测液(其中luminol和h2o2为酶的底物10),滴加到第四种固相支持物21-4上,每孔加入200μl,所述酶的底物与酶进行化学发光反应,得到第四产物23-4,用酶标仪读取化学发光强度,每隔3min读一次,共21min,取最大读数为最终信号值。也可将检测液更换为tmb单组分显色液,通过显色反应获取核酸信号。
结果如图5所示,本方法采用核酸支链信号放大系统可以从0.1μg的人肠组织dna中检测出apc的dna。
除人肠组织外,也可从血液、粪便等生物样本中提取dna,其他同本实施例,对apc进行检测。
实施例5
一种核酸检测方法,包括如下步骤:
(本实施例以检测新型冠状病毒covid-19的rna为例)
1)从ncbi的genbank中获取covid-19的n段rna序列(accessionnumber:mn908947.3),由苏州金斯瑞公司使用天然核苷酸人工合成,如seqidno.53所示,综合考虑引物长度、tm值、重复序列比率、gc含量、特异性、目标区域覆盖深度等信息,将covid-19的n段特异性rna序列的一部分作为捕获功能引物区5-5,另一部分序列作为和标记功能引物区6-5;
根据所述捕获功能引物区的序列,设计并采用天然核苷酸合成捕获功能区引物1-5-1到1-5-10,共10条,序列分别如seqidno.54-63所示;捕获功能区引物1-5包括a段和b段,其中a段和b段隔有5个碱基为t的天然核苷酸,从3’端向5’端数的19个碱基为b段,所述a段的序列与捕获功能引物区5-5的序列互补;
根据所述标记功能引物区的序列,设计并采用天然核苷酸合成标记功能区引物2-5-1到2-5-20,共20条,序列分别如seqidno.64-seqidno.83所示;标记功能区引物2-5包括c段和d段,所述c段的序列与标记功能引物区6-5的序列互补,其中c段和d段隔有5个碱基为t的天然核苷酸,从3’端向5’端数的20个碱基为d段;
包被有通用引物的固相支持物的制备同实施例1的包被有通用引物的固相支持物的制备;
干扰物:人源rna由人肝癌细胞hepg2经实施例1步骤1)中rnaiso提取法而得;
2)设计并采用天然核苷酸合成covid-19的n段rna的核酸支链信号放大系统,
其中初级信号放大链11-5如seqidno.84所示,
次级信号放大链12-5如seqidno.85所示,
以及检测引物13-5如seqidno.86所示;
初级信号放大链11-5序列包括e段和f段,从3’端向5’端数的20个碱基为e段;次级信号放大链12-5序列包括g段和h段,从3’端向5’端数的20个碱基为g段;所述e段序列为与所述标记功能区引物2-5的d段序列互补;所述f段的序列能与4条次级信号放大链12-5的g段序列互补;所述h段序列与4条检测引物(13-5)序列互补;
3)将covid-19的n段rna的10个捕获功能区引物和20个标记功能区引物等量混合,再用depc处理水稀释,得到各引物的终浓度为10fmole/μl的引物混合物,再将covid-19的n段rna的初级信号放大链11-5、次级信号放大链12-5、检测引物13-5混合,用depc处理水配制各引物终浓度为100fmole/μl的放大链混合物,将1ag的covid-19的n段rna和100ng的人源rna混合至总体积为10μl(以含100ng的人源rna的10μldepc处理水为空白对照),加入至1.5ml无酶管中,再加入5μl引物混合物,5μl放大链混合物,80μl杂交缓冲液,混合均匀,取出80μl,滴加到包被有通用引物的96孔聚苯乙烯微孔板的孔内,用密封膜将孔覆盖封紧,将96孔聚苯乙烯微孔板放入65℃恒温箱中,孵育2h后取出,冷却到室温,弃去液体,用杂交洗液洗三次,每次200μl/孔,甩干,获得含核酸支链信号放大系统的捕获有目标核酸的固相支持物19-5;
4)将带有辣根氧化酶8标记的特异性抗体7加入到抗体稀释液中,稀释500倍,混合均匀,滴加到步骤3)获得的含核酸支链信号放大系统的捕获有目标核酸的固相支持物19-5上,每孔100μl,37℃孵育30min,弃去液体,用抗体洗液洗涤三次,每次200μl/孔,弃去液体,获得第三种固相支持物20-5;
将solutiona和solutionb等体积混合配制检测液(其中luminol和h2o2为酶的底物10),滴加到第三种固相支持物20-5上,每孔加入200μl,所述酶的底物与酶进行化学发光反应,得到第三产物22-5,用酶标仪读取化学发光强度,每隔3min读一次,共21min,取最大读数为最终信号值。也可将检测液更换为tmb单组分显色液,通过显色反应获取核酸信号。
结果如图6所示,本方法在有干扰rna,即人源rna存在的情况下,可以检测到1ag的covid-19的n段rna,相当于1个分子,实现该病毒单分子检测。
除直接合成病毒rna的方式外,也可从血液、粘液、尿液、粪便等生物样本中提取病毒rna,其他同本实施例,对covid-19进行检测。
实施例6
一种核酸检测方法,包括如下步骤:
(本实施例以采用核酸支链信号放大系统检测人mirna21为例)
1)同实施例2的步骤1)
在实施例2的步骤1)的基础上,再增加下述步骤:
待检测核酸mirna21及其对照ncmirna(mirna21a,1个碱基突变,seqidno.87;mirna21b,2个碱基突变,seqidno.88;mirna21c,3个碱基突变,seqidno.89;mirna21d,22个碱基突变,seqidno.90)由上海生工合成;
2)设计合成mirna21的核酸支链信号放大系统,其中初级信号放大链11-6如seqidno.91所示,次级信号放大链12-6如seqidno.92所示,以及检测引物13-6如seqidno.93所示;初级信号放大链11-6序列包括e段和f段,从3’端向5’端数的20个碱基为e段;次级信号放大链12-6序列包括g段和h段,从3’端向5’端数的20个碱基为g段;所述e段序列为与所述标记功能区引物2-2的d段序列互补;所述f段的序列能与4条次级信号放大链12-6的g段序列互补;所述h段序列与4条检测引物(13-6)序列互补;
3)mirna21的1个捕获功能区引物1-2和1个标记功能区引物2-2混合,再用depc处理水稀释,得到终浓度为10fmole/μl的引物混合物,再将其初级信号放大链11-6、次级信号放大链12-6、检测引物13-6等量混合,用depc处理水配制各引物终浓度为100fmole/μl的放大链混合物,取10μl1pm的mirna21(不同突变碱基数的mirna21(以10μldepc处理水为空白对照),加入5μl引物混合物,5μl放大链混合物,80μl杂交缓冲液,混合均匀,取出80μl,滴加到包被有通用引物的96孔聚苯乙烯微孔板的孔内,用密封膜将孔覆盖封紧,将96孔聚苯乙烯微孔板放入40℃恒温箱中,孵育2h后取出,冷却到室温,弃去液体,用杂交洗液洗三次,每次200μl/孔,甩干,获得含核酸支链信号放大系统的捕获有目标核酸的固相支持物(19-6);
4)a将特异性抗体7加入到抗体稀释液,稀释500倍,混合均匀,滴加到步骤3)获得的含核酸支链信号放大系统的捕获有目标核酸的固相支持物(19-6)上,每孔100μl,37℃孵育30min,弃去液体,用抗体洗液洗涤三次,每次200μl/孔,弃去液体;
b将带有辣根氧化酶8标记的二抗9加入到抗体稀释液中,稀释5000倍,混合均匀,滴加到步骤a获得的固相支持物上,每孔100μl,37℃孵育30min,弃去液体,用抗体洗液洗涤三次,每次200μl/孔,弃去液体,获得第四种固相支持物21-6;
将solutiona和solutionb等体积混合配制检测液(其中luminol和h2o2为酶的底物10),滴加到第四种固相支持物21-6上,每孔加入200μl,所述酶的底物与酶进行化学发光反应,得到第四产物23-6,用酶标仪读取化学发光强度,每隔3min读一次,共21min,取最大读数为最终信号值。也可将检测液更换为tmb单组分显色液,通过显色反应获取核酸信号。结果如图7所示,而在信号放大的情况下,从图7a可以得出本方法可以检测到0.1pm的mirna21;图7b体现出该方法特异性高,可区分出1个碱基突变的mirna21。
除直接合成mirna21的方式外,也可从血液、粘液、尿液、粪便等生物样本中提取mirna,其他同本实施例,对mirna21进行检测。
以上各实施例所用聚苯乙烯微孔板也可以采用尼龙膜、纤维素膜和聚偏二氟乙烯膜中的一种进行替代,也可以进行核酸的检测。
序列表
<110>天津贝猫科技有限公司
<120>一种核酸检测方法
<160>93
<170>siposequencelisting1.0
<210>1
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>1
gctgccgatctctgccaatctttttctacttaaggagaagttgt44
<210>2
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>2
tgtgggggatggtgaaggggtttttctacttaaggagaagttgt44
<210>3
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>3
ggtgagaaacctttcaggcctttttctacttaaggagaagttgt44
<210>4
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>4
ccaatggtctctccgatgcatttttctacttaaggagaagttgt44
<210>5
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>5
agtcagcccataggtaggagtttttctacttaaggagaagttgt44
<210>6
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>6
ctaagcctgaagatgctgagtttttctacttaaggagaagttgt44
<210>7
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>7
agaaacaagtccaaagtaggtttttctacttaaggagaagttgt44
<210>8
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>8
accaggtacatgaggctccatttttataggagttggtggtaaagt45
<210>9
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>9
tcctggatctttctctgtactttttataggagttggtggtaaagt45
<210>10
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>10
agggcagctgaggtctgtcatttttataggagttggtggtaaagt45
<210>11
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>11
ggaaggtctccagaatgaagtttttataggagttggtggtaaagt45
<210>12
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>12
ccactggttcacaaagacactttttataggagttggtggtaaagt45
<210>13
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>13
caccccacagttcccggtcatttttataggagttggtggtaaagt45
<210>14
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>14
ccaaagagagtgaccttctctttttataggagttggtggtaaagt45
<210>15
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>15
tggccaggaagagaaagacctttttataggagttggtggtaaagt45
<210>16
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>16
cctggagacaccttaaattctttttataggagttggtggtaaagt45
<210>17
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>17
atctgcacttggaagtgttctttttataggagttggtggtaaagt45
<210>18
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>18
agccacctggtctggtgagctttttataggagttggtggtaaagt45
<210>19
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>19
aggatctgtgtttctggctatttttataggagttggtggtaaagt45
<210>20
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>20
ctgctgtgcccagtgtgtgttttttataggagttggtggtaaagt45
<210>21
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>21
acaacttctccttaagtag19
<210>22
<211>22
<212>rna
<213>智人(homosapiens)
<400>22
uagcuuaucagacugauguuga22
<210>23
<211>35
<212>dna
<213>人工序列(artificialsequence)
<400>23
ctgataagctatttttctacttaaggagaagttgt35
<210>24
<211>36
<212>dna
<213>人工序列(artificialsequence)
<400>24
tcaacatcagtttttttgtggagggattgaaggata36
<210>25
<211>130
<212>dna
<213>人工序列(artificialsequence)
<400>25
ttctaacacatcctacccactattctaacacatcctacccactattctaacacatcctac60
ccactattctaacacatcctacccactattctaacacatcctacccactaactttaccac120
caactcctat130
<210>26
<211>130
<212>dna
<213>人工序列(artificialsequence)
<400>26
gaggagtaggataatgtggtatgaggagtaggataatgtggtatgaggagtaggataatg60
tggtatgaggagtaggataatgtggtatgaggagtaggataatgtggtatgtgggtagga120
tgtgttagaa130
<210>27
<211>20
<212>rna
<213>人工序列(artificialsequence)
<400>27
accacauuauccuacuccuc20
<210>28
<211>43
<212>dna
<213>人工序列(artificialsequence)
<400>28
ctggacaaagcagtaaaactttttctacttaaggagaagttgt43
<210>29
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>29
tcgttcgattgccagctccgtttttctacttaaggagaagttgt44
<210>30
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>30
gcctccagttcaggaaaatgtttttctacttaaggagaagttgt44
<210>31
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>31
cagaatgaaagatgggcaagtttttctacttaaggagaagttgt44
<210>32
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>32
gaaggagttagaggaggggctttttctacttaaggagaagttgt44
<210>33
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>33
catcattacacgcctattgatttttctacttaaggagaagttgt44
<210>34
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>34
cagagtagaagtggtcagcctttttagggagtgagtagtataggt45
<210>35
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>35
gatactttattacattttgctttttagggagtgagtagtataggt45
<210>36
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>36
ctgccatgccaacaaagtcatttttagggagtgagtagtataggt45
<210>37
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>37
agtgtcagtagtagtgatggtttttagggagtgagtagtataggt45
<210>38
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>38
agccaatggttcagaaacaatttttagggagtgagtagtataggt45
<210>39
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>39
ccaggtccagcaagcatctgtttttagggagtgagtagtataggt45
<210>40
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>40
cagttgaactctggaaggcatttttagggagtgagtagtataggt45
<210>41
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>41
caggaatgtgtttctccatatttttagggagtgagtagtataggt45
<210>42
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>42
agaggcagaatcagctccattttttagggagtgagtagtataggt45
<210>43
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>43
gctgccacttgcaaagtttctttttagggagtgagtagtataggt45
<210>44
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>44
agagtgaaccatgcagtggatttttagggagtgagtagtataggt45
<210>45
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>45
agttcagagggtccaggttctttttagggagtgagtagtataggt45
<210>46
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>46
actccagatggattttcttgtttttagggagtgagtagtataggt45
<210>47
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>47
gaatgaaacagaatcagagctttttagggagtgagtagtataggt45
<210>48
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>48
cgaatcccctccaaatgagttttttagggagtgagtagtataggt45
<210>49
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>49
taatggaccaggtccagcaatttttagggagtgagtagtataggt45
<210>50
<211>130
<212>dna
<213>人工序列(artificialsequence)
<400>50
accattacctccattcctcattaccattacctccattcctcattaccattacctccattc60
ctcattaccattacctccattcctcattaccattacctccattcctcattacctatacta120
ctcactccct130
<210>51
<211>130
<212>dna
<213>人工序列(artificialsequence)
<400>51
agtgtgattgggatgaggtattagtgtgattgggatgaggtattagtgtgattgggatga60
ggtattagtgtgattgggatgaggtattagtgtgattgggatgaggtatttgaggaatgg120
aggtaatggt130
<210>52
<211>20
<212>rna
<213>人工序列(artificialsequence)
<400>52
uaccucaucccaaucacacu20
<210>53
<211>1260
<212>dna
<213>2019新型冠状病毒(sarbecovirus,severeacuterespiratorysyndrome-relatedcoronavirus)
<400>53
atgtctgataatggaccccaaaatcagcgaaatgcaccccgcattacgtttggtggaccc60
tcagattcaactggcagtaaccagaatggagaacgcagtggggcgcgatcaaaacaacgt120
cggccccaaggtttacccaataatactgcgtcttggttcaccgctctcactcaacatggc180
aaggaagaccttaaattccctcgaggacaaggcgttccaattaacaccaatagcagtcca240
gatgaccaaattggctactaccgaagagctaccagacgaattcgtggtggtgacggtaaa300
atgaaagatctcagtccaagatggtatttctactacctaggaactgggccagaagctgga360
cttccctatggtgctaacaaagacggcatcatatgggttgcaactgagggagccttgaat420
acaccaaaagatcacattggcacccgcaatcctgctaacaatgctgcaatcgtgctacaa480
cttcctcaaggaacaacattgccaaaaggcttctacgcagaagggagcagaggcggcagt540
caagcctcttctcgttcctcatcacgtagtcgcaacagttcaagaaattcaactccaggc600
agcagtaggggaacttctcctgctagaatggctggcaatggcggtgatgctgctcttgct660
ttgctgctgcttgacagattgaaccagcttgagagcaaaatgtctggtaaaggccaacaa720
caacaaggccaaactgtcactaagaaatctgctgctgaggcttctaagaagcctcggcaa780
aaacgtactgccactaaagcatacaatgtaacacaagctttcggcagacgtggtccagaa840
caaacccaaggaaattttggggaccaggaactaatcagacaaggaactgattacaaacat900
tggccgcaaattgcacaatttgcccccagcgcttcagcgttcttcggaatgtcgcgcatt960
ggcatggaagtcacaccttcgggaacgtggttgacctacacaggtgccatcaaattggat1020
gacaaagatccaaatttcaaagatcaagtcattttgctgaataagcatattgacgcatac1080
aaaacattcccaccaacagagcctaaaaaggacaaaaagaagaaggctgatgaaactcaa1140
gccttaccgcagagacagaagaaacagcaaactgtgactcttcttcctgctgcagatttg1200
gatgatttctccaaacaattgcaacaatccatgagcagtgctgactcaactcaggcctaa1260
<210>54
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>54
agggtccaccaaacgtaatgtttttctacttaaggagaagttgt44
<210>55
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>55
agggtccaccaaacgtaatgtttttctacttaaggagaagttgt44
<210>56
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>56
agggtccaccaaacgtaatgtttttctacttaaggagaagttgt44
<210>57
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>57
agcattgttagcaggattgctttttctacttaaggagaagttgt44
<210>58
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>58
acgagaagaggcttgactgctttttctacttaaggagaagttgt44
<210>59
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>59
cgccattgccagccattctatttttctacttaaggagaagttgt44
<210>60
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>60
gcttcttagaagcctcagcatttttctacttaaggagaagttgt44
<210>61
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>61
taggtcaaccacgttcccgatttttctacttaaggagaagttgt44
<210>62
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>62
cttgagtttcatcagccttctttttctacttaaggagaagttgt44
<210>63
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>63
tcagcactgctcatggattgtttttctacttaaggagaagttgt44
<210>64
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>64
catttcgctgattttggggttttttggtgtggttaggtgtagaaa45
<210>65
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>65
cgttctccattctggttacttttttggtgtggttaggtgtagaaa45
<210>66
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>66
gacgcagtattattgggtaatttttggtgtggttaggtgtagaaa45
<210>67
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>67
gccatgttgagtgagagcggtttttggtgtggttaggtgtagaaa45
<210>68
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>68
catctggactgctattggtgtttttggtgtggttaggtgtagaaa45
<210>69
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>69
ctggtagctcttcggtagtatttttggtgtggttaggtgtagaaa45
<210>70
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>70
gtgatcttttggtgtattcatttttggtgtggttaggtgtagaaa45
<210>71
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>71
gttgttccttgaggaagttgtttttggtgtggttaggtgtagaaa45
<210>72
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>72
ttctgcgtagaagccttttgtttttggtgtggttaggtgtagaaa45
<210>73
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>73
atttcttgaactgttgcgactttttggtgtggttaggtgtagaaa45
<210>74
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>74
gaagttcccctactgctgcctttttggtgtggttaggtgtagaaa45
<210>75
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>75
caagcagcagcaaagcaagatttttggtgtggttaggtgtagaaa45
<210>76
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>76
acattttgctctcaagctggtttttggtgtggttaggtgtagaaa45
<210>77
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>77
acagtttggccttgttgttgtttttggtgtggttaggtgtagaaa45
<210>78
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>78
ttagtggcagtacgtttttgtttttggtgtggttaggtgtagaaa45
<210>79
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>79
atgccaatgcgcgacattcctttttggtgtggttaggtgtagaaa45
<210>80
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>80
ctttgtcatccaatttgatgtttttggtgtggttaggtgtagaaa45
<210>81
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>81
gttggtgggaatgttttgtatttttggtgtggttaggtgtagaaa45
<210>82
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>82
agtttgctgtttcttctgtctttttggtgtggttaggtgtagaaa45
<210>83
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>83
aatcatccaaatctgcagcatttttggtgtggttaggtgtagaaa45
<210>84
<211>130
<212>dna
<213>人工序列(artificialsequence)
<400>84
actatcaacacctcaatcccatactatcaacacctcaatcccatactatcaacacctcaa60
tcccatactatcaacacctcaatcccatactatcaacacctcaatcccattttctacacc120
taaccacacc130
<210>85
<211>130
<212>dna
<213>人工序列(artificialsequence)
<400>85
agttgaggtgagagtaggttatagttgaggtgagagtaggttatagttgaggtgagagta60
ggttatagttgaggtgagagtaggttatagttgaggtgagagtaggttatgggattgagg120
tgttgatagt130
<210>86
<211>20
<212>rna
<213>人工序列(artificialsequence)
<400>86
aaccuacucucaccucaacu20
<210>87
<211>22
<212>rna
<213>人工序列(artificialsequence)
<400>87
uagcuuaucagacagauguuga22
<210>88
<211>22
<212>rna
<213>人工序列(artificialsequence)
<400>88
uagcuuaucagacagaucuuga22
<210>89
<211>22
<212>rna
<213>人工序列(artificialsequence)
<400>89
uagcuuaucagacagaucuuga22
<210>90
<211>22
<212>rna
<213>人工序列(artificialsequence)
<400>90
guaucgauaucugucuacaacu22
<210>91
<211>130
<212>dna
<213>人工序列(artificialsequence)
<400>91
tcacccacatcatccttcataatcacccacatcatccttcataatcacccacatcatcct60
tcataatcacccacatcatccttcataatcacccacatcatccttcataatatccttcaa120
tccctccaca130
<210>92
<211>130
<212>dna
<213>人工序列(artificialsequence)
<400>92
ggatgtgattggttagggaattggatgtgattggttagggaattggatgtgattggttag60
ggaattggatgtgattggttagggaattggatgtgattggttagggaattatgaaggatg120
atgtgggtga130
<210>93
<211>20
<212>rna
<213>人工序列(artificialsequence)
<400>93
uucccuaaccaaucacaucc20
起点商标作为专业知识产权交易平台,可以帮助大家解决很多问题,如果大家想要了解更多知产交易信息请点击 【在线咨询】或添加微信 【19522093243】 与客服一对一沟通,为大家解决相关问题。
与客服一对一沟通,为大家解决相关问题。
此文章来源于网络,如有侵权,请联系删除
 热门咨询
热门咨询



 商标分类
商标分类  商标转让
商标转让 



