一种用于乳腺癌患者预后预测的模型及建立方法与流程
 2021-01-08 11:01:31|
2021-01-08 11:01:31| 297|
297| 起点商标网
起点商标网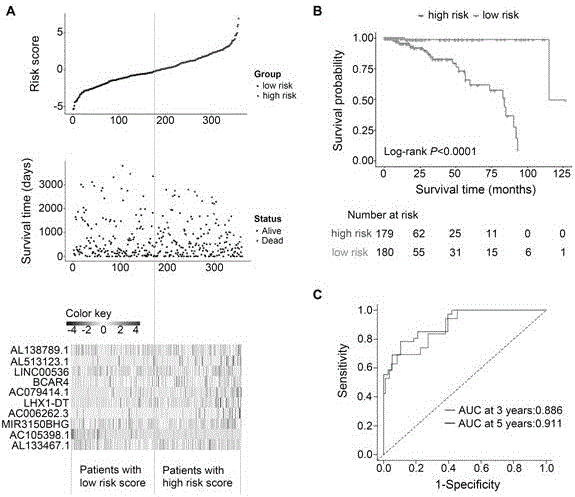
本发明属于生物医学技术领域,涉及一种用于乳腺癌患者预后预测的模型及建立方法。
背景技术:
乳腺癌是女性最常见的恶性肿瘤。自上世纪70年代末,随着生活习惯的改变和人口老龄化的加速,乳腺癌的发病率在世界范围内呈现逐年递增趋势,2018年全球女性乳腺癌新发病例高达2,088,849例,占所有新发肿瘤的11.6%;死亡病例达626,679例,占肿瘤死亡病例总数的6.6%。据我国肿瘤登记中心的统计数据显示,2015年全国约有26.86万新增乳腺癌女性患者,其发病率约37.86/10万,高居女性恶性肿瘤榜首。
目前,临床主要沿用肿瘤形态学特征及其病理学分期来判断乳腺癌患者的预后情况,常见指标包括:肿瘤大小、有无淋巴结转移、组织学分级及临床病理分期等。近年来,国内外众多研究团队相继发现,乳腺癌是一类在分子水平上具有高度异质性的恶性肿瘤,这种分子异质性致使不同患者在临床表现、治疗反应和预后生存等方面表现出显著的差异。越来越多的临床病例显示,同一病理类型或临床分期的乳腺癌患者,即使经历相同的临床治疗,其治疗敏感性及生存时间却大不相同,这为乳腺癌的精准诊疗带来了巨大的困难。
随着二代测序等高通量技术的飞速发展,大量与肿瘤发生发展密切相关的长链非编码rna(lncrnas)相继被发现。近期,大量研究表明lncrnas在乳腺癌组织、血液、尿液等体液中呈现特异的时空表达模式,其中部分分子更被应用于肿瘤早期诊断、病情监测及患者预后评估等方面。但目前多数研究注重于单一分子在乳腺癌恶性转化中的作用机制及其作为肿瘤标志物的临床价值。然而,乳腺癌的演进是由多基因经多种方式调控,单个分子对患者预后的预测能力较为有限,如何发现并整合与乳腺癌预后密切相关的多个lncrnas,对乳腺癌患者预后进行精准判定,以便选择最佳治疗方案,显著提高患者生存率,现已成为临床亟待解决的难题。
技术实现要素:
本发明针对传统单一分子预测能力有限的问题提出一种新型的用于乳腺癌患者预后预测的模型及建立方法。
为了达到上述目的,本发明是采用下述的技术方案实现的:
为了实现上述目的,本发明首先公开了一种用于乳腺癌患者预后预测的模型,其特征在于,包括预后相关lncrnas:al138789.1,al513123.1,linc00536,bcar4,ac079414.1,lhx1-dt,ac006262.3,mir3150bhg,ac105398.1,al133467.1,序列见序列表。
乳腺癌患者的预后风险由十个lncrnas的表达量与其相应回归系数的加权之和构成的风险评分进行评估,计算公式为:风险评分=(0.634×表达量al138789.1)+(0.488×表达量al513123.1)+(0.254×表达量linc00536)+(0.262×表达量bcar4)+(0.825×表达量ac079414.1)+(0.253×表达量lhx1-dt)+(1.115×表达量ac006262.3)+(-0.779×表达量mir3150bhg)+(-1.414×表达量ac105398.1)+(-0.739×表达量al133467.1)。
其次本发明提供一种含十个lncrnas的预测乳腺癌患者预后的模型的建立方法,具体包括以下步骤:
1.数据获取从gdcdataportal(https://portal.gdc.cancer.gov)下载tcga数据库中1109例乳腺癌患者的临床数据及1109例乳腺癌组织和113例正常乳腺组织的rna-seq转录组数据,其中rna-seq转录组数据以ht-seqcount形式显示。
2.差异lncrnas筛选利用r软件“deseq2”包筛选出在乳腺癌组织和正常乳腺组织中差异表达的lncrnas,筛选阈值设置为校正后的p<0.01和|log2(差异倍数)|>2。
3.候选预后相关lncrnas鉴定利用r软件“survival”和“survminer”软件包进行单因素cox回归分析、kaplan-meier生存分析共同鉴定与患者总生存期具有关联性(p<0.05)的差异表达的lncrnas作为候选预后相关lncrnas。
4.预后风险评分模型的构建排除临床信息不完整的病例(剩余718例),随机选取359例患者作为训练集,718例乳腺癌样本全部作为验证集。在训练集中,将上述候选预后相关lncrnas纳入多因素cox逐步回归分析,通过r软件“survival”和“survminer”包构建lncrna风险评分模型,公式如下:quote
5.模型的预后效能评估在训练集和验证集中,依据风险评分公式计算各患者的风险评分,将患者按风险评分的中位值分为高风险组和低风险组,并且通过kaplan-meier生存分析和log-rank检验比较不同风险组患者之间的总生存期是否存在差异,通过r软件“timeroc”包进行时间依赖性roc曲线分析以评估模型的预测能力。此外,依据常见临床病理参数对验证集内患者进行分层,通过kaplan-meier分层生存分析评估该lncrna风险评分模型在乳腺癌各亚组中的预后预测价值。进一步通过时间依赖性roc曲线比较模型与tnm分期的预后价值。
6.统计分析使用r软件(本发明使用3.5.2版本)进行统计分析,使用以下软件包:“deseq2”,“survival”,“survminer”,“timeroc”。
与现有技术相比,本发明的优点和积极效果在于:
1.本发明基于tcga数据库,通过单因素cox回归分析系统筛选出了乳腺癌预后相关lncrnas,进一步通过多因素cox逐步回归分析构建了包含十个lncrnas的预测乳腺癌患者预后的模型。kaplan-meier生存分析、时间依赖性roc曲线均显示模型具有较高的预后预测效能,优于传统的tnm分期,同时此模型能够独立预测乳腺癌不同亚组患者的预后。
2.本发明所建立的模型可以更加精准个体化的预测患者的预后,更好的指导临床决策,为患者治疗方案的选择提供参考,减少不必要的治疗,对于乳腺癌的精准诊疗具有重要的意义。
3.鉴于目前的乳腺癌tnm分期系统及分子分型对患者的预后评估能力有限,本发明的提出对使得该方向的研究取得了有效进展。本发明能对患者的预后情况进行精准评估,具有很强的实用性和指导性。
附图说明
图1为模型预测训练集中患者的预后情况。其中(a)患者的风险评分分布,生存状态和10种lncrnas的表达情况。(b)kaplan-meier生存分析进行高风险组患者和低风险组患者之间生存率的比较。(c)时间依赖性roc曲线分析评估模型的预测效能。
图2为模型预测验证集中患者的预后情况。其中(a)患者的风险评分分布,生存状态和10种lncrnas的表达情况。(b)kaplan-meier生存分析进行高风险组患者和低风险组患者之间生存率的比较。(c)时间依赖性roc曲线分析评估模型的预测效能。
图3为kaplan-meier分层生存分析评估模型的独立预测能力。其中(a)tnmi期亚组。(b)tnmii期亚组。(c)tnmiii期亚组。(d)年龄≥65亚组。(e)年龄<65亚组。(f)er阴性亚组。(g)er阳性亚组。(h)pr阴性亚组。(i)pr阳性亚组。(j)her2阴性亚组。(k)her2阳性亚组。(l)三阴性亚组。(m)非三阴性亚组。
图4为时间依赖性roc曲线比较模型与tnm分期的预后价值。
具体实施方式
为了能够更清楚地理解本发明的上述目的、特征和优点,下面结合具体实施例对本发明做进一步说明。需要说明的是,在不冲突的情况下,本申请的实施例及实施例中的特征可以相互组合。
在下面的描述中阐述了很多具体细节以便于充分理解本发明,但是,本发明还可以采用不同于在此描述的其他方式来实施,因此,本发明并不限于下面公开说明书的具体实施例的限制。
实施例1
一种用于乳腺癌患者预后预测的模型及建立方法。
1.数据获取
从gdcdataportal(https://portal.gdc.cancer.gov)下载tcga数据库中1109例乳腺癌患者的临床数据及1109例乳腺癌组织和113例正常乳腺组织的rna-seq转录组数据,其中rna-seq转录组数据以ht-seqcount形式显示。
2.差异lncrnas筛选
利用r软件“deseq2”包筛选出在乳腺癌组织和正常乳腺组织中差异表达的lncrnas,筛选阈值设置为校正后的p<0.01和|log2(差异倍数)|>2。
3.候选预后相关lncrnas鉴定
利用r软件“survival”和“survminer”软件包进行单因素cox回归分析、kaplan-meier生存分析共同鉴定与患者总生存期具有关联性(p<0.05)的差异表达的lncrnas作为候选预后相关lncrnas。
4.预后风险评分模型的构建
排除临床信息不完整的病例(剩余718例),随机选取359例患者作为训练集,718例乳腺癌样本作为验证集。在训练集中,将上述候选预后相关lncrnas纳入多因素cox逐步回归分析,通过r软件“survival”和“survminer”包构建lncrna风险评分模型,公式如下:quote
5.模型的预后效能评估
在训练集和验证集中,依据风险评分公式计算各患者的风险评分,将患者按风险评分的中位值分为高风险组和低风险组,并且通过kaplan-meier生存分析和log-rank检验比较不同风险组患者之间的总生存期是否存在差异,通过r软件“timeroc”包进行时间依赖性roc曲线分析以评估模型的预测能力。此外,依据常见临床病理参数对验证集内患者进行分层,通过kaplan-meier分层生存分析评估该lncrna风险评分模型在乳腺癌各亚组中的预后预测价值。进一步通过时间依赖性roc曲线比较模型与tnm分期的预后价值。
6.统计分析
使用r软件(3.5.2版本)进行统计分析,使用以下软件包:“deseq2”,“survival”,“survminer”,“timeroc”。
实施例2一种用于乳腺癌患者预后预测的模型的应用。
1.利用单因素cox回归分析、kaplan-meier生存分析共同鉴定乳腺癌预后相关lncrnas
利用r软件“deseq2”包筛选出在乳腺癌组织和正常乳腺组织中差异表达的lncrnas,筛选阈值设置为校正后的p<0.01和|log2(差异倍数)|>2。随后通过r软件“survival”和“survminer”软件包进行单因素cox回归分析、kaplan-meier生存分析,以p<0.05为标准鉴定71个与乳腺癌患者预后有关联性的差异表达的lncrnas。(表1)。
表1.71个与乳腺癌患者预后有关联性的差异表达的lncrnas
2.利用多因素cox逐步回归分析构建乳腺癌预后风险评分模型
在训练集中,将上述71个预后相关lncrnas纳入多因素cox逐步回归分析,最终确定al138789.1、al513123.1、linc00536、bcar4、ac079414.1、lhx1-dt、ac006262.3、mir3150bhg、ac105398.1和al133467.1作为模型的变量,其中al138789.1、al513123.1、linc00536、bcar4、ac079414.1、lhx1-dt和ac006262.3的回归系数均为正值,hr大于1,表明以上7个分子的高表达可能预示患者较差的预后情况;而mir3150bhg、ac105398.1和al133467.1的回归系数为负值,hr均小于1,提示其为乳腺癌患者预后的保护性因素。
多因素cox逐步回归分析模型中的十个lncrnas的详细信息如表2所示:
表2.多因素cox逐步回归分析模型中的十个lncrnas的详细信息
乳腺癌患者的预后风险由十个lncrnas的表达量与其相应回归系数的加权之和构成的风险评分进行评估,计算公式为:
风险评分=(0.634×表达量al138789.1)+(0.488×表达量al513123.1)+(0.254×表达量linc00536)+(0.262×表达量bcar4)+(0.825×表达量ac079414.1)+(0.253×表达量lhx1-dt)+(1.115×表达量ac006262.3)+(-0.779×表达量mir3150bhg)+(-1.414×表达量ac105398.1)+(-0.739×表达量al133467.1)。
3.模型预测训练集中患者的预后情况
依据风险评分公式,计算训练集患者的风险评分,将患者按其风险评分由小到大排列并与其生存状态一一对应,以风险评分的中位值为截断值,将患者分为高风险组和低风险组。风险评分分布,生存状态和10种lncrnas的表达情况(如图1a表示)。kaplan-meier生存分析进行高风险组患者和低风险组患者之间生存率的比较(p<0.0001)(如图1b所示)。时间依赖性roc曲线结果显示模型的3年曲线下面积为0.886,5年曲线下面积为0.911,表明模型对乳腺癌患者预后具有较高的预测价值(如图1c所示)。
4.在验证集中验证模型的预测价值
基于训练集中获得的截断值将验证集患者划分为高风险组和低风险组,每位患者的风险评分分布,生存状态和10种lncrnas的表达情况(如图2a表示)。kaplan-meier生存曲线同样显示高风险组和低风险组患者之间生存率存在显著差异(p<0.0001)(如图2b所示)。进一步时间依赖性roc曲线表明模型3年和5年的曲线下面积分别为0.734和0.781(如图2c所示),证实了该模型具有较高的预测价值,能够可靠预测乳腺癌患者预后风险。
5.模型的kaplan-meier分层生存分析
依据乳腺癌的常见临床预后因素(年龄、tnm分期、er、pr、her2)对乳腺癌患者进行分层,kaplan-meier生存分析表明在多个亚组中高风险组患者与低风险组患者的总生存期存在显著差异(p<0.05)(如图3a-m所示),表明模型在多个亚组中均具有较高的预测能力。
6.时间依赖性roc曲线比较模型与tnm分期的预后价值
进一步评估模型、tnm分期对患者预后的预测能力,时间依赖性roc曲线结果显示模型的预测能力优于tnm分期(auc:0.781vs.0.582,p=0.013),表明模型的预后价值显著优于tnm分期,使用该模型可以辅助临床更准确地预测患者预后。
以上所述,仅是本发明的较佳实施例而已,并非是对本发明作其它形式的限制,任何熟悉本专业的技术人员可能利用上述揭示的技术内容加以变更或改型为等同变化的等效实施例应用于其它领域,但是凡是未脱离本发明技术方案内容,依据本发明的技术实质对以上实施例所作的任何简单修改、等同变化与改型,仍属于本发明技术方案的保护范围。
sequencelisting
<110>山东大学第二医院
<120>一种用于乳腺癌患者预后预测的模型及建立方法
<130>1
<160>10
<170>patentinversion3.5
<210>1
<211>960
<212>dna
<213>人工序列
<400>1
actcacaatacttacatgtcccttttttaagtagtcaagatgtttttccactgcagtctg60
taagtaagagggtacttgaagaatttcctgatgatgatccattaagaaagaaactaatct120
tccagcaagaagctcatcaagatccacttcttcagcacagcataacacacatcgagaaaa180
gcagactgagaagaaggcactcagttgatttgaaggaattcaaattgtttaagtgaagga240
attttgaagactgtggatcatcttgaattttatgtatcccactggatctatctgaaactg300
tgatgtagccacaaacaactaccaggaaatgaaacaaaaattaagatgcaactgtatgac360
agtggacaaaaataaaacaaaaacaatagtaaagttaaaaaataaagcattactatagta420
tatattgttagtatagtatacacagtagttgcttaattcagaagccacttaaataggaca480
catgcaacattcggttacaaacgtgcaagacagatgagtggttttcccatttgtaatata540
actttaaaaaattatttcaacagcctaattaaatggattgagccagaatacatttaaaaa600
atctgttctcagtctgcaagtactagaaacctcataaatataagataattgtggtataat660
aaaatacatatatttgatctttgtccttggtacctggtatggagctcctaaaatccttga720
aatttcctgaatgatagaagtctttagttactcataacaagcctatttcagcgtatctga780
gttcatgctaaggtaactgagggccggccatggtttgaattttcatcaccaactacaacc840
ttgtgggaggagaaaggggctagagattgagttcaattgacaacagtgaccaatgatttg900
gtcagtcatgccttggtaattaaacttcaattaaaactctaaaacatgcaggctgaggga960
<210>2
<211>597
<212>dna
<213>人工序列
<400>2
ggaggggagccaagatggccgaataggaacagctccggtctacagctcccagcgtgagcg60
acccagaagacgggtgatttctgcatttccatctgaggtaccgggttcatctcactaggg120
agtgtcagacagtgggcgcagtttggagttgttgctacctgtgtctacccagccaaggac180
tttatgcctcaggccttcatattcaggacatgtttctcatgaaaattttggaaatgtgaa240
attacagaaaagcccaaaggaaaacaatgaaaatctcttgcaactacagtacttagaaaa300
atgcctattatcattttgttttatatccctgctgtctttatatgtacaacttgcattttg360
caagagtttagaaagttgtaaccttattttttaaatcagtgcataactaatattttttat420
gctattaaaatattattttaagatgtaatatctaatcacatgtctgcatcataatctact480
taatctccccttgttcacaattaagtagtttccaatttttcactatcaaagtaatgctac540
aacaagaaggatatcctggtatataaataattttacacaacacagttatttccttga597
<210>3
<211>2692
<212>dna
<213>人工序列
<400>3
ggctgtggccaagtgattgttgctgaacttacctgtgtcgaaaggtcagccttccagtgc60
taaggctgggcacaggcgctaaggcaaattggattgtgaatggaggccctgaggcatgtt120
ctgcactttgcaactggtctggcaggctccactcctccaccttctaagtctgccacaggc180
atgggaaacacctgggcagcgacaaaggtggttaaggcagcttcgccaagaagaggagcc240
aagatggttaactagacgtagccaggaggaacatctcccaccaagagactgagctgtcag300
gaagactggtacattctgagcagatctttggagggaaggcattgagagtggatggaagga360
tgatgcagacactgggctgaagaggaaggaagctgggaaccctgtacaggactaccgagc420
accaggacttgtttctggccccctgcaactcctgggaaagggagaactcctccagtgctc480
tcaagtgttttacagaccactaattattaaatgtggaagctctggagcagagaacctttc540
aactgaaatgatgacaaacgatgctggctgaggagagtcagacctgaaaagttaggaatc600
cctaaaatgacgtcaacctataattcctattacagagctctgatttctactgatcgtttt660
aggaaccatatctactatgtgagaaaagctgctggtgggcctaatttagatgaaaagcag720
aaaggccttaaaaaagaaatgggaaaggaatatacagaagcaagaagatgattgaaaaaa780
gatggcaattcatgtacttgttgaacaccagttctgagctgcagagcaggaggcacaaga840
cgtaaacaaaaaagtctctgttacggtcctaaagaagccaatcatctagagaaatgtatg900
cacagcaggtccagcagaatgggctttgcgctgatgagggaagcaagaggagagcatggc960
ctgggaaaggagatgatcaggctcacttcccatttctctgtggaagaaccgtggcaaaca1020
cagagtcaactcacaacgttcattggaatttttcccacttgactccttcgcaagttggaa1080
taaggagtttggaaagggaggatttcttcatataaaaatggtctgggtatatccacgcaa1140
aactaaagggagtggaagagaccctaggtctacacatttgatgggatagatccttcgggc1200
tcagcctcacctgcaggtactgaaggaagccttagtctgctatacagagcactctgcacc1260
atggggagaagatcaatatgctaaacttgggtactgggcctaaatcttacgaagtgacct1320
caaacctaggaagggtagtttcatcagaaaagatcctacaaccttctatctgaagagcat1380
gaatagctgctgcttttttggagttggggcctttcattcaccatgaagatactgcattgt1440
ggcacagtccttgaagagacaagtagagacagggtttcaccgtggtgacaggctggtctc1500
gaactcctgacctcaagtgatccgtctgcctcagcatcccaaagtgctgggattacaggc1560
acattgaagaatcagaagatgttttccccctgtgtttttgatggaaggaagaaagcagga1620
tttgccaggatgcattctgcatccaagaacatgaacggaaatgagccacagaaaatctgg1680
agtgatggcagctgttgtgagaggtggcaatgaaatggatttctcagacacagagatgtg1740
gtctcccaccaggtcccatttcagtgtatggtttcttggatttctctgccagcttcaaac1800
agtgcttctgataaagtgatttatgaagtaatgggaaatgtatgttgactcatggagtaa1860
caatgaggaaataggcattggcaccaggactctgttgacatcaggacttcagtgaggggg1920
aaattctgcagagatgtgcataaactcatcgcagctgctctaccatctgacaacctgatg1980
gtcatgggggacctcaatgcacgtgttggtggtagtgccaaaatgtggaagaaaattatt2040
ggaccacagaggattagtctcttcctgcatggtgaacagaccgagcaataacttgtaatc2100
acagacaacagcgtcaatcatataagttaggagaaaaatatcatggatatgccacaaatc2160
aaaaacagtgacatcacacccattctgccattgtttcacagcagaatgccaacaagctga2220
caatctgaatgggtctgagatcgttacccaatatgcttcaaatcaggagaacatacggca2280
acactcattgacttacatattctgggtaagacacagagcaaagaactgcaacagaggtta2340
aagtttcagaaagagaagaagcagccttcactatatcagacagatgaatccagctccaat2400
caataattgagttttggattggaggaaagaatatttggacattaaagaaaataagcagag2460
agtgcctaatctcttcaattctcaagatcatgcttaacatgaccacattaattacagcag2520
agtgagacaggcacagaagaggagggacttggaacctgacctaaatacagaagtgtcaag2580
cctagaaggaagtaaaatttggaagaaagtacatggcatatgaaagaaaaataacaaccc2640
aacaacatctgacattatggaatacttaaataaaaataaaattttagattca2692
<210>4
<211>1273
<212>dna
<213>人工序列
<400>4
ctaagaaggatacagtggctagaagtcgtcctgtcgtcctgcctcacagtaacatcgtta60
ccgaattctcagcaggtgaaccaaatgaaatggtcaactgaaagccaaccagggtaaaac120
agatcttttatttcttgttgttgttattattatttttttttatgtggggacattcaagtg180
aactgctatatgcatttgcccccagtctctccttgtgagaagtgaatttcttcaacgttt240
atagaactgaggtattacattattggatgaattaagaaaacaatctaacctgatgtgtga300
aaatttctgcttgtgagaatccgtgttatttcaattatccaatcaagagcctaattcgta360
taaaagaaacacagcagcttgttgctcatctttttatctaaggacggtttgtcttgacag420
agggtgtctggctgttttggggaaactattcctgaccttattttgactaaaaagttgcct480
gctgtaccagggcctgtgatgtgttgataaaatgccacacaaccatttctcagtgaaaag540
cccaaccgggacttgagttatgttggtggctatggagttctgcaatccacaattgatgtt600
ctctaataaccagtgaccttgagtgaactgtccccaaggcaagagcaaccacagctgcgg660
ggagcgtttggcaatcactatccccacccaagcttctcccttgggtcttgtcctgtcact720
ctggctggaatacaatggcgtaatcatagctcactgcggcctccatctcctgggttcaag780
tgattctcctgcttcagcttcccaagtatctgggactacaggcatcaaaaaatcaccatg840
taccaacctatccaaacttatccatggatgaatctatccagaagacgggagttccgatgc900
ttgtcttgctctgaatgtctgcttgtcacctgcttagggttatcgactgtgattctggga960
ctcattgttgttctacaggacccctctgactctgtggttttctctactggattaacaatg1020
atagccataggtgctttttttgttgtcctcactggagtgacagccctgtgtacggttaca1080
gtcgacgagaacttgcagaaaaccacgaggctaagactaggagtgatacgaaaaagcgga1140
agtctccaaggaactacagagccttccatgactcactcaataatcgctagcacctcgctg1200
tagttgtacattgaaccctggcatcttcgtctttggaactaagtctcctgagcattgttt1260
ttaaatagaaata1273
<210>5
<211>488
<212>dna
<213>人工序列
<400>5
ggccatcagaaggaagcctggacaggtcccttgtttctaagaattcagaggcttgctgac60
tctaactgtgcttgaagcaagatgactgcaggttgcagagtgaaccattgggaaagtttg120
cctttttggggatgaactgattttccaaccctgatgctggcggaaattgagtgatggaac180
taaggtcatctggtgctctccttgagctgcttaataaacagtaaagctattcctttctat240
ttaaatgacataaaaaggaattgatttttagtcacctttccaggaagtgcttcctgaacc300
tgaaaacctgcatggagaaattgaagaaattgttcttaaagggtaaaccaaggaaattgt360
gtccaagatccctgtttctcacaaaaaataagaaaaggaaccaggtaattagatggattg420
ttcgttcaaggcattatgtacacttcttttggttctgaggttaataaagacaaagaagag480
aaaaaggc488
<210>6
<211>1462
<212>dna
<213>人工序列
<400>6
cgcggccccgcgcggcgcggacggcgggcagcaccgtgcctgggccccctcctgctccgg60
ttccagaatgacgctcggcccatggacagagcctgcccagtcactctcggctgctattga120
tcccgaggatgatggccgcccctagccatttccctcctctctcttcggttccagaacagc180
ccaaggagcagaaaagagtgacaaggccgtgaaaggaggcgcctggggcgtagccggctc240
taggaggttaccccatgcggagtacacaccctgctacacccatcccggaggagggaggaa300
atagccagcccggagcacccctggactctgccggtcccagaggccacgcaaactccgccc360
agactcggtgaggccgcggtaatgccacgcagggaactgcgtggataaggtcgctcggat420
ctgagcgccaaggtctcccgcctctccttcccgtctagttctctccggactcaaattgcc480
ttaatctcacccatcactctctcatggaaatgatctactcgtgtgcagtacttcacagct540
tgtatcacggtctcccctgaaccgtatagcaagagggtggtggggggtggaagagaaggg600
tactatctccactttcccggccaggaggcccaagttaggaggcttgtccgagatccctag660
gtcagagcactatttaaggtgagaagttgggtggggagcttggcaccttgctccagtgtg720
cactttgcgcctggccatgttttataagcaaggggggcttgggctggttaagggggagca780
gggaccgcagggccaggaaagggcagcatccggcaggccggggcgtcacggtatgtgcct840
ggcctgggtaaacaactctggcttcccacgagcgctggagggaccctgggcatggggcag900
gggcgtgggggagggggggcaggggcgtggggtggggcagggagcgggtggaacagcgag960
gtcaagcgtggagattccagttgcgagtcagctccgcgccgcggagcaaagacaggacgt1020
ggataatcttgggagggagacgctgagctgaagaacagaaaagagagacagaggcagaag1080
tgaggagagagggacaggaaaagaaaataaaggcagaaaaccagagaaaagatgagacgg1140
agtgcggaaaagcaggcggggataaagaaaggaaagatctgtcggggaaatgcggggaga1200
agtggaggaaaagaaacagaaattagacccagagagagaccctgccacgaagaaggccgc1260
ggggacgggcgcggagccgagggccctgcaggcacggagggggcgtcccgcgggtctggg1320
ggtgtgcggctttgggacaggcgagtgcgtgctgagagaaagaggcagcgtgtttggtgg1380
gagactctgcgcccgtggaaaagcggctctgggcaggaatccaacaaataaataaaacgt1440
ctaagacggcgtgacaacaatg1462
<210>7
<211>258
<212>dna
<213>人工序列
<400>7
ctggccaagggcacgacggcatcatcatagcaacagtgctgcagggggtcgtagaacttg60
tccccacatctcttgtgtggctggcacagcatcaggtccacttgggagcctacatgtccc120
aggcttcgtgtcagacaccagtgatgcagtgctgagtagaaaacgcaaagtcagtgtctt180
cgtgacgttcatgacagtgagaggagagagagagagagagaaggtaggagaataaaactc240
ctgattttggttattgtc258
<210>8
<211>2632
<212>dna
<213>人工序列
<400>8
gcttcccctcactcctcgggccttgctcttcccccaggtgcccactgccctccctgctgt60
gtaccactttcttgctgcctgtgaaggtatttgtcctcactcttgtgctgctccttcaat120
gatagaactttgggactttctcatccagcagcaccagcctgaaaaagcagtggcttgcac180
caccacacacatacctggtctcagatataagctccatgagaacagccaccttgtttacct240
tgttcatggatacagaacagtgcctggtgctcaataaatgaacagtgagtgaataagtga300
gtgttgagtcatctaagtgagcttagatgaactgacaatgagactgtgcacaatgaagag360
aaaggaggactcttgaggaattccaccagcttgaagatggctgaagaaagatgacagaaa420
gcaatcagagggatggagggcaaactaagaaagcagagtcaagagtaggaagattgggaa480
gtggtagacaacattaggaaatactgcagaaaaacctaaaaggatgagaattaggaaaga540
cactattgaatttggtctttggggctgaaaaaaaaggcagctgcagaaaagaaattggag600
tggaaccccatttgcacagaaattatggagtaattaggacaccaggcagaaagtttggca660
atgaatggaaggagagatgacagccttgggcattcgctcgcagaacgcaaggacattcat720
tcttaattcagtagaaaagtgtcgtgttgttactgtctcaagtggaagcgccctctcact780
ccttcattgtcctaagagaacaaaaggggccttgaatttcatgactgccccagtgatgct840
tctgagagcttgagctttggaatcagactgcccacattcaaaccccagttcttcctattg900
ctggctgtgtgatttggggaaaattacttgattccctaagttttctcatctgtgaaatgg960
ggattcactcagttagtgaatattattcagctcctaccatgtgcagacactgttctagtg1020
ggggattcagcataccagcagtaaatggatctctgtacaatgccaaaaacatgaagccaa1080
ctattcatagtatttttaagtgatatcctggtgctgcttccaggaagatggagtagatat1140
acttttccctattgctcccactaagtacaactaaaaccctggacattatgtataaacatg1200
cataagaagatgctgaaaggtggtaagaaagcagactggccagggactttgcgatccaag1260
gaacaacctggtgatgagtttcctgtaccccagacttgggataaaggagctggcaaccca1320
gaaatgccaacgggagcaaataaaaaatccccaacaaaagcctgtttctctagacaaagg1380
actaggacaggggcagcctaacaaaacagaaaactcttagtaacaacttactccagctaa1440
atagcacagaaaaacctatggtccactcccacccacaccacccaagggtgtgggagtcca1500
ggctatcacaaggggcctcgcctccactggtgtggtatcgcaaaagccacagggagctgg1560
ttctttcatccccatctgacaaaaggctcccacccccatggtgtcagcggagaccacact1620
gggagcctgaacttccacccactcacagctgtaacaaggcagtccctccacctccctgct1680
ggggtggtgtcagaggaggccaagtggagagtcaggactttcaccatagcccagtgacaa1740
tgaagccacctccctatggtgtcagtggaggccataaggaacagtaacgaggtctagtgg1800
gaatccagaactgccaccctgcccagcagtaaccaggagcccctcccctccgtgtggaca1860
gaggccaagtgctgaagctggactttgctccccctagccctgctaggtggctattagaac1920
agccagctaaaactggtttcaccaagacagatttaggaagacagtgccccgggcaggaaa1980
atgaaggcacggagaggttaaatataataacttgctagtggtcaatcagtaggcagtaga2040
ggagccagcatataaacccaagctgcccagctgcagaaccacattcttgtcccatatgct2100
atgcctctcaaaaaaggaggccctctccttgggacacaaaggtaataaaaatgcctgccc2160
tgagctgtgtctctgtctaggtacacagttcatgagtgaaggagctcctggagtcatgaa2220
taatggaagtccagattggtcaagtaacttgtcccagccccagcccatgcatctggtatg2280
tggttggacaggtttctaactcattacgtaacatcaagaacattttaaactgctttgaga2340
catttgtgaattaagatctcaataaatagtcctccctggcaccagctccatccacattaa2400
atgagacttgaaaatttctttgaatggatattagtttagaaggagctaagttattagttg2460
gcctcacattcctcagttgggccctgggcccagcatgccacttctcctggttattatggt2520
tcacatctgaggtaacccatcgggaaactggatgggacaacaaactaaaaatgttttagt2580
ttggacctgtccaaaaccaagagaataaaaacaaaatggatagtcaagatga2632
<210>9
<211>108
<212>dna
<213>人工序列
<400>9
ggctgcgtctgaaggtgatgagttatctccattataacagagtcagttacacataaaact60
cgtcctcctctttcctctcttctcactactccacttgactagtctaaa108
<210>10
<211>993
<212>dna
<213>人工序列
<400>10
gctctgctctcccaggcactactgagcgcgctcgcggtcgccaggagcggattgggcccc60
cgccgcgcgcaagccttggtgacaggggatggcgcggagctgcctctccgggaaagttgg120
atctgtgcgcccagacatggaaaagggattaggacaccggcagatcttgccctcacagag180
cggaggtgggggccttggaaggggcacaccgcccgtaggcgcgaaatccaagaagaaaat240
gacactcatcagccagcctcgcgcggggagaaagggcggggtggggggtagtggcaccct300
gcccgtcctctcccacccccacagtccgggcccacagtccaggagtcacaagggatgtga360
atccccaggcataatgtgaacccccaacaaacccaaggcctcagaatgcgcctcacctct420
ccctccccaccagcagaaacatcctcccactctcccaacccctgctccagccacgccaac480
cctttaaccttgtcccagaatgtctaaactgtatctgcctctgtgcctgtgcacatgaag540
ttgccttggcccggcgtttgctccacagccttctgccaaactcctactgcttgatggctg600
gaagccttcgcaccctggcgctctcctcccaggtcagcagtcactggccttcctgtgcat660
ccagcccagctttgaacagtcctgggccccaacccttatctttttccgagtaattacatg720
gttgcatgattggcttcaccgcctctggatcagatgagcgctactaatgaaggatacaag780
ttgtaaaactaggttttatgtgaactccagtcatcacagtttaggtgcggctagcgattt840
tacagatgcggggttagtaactggcccaaatccagggcttccgggatgctgttttccttt900
tttgtttcttagcagggatggcagatgtttgtagaacagatccgtgtaaaagtcagtatc960
ctaagagattaaattgtaaagcaggacaagata993
起点商标作为专业知识产权交易平台,可以帮助大家解决很多问题,如果大家想要了解更多知产交易信息请点击 【在线咨询】或添加微信 【19522093243】 与客服一对一沟通,为大家解决相关问题。
与客服一对一沟通,为大家解决相关问题。
此文章来源于网络,如有侵权,请联系删除
 热门咨询
热门咨询



 商标分类
商标分类  商标转让
商标转让 



