一种合成抗体文库及其构建方法和用途与流程
 2021-02-02 08:02:45|
2021-02-02 08:02:45| 293|
293| 起点商标网
起点商标网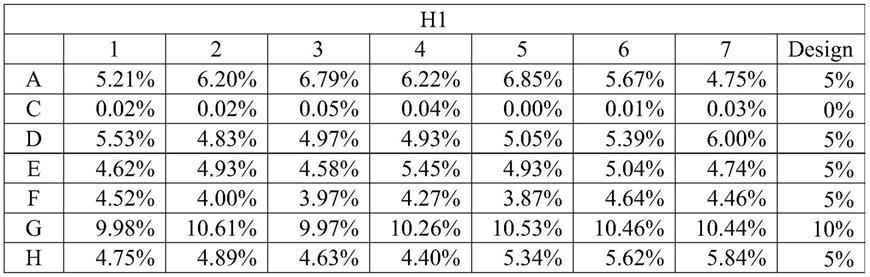
[0001]
本发明涉及抗体文库构建领域,特别是涉及一种合成抗体文库及其构建方法和用途。
背景技术:
[0002]
抗体是可以特异性识别其抗原的一种蛋白质,常规的抗体都有两条多肽链,一条重链(hc)和一条轻链(lc)通过二硫键连接在一起组成一个异源二聚体。重链的fc端又通过额外的二硫键相连成为异源四聚体。抗体是通过轻、重链上6个互补决定区(cdr)识别其抗原的,轻链上有3个互补决定区,分别是cdrl1、cdrl2和cdrl3,与之相对应,重链上的3个互补决定区分别是cdrh1、cdrh2和cdrh3。这6个cdr区是抗体识别和结合其抗原的主要区域。人们在研究中发现,骆驼类的动物中,抗体只有一条重链,被称做单域抗体,单域抗体的n端缺乏常规抗体的ch1区域,只有100个左右氨基酸残基,直径只有十几个纳米,因此也被称为纳米抗体。与常规抗体类似,纳米抗体上也有3个互补决定区:cdrh1、cdrh2和cdrh3。
[0003]
杂交瘤技术的发明使得人类第一次能够制备高质量的单克隆抗体。此技术将免疫b淋巴细胞与骨髓瘤细胞融合成杂交瘤细胞,得到的杂交细胞具有双亲细胞的遗传特性,既能像b淋巴细胞一样分泌抗体,又能像骨髓瘤细胞一样无限增殖,成功克服了b淋巴细胞在体外存活周期短的问题。对杂交瘤细胞进行培养,即可得到单克隆抗体。它的优点是可以制备出高纯度的单抗,并且可以进行单克隆抗体的大量生产。它的缺点是需要饲养和操作动物,操作步骤繁琐;利用杂交瘤技术生产出的单克隆抗体多为鼠源性,作为治疗性的抗体会产生人抗鼠抗体(hama)反应,在人体循环系统中很快被清除等。
[0004]
转基因老鼠技术通过转基因的手段把小鼠自身的抗体表达系统破坏掉,再引进人的抗体生成系统。这种转基因小鼠针对某种抗原就可以直接产生全人源的抗体。但它也有操作繁琐,成本高以及不能高通量筛选等缺点。
[0005]
单b细胞抗体直接对b细胞进行单细胞水平的分离,分析与筛选,从而精准、高效的筛选出分泌目标抗体分子的b细胞,再结合单细胞测序技术,即可得到目标抗体序列。但是,此技术也有成本高,技术难度也很高。从单细胞中克隆抗体基因并进行体外表达验证,工作量大、时间长,因此,该方法不适用于大通量的筛选。
[0006]
天然蛋白质是由20种氨基酸组成的,大数据分析表明,在抗体的cdr区每种氨基酸的分布是不均匀的,例如tyr,ser,gly,asp,arg的含量(或称出现的概率)比平均数高很多,因此有文献报导只用20种天然氨基酸中的少数几种来构建合成抗体文库。但这种构建策略忽视了其它含量低的氨基酸的作用,这些氨基酸虽然含量低,但在不同的情况下也起着不可忽视甚至是非常重要的作用。
[0007]
合成抗体是利用一个已知的天然抗体作为模板,通过基因工程的手段对其高可变区(即6个cdr区)进行随机突变,突变后形成一个庞大的抗体文库,文库分子一般展示在噬菌体(或其它细胞,如酵母,哺乳动物细胞)表面,以供筛选。目前,合成抗体文库的代表是德国morphosys公司的hucal文库系列,从此文库筛选出来的il23抗体已经被fda批准用于治
疗银屑病,另有更多的合成抗体的药物候选分子处于临床实验和早期开发阶段。morphosys公司的hucal文库突变策略设计精巧,但也非常复杂,不利于一般的科研人员构建,因此越来越多的研究者都在开发简便易行,但同样有效的突变策略来构建合成抗体文库。
技术实现要素:
[0008]
鉴于以上所述现有技术的缺点,本发明的目的在于提供一种合成抗体文库及其构建方法和用途,用于解决现有技术中的问题。
[0009]
为实现上述目的及其他相关目的,本发明提供一种合成抗体文库的构建方法,所述构建方法包括将模板抗体cdr的核苷酸序列突变,使突变后的所有抗体cdr中的每个位点上每种氨基酸的概率如下:
[0010]
met 0.7~1.5%;
[0011]
asn 0.7~1.5%;
[0012]
pro 0.3~2.1%;
[0013]
trp 0.6~1.4%;
[0014]
gly 9.8~14%;
[0015]
ser 7~14%;
[0016]
tyr 10~22%;
[0017]
ala、asp、glu、phe、his、ile、lys、leu、gln、arg、thr、val各3.1~7.4%;
[0018]
不含cys。
[0019]
本发明还提供一种合成抗体文库,所述合成抗体文库通过以上方法获得。
[0020]
本发明还提供编码所述合成抗体文库中的一个或多个抗体的多核苷酸或多核苷酸文库。
[0021]
本发明还提供由所述的多核苷酸编码获得的分离的多肽。
[0022]
本发明还提供所述多核苷酸或多核苷酸文库、多肽、合成抗体文库在抗体体外筛选、抗体发现中的用途。
[0023]
如上所述,本发明的合成抗体文库及其构建方法和用途,具有以下有益效果:避免了用杂交瘤方法筛选抗体,不需要操作动物或者动物细胞,方法和筛选过程简单,不需要人源化,就能够得到有治疗或检测作用的单克隆抗体。文库是体外构建的,目的分子也经体外筛选而来,不需要用到昂贵的仪器和试剂,成本大幅降低;可以进行高通量筛选,筛选周期较短,因此大大的节省了资金和人力成本。
附图说明
[0024]
图1显示为本发明的合成抗体文库构建流程图示意图(x代表本发明中的多核苷酸)。
[0025]
图2显示为本发明的阳性抗体蛋白用sds-page检测其纯度和分子量大小结果图。
[0026]
图3显示为本发明的阳性抗体蛋白经体积排阻色谱柱(sec)检测其有无聚集结果图;其中,图3-1至图3-6分别为rbd-b7、s1-a11、s-d8、s-d9、s-f12、s-g6的结果图。
[0027]
图4显示为本发明的阳性抗体蛋白用生物膜干涉实验(bli)检测其与各自抗原的亲和力结果图;其中,图4-1至图4-6分别为rbd-b7、s1-a11、s-d8、s-d9、s-f12、s-g6的结果
图。
具体实施方式
[0028]
本发明提供一种合成抗体文库的构建方法,所述构建方法包括如下步骤:将模板抗体cdr区的核苷酸序列突变,使所有抗体cdr中的每个位点上每种氨基酸的概率如下:
[0029]
met 0.7~1.5%;
[0030]
asn 0.7~1.5%;
[0031]
pro 0.3~2.1%;
[0032]
trp 0.6~1.4%;
[0033]
gly 9.8~14%;
[0034]
ser 7~14%;
[0035]
tyr 10~22%;
[0036]
ala、asp、glu、phe、his、ile、lys、leu、gln、arg、thr、val各3.1~7.4%;
[0037]
cys 0。
[0038]
所述概率是指在合成抗体文库的所有抗体的cdr中,某一种氨基酸在某一特定位点上出现的次数除以抗体的总数量,再乘以100%。例如:假设抗体文库中所有抗体的总数量为100,在100个抗体的cdrh1的第一个位点上met共出现1次,其概率即为1/100*100%=1%,其他位点上或其他cdr的概率计算方法同理。
[0039]
在一种实施方式中,met的概率选自以下任一范围:0.7~0.9%,0.9~1.1%,1.1~1.3%,1.3~1.5%。
[0040]
在一种实施方式中,asn的概率选自以下任一范围:0.7~0.9%,0.9~1.1%,1.1~1.3%,1.3~1.5%。
[0041]
在一种实施方式中,pro的概率选自以下任一范围:0.3~0.7%,0.7~1.1%,1.1~1.5%,1.5~2.1%。
[0042]
在一种实施方式中,trp的概率选自以下任一范围:0.6~0.8%,0.8~1.0%,1.0~1.2%,1.2~1.4%。
[0043]
在一种实施方式中,gly的概率选自以下任一范围:9.8~10.5%,10.5~11.0%,11.0~11.5%,11.5~12.0%,12.0~12.5%,12.5~13.0%,13.0~13.5%,13.5~14.0%。
[0044]
在一种实施方式中,ser的概率选自以下任一范围:7~9%,9~11%,11~13%,13~14%。
[0045]
在一种实施方式中,tyr的概率选自以下任一范围:10~13%,13~15%,15~17%,17~19%,19~22%。
[0046]
在一种实施方式中,ala、asp、glu、phe、his、ile、lys、leu、gln、arg、thr、val的概率选自以下任一范围:3.1~4.0%,4.0~5.0%,5.0~6.0%,6.0~7.4%。
[0047]
所述模板抗体针对的抗原种类不做具体限定。例如,在本发明的一种实施例中使用的1zvh抗体是抗溶菌酶的抗体。
[0048]
在一种实施方式中,所述模板抗体的形式选自igg、scfv、fab或f(ab
’
)2中的一种或几种。对应的,模板抗体的cdr区包括cdrl1、cdrl2、cdrl3、cdrh1、cdrh2和cdrh3。
[0049]
在一种实施方式中,所述模板抗体的形式为纳米抗体。对应的,所述纳米抗体的cdr区包括cdrh1、cdrh2和cdrh3。
[0050]
所述纳米抗体可以是天然抗体,也可以是通过基因工程合成的重组蛋白。所述天然抗体可以来源于骆驼科动物。所述骆驼科哺乳动物包括骆驼、羊驼、骆马和原驼。在一种实施方式中,所述纳米抗体选自蛋白质结构数据库(pdb)编号为1zvh的纳米抗体,1zvh是一个来源于骆驼的纳米抗体。
[0051]
利用本发明的合成抗体文库的构建方法得到的抗体的cdr长度无具体限定。
[0052]
在一种实施例中,利用所述合成抗体文库的构建方法得到的抗体的cdrh1、cdrh2和cdrh3的氨基酸长度分别为7、8和14个氨基酸。在本发明的其它实施例中,所述构建方法也可以得到cdr长度与前述举例的长度不同的抗体。
[0053]
在一种实施方式中,所述突变为定点突变。定点突变技术可以随心所欲地在已知dna序列中取代、插入或缺失一定长度的核苷酸片段。本领域技术人员均了解,定点突变的方法可以选自寡核苷酸引物介导的定点突变、pcr介导的定点突变及盒式突变,或者在前述方法上进行改进的方法例如kunkel突变法。
[0054]
在本发明的某些实施例中,采用kunkel突变法,该法中通过利用含突变碱基(即含所述多核苷酸包括的碱基)的引物组,使模板抗体的cdr区含突变碱基序列,从而产生与模板抗体有不同cdr区的各种抗体。
[0055]
在一种实施方式中,采用kunkel突变法构建合成抗体文库,所述构建方法包括以下步骤:
[0056]
1)设计含突变碱基的引物组,以模板抗体的含尿嘧啶单链dna为模板进行复制,产生异源双链dna分子;
[0057]
2)将步骤1)的异源双链dna分子转化入大肠杆菌细胞中,即获得合成抗体文库。
[0058]
本领域技术人员均了解,模板抗体的含尿嘧啶单链dna的合成为现有技术。例如通过将模板抗体的核苷酸序列克隆至噬菌体展示载体后,再转化至大肠杆菌中培养获得。
[0059]
所述含突变碱基的引物组包括多条引物,各引物的核苷酸序列不做具体限定。在一种实施方式中,所述引物的核苷酸序列如seq id no.3~5所示:
[0060]
tgtgcagcaagtggaxxxxxxxctaggctggtttcgt(seq id no.3)
[0061]
gaaggagttgctgcaxxxxxxxxtactacgccgatagc(seq id no.4)
[0062]
tactattgtgcggccxxxxxxxxxxxxxxaactactggggccaa(seq id no.5)
[0063]
其中x代表met、asn、pro、trp、gly、ser、tyr、ala、asp、glu、phe、his、ile、lys、leu、gln、arg、thr或val对应的核苷酸序列。
[0064]
在一种较佳实施例中,所述含突变碱基的引物组中的核苷酸序列对应的各氨基酸的概率如下:
[0065]
met 1%;asn 1%;pro 1%;trp 1%;gly 10%;ser 10%;tyr 16%;ala、asp、glu、phe、his、ile、lys、leu、gln、arg、thr、val各为5%;cys为0。
[0066]
经过发明人研究发现:met易氧化所以其概率不宜过高;asn易糖基化,糖基化后影响抗原的识别,其概率也不宜过高;pro易羟基化并破坏蛋白的三维结构。trp易氧化,并且概率高后易于形成过强的疏水相互作用,所以其概率不宜高;gly、ser、tyr在抗原抗体的识别中有重要作用,所以其概率较高;因为cys会形成额外的二硫键,对抗原抗体的识别的贡
献也很小,所以氨基酸混合物中不包括cys。
[0067]
在一种实施方式中,所述构建方法还包括将获得合成抗体文库后进行质控。质控的目的为看合成抗体文库是否构建成功。质控标准为突变后的抗体cdr区域的氨基酸分布若符合所述多肽中的每种氨基酸的数量分布,则合成抗体文库构建成功,若不符合则构建失败。
[0068]
在一种实施方式中,突变后的抗体cdr区域的氨基酸分布通过以下方法获得:pcr扩增文库中的分子,再对扩增产物测序,分析可得cdr区域每种氨基酸的分布。
[0069]
本发明还提供一种合成抗体文库,所述合成抗体文库通过以上方法获得。
[0070]
在一种实施方式中,所述合成抗体文库的实际库容可达3
×
109cfu以上。例如可以达到10
10
cfu、10
11
cfu或10
12
cfu以上。
[0071]
本发明的所述合成抗体文库构建的抗体的cdr长度无具体限定。
[0072]
在一种实施例中,所述合成抗体文库构建的抗体的cdrh1、cdrh2和cdrh3的氨基酸长度分别为7、8和14个氨基酸。在本发明的其它实施例中,构建文库的cdr长度可以与前述举例的长度不同。
[0073]
本发明还提供编码所述合成抗体文库中的一个或多个抗体的多核苷酸或多核苷酸文库。
[0074]
本发明还提供编码所述合成抗体文库中的所有抗体的多核苷酸或多核苷酸文库。
[0075]
所述多核苷酸的核苷酸序列不做特别限定,只要该多核苷酸编码的抗体cdr中各位点的氨基酸的概率满足以上要求即可。
[0076]
所述多核苷酸可以委托基因合成公司通过现有的化学合成技术合成。
[0077]
本发明还提供所述多核苷酸编码获得的多肽。
[0078]
本发明还提供所述多核苷酸或多核苷酸文库、所述多肽、合成抗体文库在抗体体外筛选、抗体发现中的用途。
[0079]
通过筛选本发明中的合成抗体文库,可以得到用于治疗、检测等方面的单克隆抗体分子。
[0080]
例如,所述合成抗体文库可以用于筛选抗covid19的spike全长蛋白s、片段s1或rbd的抗体。
[0081]
以下通过特定的具体实例说明本发明的实施方式,本领域技术人员可由本说明书所揭露的内容轻易地了解本发明的其他优点与功效。本发明还可以通过另外不同的具体实施方式加以实施或应用,本说明书中的各项细节也可以基于不同观点与应用,在没有背离本发明的精神下进行各种修饰或改变。
[0082]
在进一步描述本发明具体实施方式之前,应理解,本发明的保护范围不局限于下述特定的具体实施方案;还应当理解,本发明实施例中使用的术语是为了描述特定的具体实施方案,而不是为了限制本发明的保护范围;在本发明说明书和权利要求书中,除非文中另外明确指出,单数形式“一个”、“一”和“这个”包括复数形式。
[0083]
当实施例给出数值范围时,应理解,除非本发明另有说明,每个数值范围的两个端点以及两个端点之间任何一个数值均可选用。除非另外定义,本发明中使用的所有技术和科学术语与本技术领域技术人员通常理解的意义相同。除实施例中使用的具体方法、设备、材料外,根据本技术领域的技术人员对现有技术的掌握及本发明的记载,还可以使用与本
发明实施例中所述的方法、设备、材料相似或等同的现有技术的任何方法、设备和材料来实现本发明。
[0084]
纳米抗体1zvh是一个公开发表的抗溶菌酶的天然抗体,核苷酸序列如seq id no.1所示,氨基酸序列如seq id no.2所示,它的cdrh1有7个氨基酸残基(27-33),cdrh2有8个氨基酸残基(51-58),cdrh3有18个残基(93-102)。
[0085]
dvqlvesgggsvqaggslrlscaasgyiasinylgwfrqapgkeregvaavspaggtpyyadsvkgrftvsldnaentvylqmnslkpedtalyycaaarqgwyiplnsygynywgqgtqvtvss(seq id no.2)
[0086]
其中下划线处分别为cdrh1、cdrh2、cdrh3。
[0087]
实施例以已知的抗溶菌酶的纳米抗体(来自于骆驼,pdb:1zvh)作为抗体模板为例展示构建合成纳米抗体文库的过程。总体流程如下:以1zvh作为构建合成纳米单抗的基础,利用基因工程的方法,对其3个高可变区(即cdrh1,cdrh2和cdrh3)进行基因突变,将所述多核苷酸通过kunkel突变的方法,逐一插入到cdrh1的7个位点中、cdrh2的8个位点中,以及在cdrh3的99-112位区间插入13或14个多核苷酸(如图1所示),突变后的分子(3
×
109cfu)展示在丝状噬菌体表面组成一个纳米抗体文库,此文库中的每个分子具有了与原纳米抗体不一样的cdr区,从而我们有可能从此文库中筛选到结合不同抗原的抗体分子。
[0088]
实施例1模板单链dna分子的获得
[0089]
1.模板1zvh的获得
[0090]
模板1zvh野生型纳米单抗的氨基酸序列经检索蛋白质三维结构数据库(protein data bank)获得,经反向翻译获得它的编码dna序列,将此序列化学合成出来之后,克隆至噬菌体展示载体pcomb3xss的两个sfi i位点,与载体上的m13噬菌体的p3蛋白组成融合蛋白。dna测序保证序列和读码框架均无错误。
[0091]
2.1zvh的展示实验
[0092]
将上述噬菌体展示载体(1zvh/pcomb3xss)转化至大肠杆菌xl1-blue当中,用辅助噬菌体m13ko7(neb)侵染上述xl1-blue,使之分泌展示1zvh野生型纳米单抗的噬菌体颗粒。用elisa的方法检测1zvh与溶菌酶蛋白的结合来确保1zvh的展示。
[0093]
3.1zvh单链dna的制备
[0094]
1zvh/pcomb3xss的质粒dna转化大肠杆菌cj236细胞,产生插入尿嘧啶的单链dna作为基因突变文库的模板,用标准方法纯化的单链dna,用紫外分光光度计和电泳法检测所纯化的单链dna浓度和纯度。
[0095]
实施例2纳米抗体文库的构建
[0096]
1.双链杂合文库分子的制备
---
kunkel突变法
[0097]
以上述制备的单链1zvh/pcomb3xss dna为模板,加入3组含突变碱基的引物,cdrh1的突变引物如seq id no.3所示,cdrh2的突变引物如seq id no.4所示,cdrh3的突变引物如seq id no.5所示,上述引物分别与1zvh上的cdrh1,cdrh2和cdrh3配对(如图1所示),在dna聚合酶,连接酶的作用下,合成互补的、闭环的、突变好的异源双链dna分子(即文库分子)。
[0098]
2.文库的获得
[0099]
将上述合成的异源双链dna分子,用电穿孔法导入大肠杆菌tg1细胞(已预侵染辅助噬菌体m13k07),序列稀释法测定电转的效率,计算电转获得的转化子,即所建文库的库
容。经计算,库容为3
×
109cfu。
[0100]
3.文库的qc
[0101]
pcr扩增文库分子,用二代dna测序(ngs)方法对扩增结果测序,共得到20112条结果,对所有cdr区域进行分析,检查每个位点,每种氨基酸的分布是否符合设计。结果如表1,表2,表3)
[0102]
表1.cdr h1各位点氨基酸残基比例及与设计(design)之间的比较
[0103][0104][0105]
表2.cdr h2各位点氨基酸残基比例及与设计(design)之间的比较
[0106][0107]
表3.cdr h3各位点氨基酸残基比例及与设计(design)之间的比较
[0108][0109]
[0110]
表3(续)cdr h3各位点氨基酸残基比例及与设计(design)之间的比较
[0111][0112]
实施例3构建的抗体文库在抗体发现中的作用
[0113]
本文库可以成为针对不同靶标分子的抗体来源,这些抗体成为治疗性或检测性抗体的先导分子。下面以新冠covid19的spike蛋白为例,证明构建的抗体文库在抗体发现中的作用。
[0114]
1.抗原的获得
[0115]
covid19的spike全长蛋白s及片段s1和rbd均购自商业公司。
[0116]
2.文库的筛选及阳性克隆的初步验证
[0117]
分别以上述s,s1和rbd蛋白为抗原,用标准elisa方法筛选我们所建的纳米抗体文库。经过4轮筛选后,3个蛋白均有噬菌体抗体的富集。分离单克隆进行elisa的确证抗原与噬菌体抗体的结合,阳性分子送第三方公司进行dna测序。测序结果表明,针对s蛋白我们筛选到5个单克隆抗体,s1筛到1个单克隆抗体,rbd筛到3个单克隆抗体。
[0118]
3.阳性克隆的表达和纯化
[0119]
将阳性克隆分子的编码dna分别亚克隆至蛋白表达载体pet22b中,转化入大肠杆菌bl21中。标准方法进行蛋白质的表达与纯化
[0120]
4.阳性克隆的鉴定
[0121]
阳性抗体蛋白经紫外分光光度计测试浓度后用sds-page检测其纯度和分子量大小,浓度数据见表4,sds-page结果见图2。
[0122]
表4紫外分光光度计测试浓度结果
[0123]
纳米抗体名称浓度(mg/ml)体积rbd-b70.1371mls1-a110.3511mls-d30.3361mls-d80.1651mls-d90.3271mls-f120.9391mls-g60.3721ml
[0124]
阳性抗体蛋白经体积排阻色谱柱(sec)检测其有无聚集,hplc采用的是waters的acquiy acr系统,色谱柱是xbridge protein beh sec column(3.5μm,7.8mm x 300mm),1
×
pbs buffer,流速0.5ml/min。结果如图3所示,阳性抗体蛋白均无聚集。
[0125]
阳性抗体蛋白用生物膜干涉实验(bli)检测其与各自抗原的亲和力。使用octet red96仪器进行生物膜干涉法。将生物素化标记的各抗原用pbs-t(1xpbs,0.005%(v/v)tween20)稀释至终浓度为2μg/ml,稀释后的蛋白在30度下固定到链霉亲和素标签上,换到pbs-t中平衡(baseline)120s,然后与不同浓度的阳性抗体蛋白结合(association)300s,随后将传感器浸泡在pbs-t溶液中进行解离。数据处理使用的是data analysis 10.0软件,按照1:1化学计量学来拟合。阳性抗体蛋白亲和力及部分生物物理特性如图4和表5所示。
[0126]
表5阳性抗体蛋白亲和力及部分生物物理特性总结
[0127][0128]
以上的实施例是为了说明本发明公开的实施方案,并不能理解为对本发明的限制。此外,本文所列出的各种修改以及发明中方法的变化,在不脱离本发明的范围和精神的前提下对本领域内的技术人员来说是显而易见的。虽然已结合本发明的多种具体优选实施例对本发明进行了具体的描述,但应当理解,本发明不应仅限于这些具体实施例。事实上,各种如上所述的对本领域内的技术人员来说显而易见的修改来获取发明都应包括在本发明的范围内。
起点商标作为专业知识产权交易平台,可以帮助大家解决很多问题,如果大家想要了解更多知产交易信息请点击 【在线咨询】或添加微信 【19522093243】 与客服一对一沟通,为大家解决相关问题。
与客服一对一沟通,为大家解决相关问题。
此文章来源于网络,如有侵权,请联系删除
 热门咨询
热门咨询

tips


 商标分类
商标分类  商标转让
商标转让 



