使用抗MUC1*抗体的诊断方法与流程
 2021-02-02 07:02:53|
2021-02-02 07:02:53| 322|
322| 起点商标网
起点商标网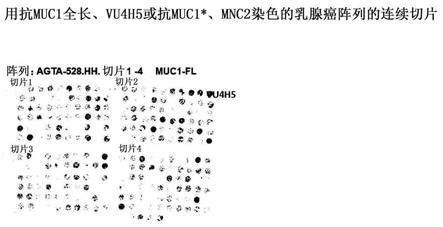
使用抗muc1*抗体的诊断方法
技术实现要素:
[0001]
本发明涉及诊断癌症和确定采用muc1*靶向治疗剂治疗以muc1异常表达为特征的癌症或癌症转移的患者的合适性的方法,包括使诊断或疑似患有癌症的患者的细胞或组织与结合无串联重复域的muc1形式的抗体接触,其中该抗体与剪切或短截形式的muc1的特异性结合的存在,以及其中这种结合为异常模式,指示muc1*靶向治疗适合用于治疗该患者。
[0002]
此处,我们将muc1*定义为muc1的跨膜剪切产物,其功能是生长因子受体并且没有串联重复序列。然而,muc1可以被剪切不同位点的不同酶剪切。何种剪切酶剪切muc1可以是组织特异性的或患者特异性的。muc1*的胞外结构域的构象可能变化,取决于哪种剪切酶对其剪切。抗muc1*抗体可以与剪切后残留的跨膜受体的胞外域结合。
[0003]
在一方面中,该抗体可以结合muc1生长因子的一级序列(psmgfr)、psmgfr n-10、psmgfr c-10的肽,或可以结合psmgfr n-10但不结合psmgfr c-10,或可以结合psmgfr c-10但不结合psmgfr n-10,或可以结合psmgfr n+20肽如n+20/c-22、n+20/c-41或n+20/c-27肽或n+9/c-9肽。该抗体可以结合具有n端延伸超出psmgfr序列的序列的肽。该抗体可以结合序列n+20-psmgfr或n+9-psmgfr的肽。在本发明的一个方面中,使用抗muc1*抗体或其片段的诊断测试用于筛选患者以确定其从muc1*靶向治疗中的潜在益处。在本发明的一个方面中,用于诊断的抗体和引入治疗的抗体或其片段衍生自相同抗体。诊断抗体和治疗抗体的种类不必是相同的。
[0004]
在一个实施例中,(i)使来自诊断或疑似癌症患者的可疑细胞或组织样本(可以是活检)与抗muc1*抗体接触;(ii)使来自患者或健康供体的正常细胞或组织样本与相同的抗muc1*抗体接触,该抗体可以是存档的参考样本;(iii)检测抗体结合;(iv)将抗体与可疑样本结合的程度和模式与正常样本比较;(v)确定可疑样本过表达muc1*或以统一的模式表达muc1*,而不是局限于顶端边界的表达,表明该患者患有muc1*阳性癌症;(vi)向患者给予加入抗muc1*抗体或其片段的治疗剂。
[0005]
在本发明的另一方面中,抗muc1*抗体可以附连于成像剂而用于患者中作为全身诊断以确定患者是否患有muc1*阳性肿瘤,或取决于特异性抗体,患者是否将从包含与显像剂附连的抗体的全部或片段的治疗剂中受益。诊断抗体和治疗性抗体的种类不必是相同的。在骆驼科动物中产生的抗体对于体内诊断分析测试是特别有用的,因为骆驼科动物产生的小单价抗体在人体内的半衰期很短。
[0006]
在本发明的另一方面中,可以附着于成像剂上的抗muc1*抗体在外科手术中用于检测或标记癌性组织,因此它们可以在手术期间切除。
[0007]
在本发明的另一方面中,使用与具有psmgfr肽的一些或全部序列的肽结合的抗muc1*抗体或其片段诊断和/或治疗乳腺癌。
[0008]
在本发明的另一方面中,使用结合至具有一些或全部的psmgfr肽序列,在n端延伸至多达20个氨基酸的肽的抗muc1*抗体或其片段诊断和/或治疗胰腺癌。
[0009]
在本发明的另一方面中,使用结合至具有一些或全部的psmgfr肽序列,在n端延伸至多达20个氨基酸的肽的抗muc1*抗体或其片段诊断和/或治疗食道癌。
[0010]
在本发明的另一方面中,使用结合至具有一些或全部的psmgfr肽序列,在n端延伸多达20个氨基酸的肽的抗muc1*抗体或其片段诊断和/或治疗前列腺癌。
[0011]
在一方面中,muc1*靶向治疗可以是癌症免疫疗法。muc1*靶向治疗剂可以是car t、bite、adc(抗体药物偶联物)、双特异性抗体或抗体模拟物。
[0012]
muc1*靶向治疗剂可以是与剪切形式的muc1结合的抗体,其中剪切形式是在剪切后保留的跨膜受体的胞外结构域。该抗体可以与称为muc1生长因子的一级序列(psmgfr)的肽结合,或与在n端延伸超出psmgfr序列至多达20个氨基酸的肽结合。治疗中使用的抗体可以源自诊断测试中使用的抗体,但不必在同一物种的动物中产生。
[0013]
本发明的方法可以是体外测试。该测试可以在组织样本、体液样本或血液样本上进行。
[0014]
在另一方面中,测试可以是体内测试。成像剂可以附连于抗体。
[0015]
在另一方面中,本发明可以包括第二抗体,并且步骤可以包括确定第一抗体与第二抗体的量的比率。第一抗体可以结合剪切后保留的跨膜受体的胞外结构域,且第二抗体可以结合muc1胞外结构域的剪切位点n端的部分,如串联重复序列。
[0016]
在另一方面中,关于所有上述方法,非人的、人类的或人源化的抗muc1*抗体或抗体片段或抗体样蛋白可以特异性地结合
[0017]
(i)muc1的psmgfr区;
[0018]
(ii)seq id no:4所示的psmgfr肽;
[0019]
(iii)psmgfr n+20/c-22,具有的氨基酸序列的肽;
[0020]
(iv)psmgfr n+12/c-22,具有的氨基酸序列的肽;
[0021]
(v)psmgfr n+9/c-30,具有的氨基酸序列的肽;
[0022]
(vi)psmgfr n+20/c-41,具有的氨基酸序列的肽;
[0023]
(vii)psmgfr n+20/c-27,具有的氨基酸序列的肽;或
[0024]
(viii)psmgfr n+9/c-9,具有的氨基酸序列的肽。
[0025]
与剪切后保留的跨膜受体的胞外域结合的抗体可以是与psmgfr n+20/c-27反应性的sdix sry多克隆抗体、mnc2单克隆抗体、mne6单克隆抗体或单克隆抗体1e4,29h1,31a1,32c1和45c11;与psmgfr n+9/c-9反应性的17h6、39h5、3c5、8a9;与psmgfr反应性的18g12、20a10、25e6、28f9和18b4,以及也与psmgfr反应性的mnc2和mne6。这些抗体可以是人类的、人源化的、鼠的、骆驼科的、美洲驼的、羊驼的、骆驼的、兔的、山羊的、仓鼠或其他非人
物种的。
[0026]
通过以下对本发明的描述、所附的附图和所附的权利要求书,将更充分地理解本发明的这些和其他目的。
附图说明
[0027]
通过以下给出的详细描述和附图,将更全面地理解本发明,且附图仅是为了举例说明而提供,因此不构成对本发明的限制。
[0028]
图1a-1d显示了根据allred评分系统的乳腺癌组织阵列的相邻连续切片的照片和病理学家评分的图形表示。病理学家评分为0-3,其中0表示未染色,且3是最大染色。图表也用颜色编码,其中病理学家评分为零是黑色,1是黄色,2是橙色,而3是红色。用识别全长muc1的抗体探测时评分为零而用识别muc1*的抗体探测时呈阳性的组织着色为绿色;且缺失或无法解释的组织评分为-1。图1a显示了乳腺癌组织阵列用vu4h5染色后的照片,vu4h5是与全长muc1的串联重复结构域结合的抗体。图1b显示了图1a所示的组织的病理学家评分的图表。图1c显示了乳腺癌组织阵列用mnc2染色后的照片,mnc2是与muc1*的psmgfr区内的表位结合的抗体。图1d显示了图1c中所示的组织的病理学家评分的图表。
[0029]
图2a-2b显示了图1a和图1c中所示的阵列的病理学家评分的饼图。图2a显示与全长muc1的串联重复结合的抗体错过了30%的乳腺癌。图2b显示抗muc1*抗体mnc2识别95%的乳腺癌。抗muc1全长仅与10%的乳腺肿瘤牢固结合,而抗muc1*抗体mnc2与约50%的乳腺肿瘤牢固结合。
[0030]
图3a-3b显示了用抗muc1*抗体humnc2-scfv-fc染色后的乳腺癌阵列br1141的病理学家评分的饼图和照片。
[0031]
图4a-4c显示了用抗muc1*抗体humnc2-scfv-fc染色后的乳腺癌阵列br1141的各个乳腺癌样本的2个不同放大倍数的照片。图中指示了阵列中的位置、癌症亚型、肿瘤等级、tnm(肿瘤分期,淋巴结转移和远端转移)和病理学家评分。使用了标准的免疫组织化学方法。使用了抗体显示正常组织的预期染色而不染色基质的最高浓度滴定抗体浓度。抗体通过其fc区缀合于生物素,以避免由于抗人二级抗体染色宿主抗体以及b细胞滤泡所致的假阳性。图4a显示了位置a7上的样本,其对于humnc2反应性细胞呈阴性。图4b显示了位置a9上的样本,其是2级癌症,具有淋巴结转移,对于humnc2反应性评分+1。图4c显示了b10位置上的样本,其是更大的2级肿瘤,具有淋巴结转移,对于humnc2反应性评分+2。
[0032]
图5a-5b显示了用抗muc1*抗体humnc2-scfv-fc染色后的来自乳腺癌阵列br1141的单个乳腺癌样本的两个不同放大倍数下的照片。图5a显示了位置d7上的样本,其是2级癌症,没有淋巴结转移,对于humnc2反应性评分+3。图5b显示了位置f6上的样本,其是2级肿瘤,具有淋巴结转移,对于humnc2反应性的评分+4。
[0033]
图6a-6b显示了用抗muc1*抗体humnc2-scfv-fc染色后的卵巢癌阵列bc1115a的病理学家评分的饼图和照片。
[0034]
图7a-7c显示了用抗muc1*抗体humnc2-scfv-fc染色后的不同癌症亚型的放大照片。图7a显示了病理学家评分+4的2级乳腺肿瘤的照片。图7b显示了病理学家评分+3的2级卵巢肿瘤的照片。图7c显示了病理学家评分+3的3级胰腺肿瘤的照片。超过1,000个肿瘤样本的ihc研究表明,humnc2-scfv识别95%的乳腺癌(90%三阴性)、83%的卵巢癌、78%的胰
腺癌和71%的肺癌。
[0035]
图8a-8d显示了用抗muc1*抗体humnc2-scfv-fc染色后的不同癌症亚型的放大照片。图8a显示了病理学家评分+2的2级乳腺肿瘤的照片。图8b显示了病理学家评分+3的3级卵巢肿瘤的照片。图8c显示了淋巴结转移病理学家评分+3的3级胰腺肿瘤的照片。图8d显示了病理学家评分+3的肺癌的照片。
[0036]
图9a-9i显示了用抗muc1*抗体humnc2-scfv-fc染色后的各种正常组织的放大照片。使用的条件和浓度与用于研究癌组织的条件和浓度相同。图9a显示了正常的肾上腺组织。图9b显示了正常的脑组织。图9c显示了正常的乳房组织。图9d显示了正常的胃组织。图9e显示了正常的心脏组织。图9f显示了正常的肾脏组织。图9g显示了正常的睾丸组织。图9h显示了正常的肠组织。图9i显示了正常肝组织。
[0037]
图10a-10f显示了用抗muc1*抗体humnc2-scfv-fc染色后的正常肾脏组织的照片。使用的条件和浓度与用于研究癌组织的条件和浓度相同。图10a显示了具有受限于顶端边界的humnc2反应性(其为正常表达)的正常肾脏组织。图10b是更大程度放大后的相同组织。图10c显示了具有不可检测的humnc2反应性的正常肾脏组织的另一个实例。图10d是更大放大后的相同组织。图10e显示了具有受限于顶端边界的humnc2反应性(其为正常表达)的正常肾脏组织的另一个实例。图10f是更大放大后的相同组织。进一步的研究表明,小于10%的正常肾脏组织在远端收集小管中显示出humnc2反应性,其中这种反应性严格限于顶端边界,其为正常表达模式。
[0038]
图11a-11b显示了用抗muc1*抗体humnc2-scfv-fc染色后的食道癌阵列bc001113的病理学家评分饼图和照片。
[0039]
图12a-12f显示了用抗muc1*抗体humnc2-scfv-fc染色后的来自食道癌阵列bc001113的单个食道癌样本的2个不同放大倍数的照片。图中指示了阵列中的位置、癌症亚型、肿瘤等级和病理学家评分。图12a显示了位置a4上的样本,其对于humnc2反应性细胞呈阴性。图12b以更大的放大倍数显示了相同样本。图12c显示了位置d2上的样本,病理学家将其评分为humnc2反应性微量。图12d以更大的放大倍数显示了相同样本。图12e显示了位置b8上的样本,病理学家对其评分humnc2反应性+1。图12f以更大的放大倍数显示了相同样本。
[0040]
图13a-13d显示了用抗muc1*抗体humnc2-scfv-fc染色后的来自食道癌阵列bc001113的单个食道癌样本的两种不同放大倍数的照片。图中指示了阵列中的位置、癌症亚型、肿瘤等级和病理学家评分。图13a显示了位置d6上的样本,其是4级肿瘤,病理学家评分为+2。图13b以更大的放大倍数显示了相同样本。图13c显示了位置d5上的样本,其是3级肿瘤,病理学家评分为+3。图12d以更大的放大倍数显示了相同样本。
[0041]
图14a-14b显示了用抗muc1*抗体humnc2-scfv-fc染色后的胰腺癌阵列pa805b的病理学家评分饼图和照片。
[0042]
图15a-15d显示了用抗muc1*抗体humnc2-scfv-fc染色后的来自胰腺癌阵列pa805b的单个胰腺癌样本的两个不同放大倍数的照片。图中显示了阵列中的位置、癌症亚型、肿瘤等级和病理学家评分。图15a显示了位置f3上的样本,其是3级肿瘤,病理学家评分为+3。图15b以更大的放大倍数显示了相同样本。图15c显示了b1位置上的样本,其是1级肿瘤,病理学家评分为+2。图15d以更大的放大倍数显示了相同样本。
[0043]
图16a-16d显示了用抗muc1*抗体humnc2-scfv-fc染色后的来自胰腺癌阵列pa805b的各个胰腺癌样本的两个不同放大倍数的照片。图中显示了阵列中的位置、癌症亚型、肿瘤等级和病理学家评分。图16a显示了位置a2上的样本,其是1级肿瘤,病理学家评分为+2。图16b以更大的放大倍数显示了相同样本。图16c显示了位置c3上的样本,其是2级肿瘤,病理学家评分为+2。图16d以更大的放大倍数显示了相同样本。
[0044]
图17a-17d显示了用抗muc1*抗体humnc2-scfv-fc染色后的来自胰腺癌阵列pa805b的各个胰腺癌样本的两个不同放大倍数的照片。图中显示了阵列中的位置、癌症亚型、肿瘤等级和病理学家评分。图17a显示了位置c6上的样本,其是2级肿瘤,病理学家评分为+2。图17b以更大的放大倍数显示了相同样本。图17c显示了d1位置上的样本,其是更大的3级肿瘤,具有淋巴结转移,病理学家评分为+3。图17d以更大的放大倍数显示了相同样本。
[0045]
图18a-18d显示了用抗muc1*抗体humnc2-scfv-fc染色后的来自胰腺癌阵列pa805b的各个胰腺癌样本的两个不同放大倍数的照片。图中显示了阵列中的位置、癌症亚型、肿瘤等级和病理学家评分。图18a显示了位置e2上的样本,其是1级肿瘤,病理学家评分为+2。图18b以更大的放大倍数显示了相同样本。图18c显示了e10位置上的样本,其是较小的3级肿瘤,具有淋巴结转移,病理学家评分为+3。图18d以更大的放大倍数显示了相同样本。
[0046]
图19显示了作为对照的仅用二级抗体染色的胰腺癌阵列pa805b的照片。
[0047]
图20a-20b显示了用抗muc1*单克隆抗体或抗muc1*多克隆抗体染色的胰腺癌阵列pa805b的照片。图20a显示了用抗muc1*单克隆抗体humnc2-scfv染色的胰腺癌阵列的照片。图20b显示了用抗muc1*多克隆抗体sdix染色的胰腺癌阵列的照片。通过用psmgfr肽免疫化动物,产生多克隆和单克隆抗体两者。标示出画圈的样本,因为用单克隆抗体相对于多克隆抗体探测时它们显示出不同的染色。用humnc2-scfv探测时,每个样本下方的数字表示病理学家评分,其后是斜线标记,然后是肿瘤等级。
[0048]
图21a-21d显示了来自胰腺癌阵列的单个肿瘤组织样本的照片,比较了当用单克隆抗体mnc2或多克隆抗体sdix探测患者样品时的染色强度和染色模式,其中两种抗体均结合psmgfr肽。在图中,(a)用mnc2染色,(b)是相同组织,但放大倍数更大,(c)用sdix染色,且(d)是相同组织,但放大倍数更大。
[0049]
图22a-22d显示了胰腺癌阵列的单个肿瘤组织样本的照片,比较了当用单克隆抗体mnc2或多克隆抗体sdix探测患者样品时的染色强度和染色模式,其中两种抗体均结合psmgfr肽。在图中,(a)用mnc2染色,(b)是相同组织,但放大倍数更大,(c)用sdix染色,(d)是相同组织,但放大倍数更大。
[0050]
图23a-23d显示了胰腺癌阵列的单个肿瘤组织样本的照片,比较了当用单克隆抗体mnc2或多克隆抗体sdix探测患者样品时的染色强度和染色模式,其中两种抗体均结合psmgfr肽。在图中,(a)用mnc2染色,(b)是相同组织,但放大倍数更大,(c)用sdix染色,(d)是相同组织,但放大倍数更大。
[0051]
图24a-24d显示了胰腺癌阵列的单个肿瘤组织样本的照片,比较了当用单克隆抗体mnc2或多克隆抗体sdix探测患者样品时的染色强度和染色模式,其中两种抗体均结合psmgfr肽。在图中,(a)用mnc2染色,(b)是相同组织,但放大倍数更大,(c)用sdix染色,(d)是相同组织,但放大倍数更大。
[0052]
图25a-25d显示了来自胰腺癌阵列的单个肿瘤组织样本的照片,比较了当用单克隆抗体mnc2或多克隆抗体sdix探测患者样品时的染色强度和染色模式,其中两种抗体均结合psmgfr肽。在图中,(a)用mnc2染色,(b)是相同组织,但放大倍数更大,(c)用sdix染色,(d)是相同组织,但放大倍数更大。
[0053]
图26a-26d显示了来自胰腺癌阵列的单个肿瘤组织样本的照片,比较了当用单克隆抗体mnc2或多克隆抗体sdix探测患者样品时的染色强度和染色模式,其中两种抗体均结合psmgfr肽。在图中,(a)用mnc2染色,(b)是相同组织,但放大倍数更大,(c)用sdix染色,(d)是相同组织,但放大倍数更大。
[0054]
图27a-27d显示了来自胰腺癌阵列的单个肿瘤组织样本的照片,比较了当用单克隆抗体mnc2或多克隆抗体sdix探测患者样品时的染色强度和染色模式,其中两种抗体均结合psmgfr肽。在图中,(a)用mnc2染色,(b)是相同组织,但放大倍数更大,(c)用sdix染色,(d)是相同组织,但放大倍数更大。
[0055]
图28a-28d显示了来自胰腺癌阵列的单个肿瘤组织样本的照片,比较了当用单克隆抗体mnc2或多克隆抗体sdix探测患者样品时的染色强度和染色模式,其中两种抗体均结合psmgfr肽。在图中,(a)用mnc2染色,(b)是相同组织,但放大倍数更大,(c)用sdix染色,(d)是相同组织,但放大倍数更大。
[0056]
图29a-29d显示了来自胰腺癌阵列的各个肿瘤组织样本的照片,比较了当用单克隆抗体mnc2或多克隆抗体sdix探测患者样品时的染色强度和染色模式,其中两种抗体均结合psmgfr肽。在图中,(a)用mnc2染色,(b)是相同组织,但放大倍数更大,(c)用sdix染色,(d)是相同组织,但放大倍数更大。
[0057]
图30a-30d显示了来自胰腺癌阵列的单个肿瘤组织样本的照片,比较了当用单克隆抗体mnc2或多克隆抗体sdix探测患者样品时的染色强度和染色模式,其中两种抗体均结合psmgfr肽。在图中,(a)用mnc2染色,(b)是相同组织,但放大倍数更大,(c)用sdix染色,(d)是相同组织,但放大倍数更大。
[0058]
图31a-31d显示了来自胰腺癌阵列的单个肿瘤组织样本的照片,比较了当用单克隆抗体mnc2或多克隆抗体sdix探测患者样品时的染色强度和染色模式,其中两种抗体均结合psmgfr肽。在图中,(a)用mnc2染色,(b)是相同组织,但放大倍数更大,(c)用sdix染色,(d)是相同组织,但放大倍数更大。
[0059]
图32a-32d显示了来自胰腺癌阵列的单个组织样本的照片,但显示的样本是正常的胰腺组织。比较了单克隆抗体mnc2与多克隆抗体sdix的染色强度和染色模式。在图中,(a)用mnc2染色,(b)是相同组织,但放大倍数更大,(c)用sdix染色,(d)是相同组织,但放大倍数更大。
[0060]
图33a-33d显示了来自胰腺癌阵列的各个组织样本的照片,但显示的样本是正常胰腺组织。比较了单克隆抗体mnc2与多克隆抗体sdix的染色强度和染色模式。在图中,(a)用mnc2染色,(b)是相同组织,但放大倍数更大,(c)用sdix染色,(d)是相同组织,但放大倍数更大。
[0061]
图34a-34d显示了来自胰腺癌阵列的单个组织样本的照片,但显示的样本是正常胰腺组织。比较了单克隆抗体mnc2与多克隆抗体sdix的染色强度和染色模式。在图中,(a)用mnc2染色,(b)是相同组织,但放大倍数更大,(c)用sdix染色,(d)是相同组织,但放大倍
数更大。
[0062]
图35a-35b显示了癌细胞和正常造血干细胞上的muc1*表达的动画。图35a描绘了通过抗muc1*单克隆抗体mnc2探测的癌细胞上的muc1*。图35b描绘了通过抗muc1*单克隆抗体mnc3探测的正常造血干细胞上的muc1*。两种抗体都是通过用psmgfr肽免疫化的动物产生的。mnc2不结合正常的造血干细胞,但mnc3会结合。
[0063]
图36a-36b显示了用抗psmgfr抗体染色的人造血干细胞的facs分析。图36a显示了facs结果的图表,显示sdix多克隆抗体和mnc3单克隆抗体识别几乎100%的造血干细胞,但mnc2单克隆抗体不与它们结合。图36b显示了facs扫描的覆盖图,其表明mnc2结合与对照抗体没有差别,而mnc3在细胞群体中产生明显的偏移。通过用psmgfr肽免疫产生了所有三种抗体。
[0064]
图37显示了添加剪切酶mmp9的催化结构域,随后用抗全长muc1抗体vu4h5或抗muc1*抗体mnc2探测之后表达90%全长muc1的细胞的facs分析的图表。
[0065]
图38a-38c列出了通过使用源自muc1*胞外结构域序列的三种不同肽中的中一种免疫化动物而产生的新抗muc1*单克隆抗体。图38a列出了当用psmgfr肽免疫化动物时产生的单克隆抗体。图38b列出了当用psmgfr n+20/c-27肽免疫化动物时产生的单克隆抗体。图38c列出了当用psmgfr n+9/c-9肽免疫化动物时产生的单克隆抗体。-1或-2的标识是指来自同一孔的姐妹克隆。给出了原液抗体溶液的浓度。
[0066]
图39显示了测试用psmgfr肽免疫化而产生的单克隆抗体结合衍生自muc1*胞外结构域序列的其他肽的能力的elisa实验的图表。首先基于它们与免疫肽结合的能力而选择所有单克隆抗体。为了进一步阐明抗体结合的该肽内的表位,测试了抗体结合psmgfr肽、n-10肽或c-10肽的能力。
[0067]
图40a-40b显示了与t47d乳腺癌细胞结合的新psmgfr抗muc1*单克隆抗体的facs分析的图表。图40a显示了平均荧光强度。图40b显示了用各自抗体染色为阳性的细胞的百分比。
[0068]
图41显示了测试通过用psmgfr n+20/c-27肽免疫化而产生的单克隆抗体结合衍生自muc1*胞外结构域序列的其他肽的能力的elisa实验的图表。首先基于它们与免疫肽结合的能力而选择所有单克隆抗体。为了进一步阐明与抗体结合的该肽内的表位,测试了抗体与psmgfr肽、n-10肽或c-10肽结合的能力。
[0069]
图42a-42b显示了结合t47d乳腺癌细胞的新psmgfr n+20/c-27抗muc1*单克隆抗体的facs分析的图表。图42a显示了平均荧光强度。图42b显示了用各自抗体染色呈阳性的细胞的百分比。
[0070]
图43显示了测试通过用psmgfr n+9/c-9肽免疫化而产生的单克隆抗体结合衍生自muc1*胞外结构域序列的其他肽的能力的elisa实验的图表。首先基于它们与免疫肽结合的能力而选择所有单克隆抗体。为了进一步阐明与抗体结合的该肽内的表位,测试了抗体与psmgfr肽、n-10肽或c-10肽结合的能力。
[0071]
图44a-44b显示了与t47d乳腺癌细胞结合的新psmgfr n+9/c-9抗muc1*单克隆抗体的facs分析图。图44a显示了平均荧光强度。图44b显示了用各自抗体染色呈阳性的细胞的百分比。
[0072]
图45a-45c显示了通过标准ihc方法用各种抗muc1*抗体染色的来自胰腺癌阵列的
相邻连续切片的照片。图45a显示了用sdix多克隆抗muc1*抗体染色的阵列,其中该抗体的免疫原是psmgfr肽。图45b显示了用18b4单克隆抗muc1*抗体染色的阵列,其中该抗体的免疫原是psmgfr肽。图45c显示了用1e4单克隆抗muc1*抗体染色的阵列,其中该抗体的免疫原是psmgfr n+20/c-27肽。
[0073]
图46a-46f显示了图45所示的胰腺癌阵列的各个样品的照片。图46a显示了用sdix多克隆抗体染色的样品。图46b显示了更大放大倍数的相同组织样本。图46c显示了用18b4单克隆抗体染色的相邻组织切片。图46d显示了更大放大倍数的相同组织样本。图46e显示了用1e4单克隆抗体染色的相邻组织切片。图46f显示了更大放大倍数的相同组织样本。
[0074]
图47a-47d显示了图45所示的胰腺癌阵列的各个样本的照片。图47a显示了用sdix多克隆抗体染色的样本。图47b显示了更大放大倍数的相同组织样本。图47c显示了用18b4单克隆抗体染色的相邻组织切片。图47d显示了更大放大倍数的相同组织样本。
[0075]
图48a-48d显示了图45所示的胰腺癌阵列的各个样本的照片。图48a显示了用sdix多克隆抗体染色的样本。图48b显示了更大放大倍数的相同组织样本。图48c显示了用18b4单克隆抗体染色的相邻组织切片。图48d显示了更大放大倍数的相同组织样本。
[0076]
图49a-49d显示了来自图45所示的胰腺癌阵列的各个样本的照片。图49a显示了用sdix多克隆抗体染色的样本。图49b显示了更大放大倍数的相同组织样本。图49c显示了用1e4单克隆抗体染色的相邻组织切片。图49d显示了更大放大倍数的相同组织样本。图49a与图49c比较,显而易见的是,通过psmgfr n+20/c-27肽免疫化而产生的单克隆抗体识别的肿瘤细胞数量不同于通过用psmgfr肽免疫化而产生的多克隆抗体sdix识别的细胞数量。
[0077]
图50a-50d显示了来自图45所示的胰腺癌阵列的各个样本的照片。图50a显示了用sdix多克隆抗体染色的样本。图50b显示了更大放大倍数的相同组织样本。图50c显示了用1e4单克隆抗体染色的相邻组织切片。图50d显示了更大放大倍数的相同组织样本。比较图50a和图50c,显而易见的是,通过用psmgfr n+20/c-27肽免疫化产生的单克隆抗体识别的肿瘤细胞数量不同于通过用psmgfr肽免疫化而产生的多克隆抗体sdix识别的细胞数量。
[0078]
图51a-51c显示了通过标准ihc方法用各种抗muc1*抗体染色的胰腺癌阵列的相邻连续切片的照片。图51a显示了用sdix多克隆抗muc1*抗体染色的阵列,其中该抗体的免疫原是psmgfr肽。图51b显示了用20a10单克隆抗muc1*抗体染色的阵列,其中该抗体的免疫原是psmgfr肽。图51c显示了用29h1单克隆抗muc1*抗体染色的阵列,其中该抗体的免疫原是psmgfr n+20/c-27肽。
[0079]
图52a-52d显示了通过标准ihc方法用各种抗muc1*抗体染色的胰腺癌阵列的相邻系列切片的照片。图52a显示了用17h6单克隆抗muc1*抗体染色的阵列,其中抗体的免疫原是psmgfr n+9/c-9肽。图52b显示了用32c1单克隆抗muc1*抗体染色的阵列,其中抗体的免疫原是psmgfr n+20/c-27肽。图52c显示了用45c11单克隆抗muc1*抗体染色的阵列,其中该抗体的免疫原是psmgfr n+20/c-27肽。图52d显示了用31a1单克隆抗muc1*抗体染色的阵列,其中抗体的免疫原是psmgfr n+20/c-27肽。
[0080]
图53a-53f显示了通过标准ihc方法用识别全长muc1的抗体或仅识别muc1*的抗体染色的来自胰腺癌阵列的相邻连续切片的照片和病理学家染色评分的图形表示。图53a显示了用抗体5e5染色的胰腺癌阵列,抗体是与全长muc1的串联重复结构域中的捕获o连接聚糖结合的抗体。图53b显示了该阵列中每个样本的病理学家评分。图53c显示了用抗muc1*抗
体29h1染色的胰腺癌阵列,该抗体是与muc1*的psmgfr n+20/c-27肽结合的抗体。图53d显示了阵列中每个样本的病理学家评分。图53e显示了用抗体vu4h5染色的胰腺癌阵列,该抗体是与全长muc1的串联重复结构域中的表位结合的抗体。图53f显示了所述阵列中每个样本的病理学家评分。如该图所示,抗体5e5识别vu4h5不识别的一些样本,然而,抗muc1*抗体29h1会识别由识别全长muc1的两种抗体识别的样本以及未被任一种抗muc1抗体识别的其它样本。这些发现表明,与包含n端延伸超出psmgfr序列的氨基酸的肽结合的抗muc1*抗体不能识别全长muc1,并且与psmgfr n+20/c-27结合的抗体可以识别胰腺癌上普遍存在的表位。
[0081]
图54a-54c显示了通过标准ihc方法用各种抗muc1*抗体染色的来自食道癌阵列的相邻连续切片的照片。图54a显示了用sdix多克隆抗muc1*抗体染色的阵列,其中抗体的免疫原是psmgfr肽。图54b显示了用20a10单克隆抗muc1*抗体染色的阵列,其中该抗体的免疫原是psmgfr肽。图54c显示了用29h1单克隆抗muc1*抗体染色的阵列,其中该抗体的免疫原是psmgfr n+20/c-27肽。图54d显示了用31a1单克隆抗muc1*抗体染色的阵列,其中该抗体的免疫原是psmgfr n+20/c-27肽。该图表明,尽管结合程度不同,但都与psmgfr肽结合的sdix和20a10抗体可以识别相同肿瘤组织样本,而与psmgfr n+20/c-27肽结合的抗体也可以结合更多食道肿瘤样本以及由抗psmgfr抗体识别的那些中的大多数。这些结果与以下观点相吻合:结合psmgfr n+20/c-27肽的抗体通常比结合psmgfr肽的抗体对食道癌更具特异性,但某些患者可能患有被结合psmgfr肽的抗muc1*抗体更好识别的食道癌。
[0082]
图55a-55c显示了通过标准ihc方法用各种抗muc1*抗体染色的来自食道癌阵列的相邻连续切片的照片。图55a显示了用sdix多克隆抗muc1*抗体染色的阵列,其中该抗体的免疫原是psmgfr肽。图55b显示了用17h6单克隆抗muc1*抗体染色的阵列,其中该抗体结合psmgfr n+9/c-9肽。
[0083]
图55c显示了用mnc2单克隆抗muc1*抗体染色的阵列,其中该抗体结合psmgfr肽。图55d显示了用45c11单克隆抗muc1*抗体染色的阵列,其中该抗体结合psmgfr n+20/c-27肽。这些结果与以下想法一致:对于大多数食道癌,muc1被暴露psmgfr序列的n端的隐蔽表位的酶剪切。
[0084]
图56a-56f显示了通过标准ihc方法用识别全长muc1的抗体或仅识别muc1*的抗体染色的来自食道癌阵列的相邻连续切片的照片和病理学家染色分数的图形表示。图56a显示了用抗体5e5染色的食道癌阵列,该抗体是结合全长muc1的串联重复结构域中的捕获的o连接聚糖的抗体。图56b显示了阵列中每个样品的病理学家评分。图56c显示了用抗muc1*抗体29h1染色的食道癌阵列,该抗体是结合muc1*的psmgfr n+20/c-27肽的抗体。图56d显示了阵列中每个样本的病理学家评分。图56e显示了用抗体vu4h5染色的食道癌阵列,该抗体是结合全长muc1的串联重复结构域中的表位的抗体。图56f显示了阵列中每个样本的病理学家评分。如该图所示,抗体5e5会识别vu4h5不识别的一些样本,然而,抗muc1*抗体29h1识别被两种识别全长muc1的抗体识别的样本以及不被任一种抗muc1抗体识别的其它样本。这些发现表明,结合包含n端延伸超出psmgfr序列的氨基酸的肽的抗muc1*抗体不识别全长muc1,而结合psmgfr n+20/c-27肽的抗体识别食道癌上普遍存在的表位。
[0085]
图57a-57g显示了用均识别全长muc1的抗体5e5或vu4h5或仅识别muc1*并结合psmgfr n+20/c-27肽的29h1染色的前列腺癌阵列的照片。图57a显示了用抗体5e5染色的食
道癌阵列。图57b显示了用抗体29h1染色的食道癌阵列。图57b显示了用抗体29h1染色的食道癌阵列。图57c显示了用抗体vu4h5染色的食道癌阵列。图57d显示仅用二抗染色作为对照的食道癌阵列。图57e显示了更大放大倍数下的图57a中用红色框标记的组织,其中染色采用5e5。图57f显示了更大放大倍数下的图57b中用红色框标记的组织,其中染色采用29h1。图57g显示了更大放大倍数下的图57c中用红色框标记的组织,其中染色采用vu4h5。红色虚线框表示许多食道肿瘤样本中对识别全长muc1的抗体染色呈阴性但在用抗muc1*抗体(尤其是结合psmgfr n+20/c-27肽的抗体)探测时高度阳性的唯一患者样本。
[0086]
图58a-58c显示了通过标准ihc方法用各种抗muc1*抗体染色的前列腺癌阵列的相邻连续切片的照片。图58a显示了用sdix多克隆抗muc1*抗体染色的阵列,其中抗体的免疫原是psmgfr肽。图58b显示了用18b4单克隆抗muc1*抗体染色的阵列,其中该抗体结合psmgfr肽。图58c显示了用1e4单克隆抗muc1*抗体染色的阵列,其中该抗体结合psmgfr n+20/c-27肽。
[0087]
图59a-59e显示了通过标准ihc方法用各种抗muc1*抗体染色的来自前列腺癌阵列的相邻连续切片的照片。图59a显示了用结合psmgfr肽而不结合c-10肽的mnc2单克隆抗体染色的阵列。图59b显示了用结合psmgfr肽的18b4抗体染色的阵列。图59c显示了用结合psmgfr n+20/c-27肽的32c1抗体染色的阵列。图59d显示了用sdix多克隆抗muc1*抗体染色的阵列,其中该抗体的免疫原是psmgfr肽。图59e显示了用结合psmgfr n+20/c-27肽的31a1单克隆抗muc1*抗体染色的阵列。
[0088]
图60a-60f显示了通过标准ihc方法用识别全长muc1的抗体或仅识别muc1*的抗体染色的来自前列腺癌阵列的相邻连续切片的照片和病理学家染色评分的图形表示。图60a显示了用抗体5e5染色的前列腺癌阵列,该抗体结合全长muc1的串联重复结构域中的捕获的o连接聚糖。图60b显示了阵列中每个样品的病理学家评分。图60c显示了用抗muc1*抗体29h1染色的前列腺癌阵列,该抗体是结合muc1*的psmgfr n+20/c-27肽的抗体。图60d显示了阵列中每个样本的病理学家评分。图60e显示了用抗体vu4h5染色的前列腺癌阵列,该抗体是结合全长muc1的串联重复结构域中的表位的抗体。图60f显示了阵列中每个样本的病理学家评分。如该图所示,抗体5e5可以识别vu4h5不识别的一些样本,然而,抗muc1*抗体29h1可以识别被两种识别全长muc1的抗体识别的样本以及不被任一种抗muc1抗体识别的其它样本。这些发现表明,结合包含n端超出psmgfr序列的氨基酸的肽的抗muc1*抗体不能识别全长muc1,而结合psmgfr n+20/c-27的抗体肽识别前列腺癌中普遍存在的表位。
[0089]
图61a-61g显示了用均识别全长muc1的的抗体5e5或vu4h5或仅识别muc1*并结合psmgfr n+20/c-27肽的29h1染色的前列腺癌阵列的照片。图61a显示了用抗体5e5染色的前列腺癌阵列。图61b显示了用抗体29h1染色的前列腺癌阵列。图61b显示了用抗体29h1染色的前列腺癌阵列。图61c显示了用抗体vu4h5染色的前列腺癌阵列。图61d显示了作为对照的仅用二抗体染色的前列腺癌阵列。图61e显示了更大放大倍数的图61a中用红色框标记的组织,其中染色采用5e5进行。图61f显示了更大放大倍数的图61b中用红色框标记的组织,其中染色采用29h1进行。图61g显示了更大放大倍数的图61c中用红色框标记的组织,其中染色采用vu4h5进行。红色虚线框表示许多前列腺肿瘤样本中对识别全长muc1的抗体呈阴性而在用抗muc1*抗体而尤其是结合psmgfr n+20/c-27肽的那些抗体探测时呈高度阳性的唯一患者的样本。
[0090]
图62a-62b显示了用两种不同的抗muc1*单克隆抗体染色的乳腺癌阵列br1141的相邻连续切片的照片。图62a显示了用抗muc1*单克隆抗体mnc2染色的乳腺癌阵列的照片。图62b显示了用抗muc1*单克隆抗体20a10染色的乳腺癌阵列的照片。两种单克隆抗体均结合psmgfr肽、n-10肽但不结合c10肽。
[0091]
图63a-63b显示了用两种不同抗muc1*单克隆抗体染色的乳腺癌阵列br1141的相邻连续切片的照片。图63a显示了用抗muc1*单克隆抗体mnc2染色的乳腺癌阵列的照片。图63b显示了用抗muc1*单克隆抗体25e6染色的乳腺癌阵列的照片。两种单克隆抗体均结合psmgfr肽和n-10肽。mnc2无法与c-10肽结合,而25e6显示出与c-10肽较低水平的结合,表明它们可以结合不同的表位。
[0092]
图64a-64b显示了用两种不同的抗muc1*单克隆抗体染色的乳腺癌阵列br1141的相邻连续切片的照片。图64a显示了用抗muc1*单克隆抗体mnc2染色的乳腺癌阵列的照片。图64b显示了用抗muc1*单克隆抗体18b4染色的乳腺癌阵列的照片。两种单克隆抗体均结合psmgfr肽。然而,与mnc2不同,18b4不能结合n-10表位,表明它们可以结合不同表位,并且18b4可能需要psmgfr肽的10个n端氨基酸以结合。
[0093]
图65a-65b显示了用两种不同的抗muc1*单克隆抗体染色的乳腺癌阵列br1141的相邻连续切片的照片。图65a显示了用抗muc1*单克隆抗体mnc2染色的乳腺癌阵列的照片。图65b显示了用抗muc1*单克隆抗体18g12染色的乳腺癌阵列的照片。两种单克隆抗体均结合psmgfr肽。然而,与mnc2不同,18g12一定程度与c-10表位结合,表明它们可以结合psmgfr肽内的不同表位。
[0094]
图66a-66b显示了用两种不同的抗muc1*单克隆抗体染色的乳腺癌阵列br1141的相邻连续切片的照片。图66a显示了用抗muc1*单克隆抗体mnc2染色的乳腺癌阵列的照片。图66b显示了用抗muc1*单克隆抗体8a9染色的乳腺癌阵列的照片。单克隆抗体mnc2可以结合psmgfr肽,而8a9可以结合psmgfr n+9/c-9肽。它们结合的肽以及非常不同的染色模式表明它们结合不同的muc1*表位。
[0095]
图67a-67b显示了用两种不同的抗muc1*单克隆抗体染色的乳腺癌阵列br1141的相邻连续切片的照片。图67a显示了用抗muc1*单克隆抗体mnc2染色的乳腺癌阵列的照片。图67b显示了用抗muc1*单克隆抗体28f9染色的乳腺癌阵列的照片。两种单克隆抗体均结合psmgfr肽。
[0096]
图68a-68b显示了用两种不同的抗muc1*单克隆抗体染色的乳腺癌阵列br1141的相邻系列切片的照片。图68a显示了用抗muc1*单克隆抗体mnc2染色的乳腺癌阵列的照片。图68b显示了用抗muc1*单克隆抗体17h6染色的乳腺癌阵列的照片。单克隆抗体mnc2结合psmgfr肽,而17h6结合psmgfr n+9/c-9肽。它们结合的肽以及非常不同的染色模式表明它们结合不同的muc1*表位。
[0097]
图69a-69b显示了用两种不同的抗muc1*单克隆抗体染色的乳腺癌阵列br1141的相邻连续切片的照片。图69a显示了用抗muc1*单克隆抗体mnc2染色的乳腺癌阵列的照片。图69b显示了用抗muc1*单克隆抗体3c5染色的乳腺癌阵列的照片。单克隆抗体mnc2结合psmgfr肽,而3c5结合psmgfr n+9/c-9肽。它们结合的肽以及非常不同的染色模式表明它们结合不同的muc1*表位。
[0098]
图70a-70g显示了用四种不同的抗muc1*单克隆抗体染色的乳腺癌阵列br1007的
相邻连续切片的照片。图70a显示了用结合psmgfr肽的抗muc1*单克隆抗体20a10染色的乳腺癌阵列的照片。图70b显示了用结合psmgfr n+20/c-27肽的抗muc1*单克隆抗体29h1染色的乳腺癌阵列的照片。图70c显示了用结合psmgfr n+20/c-27肽的抗muc1*单克隆抗体45c11染色的乳腺癌阵列的照片。图70d显示了用结合psmgfr n+20/c-27肽的抗muc1*单克隆抗体320染色的乳腺癌阵列的照片。图70e显示了用结合psmgfr肽的抗muc1*单克隆抗体18b4染色的乳腺癌阵列的照片。图70f显示了用结合psmgfr n+20/c-27肽的抗muc1*单克隆抗体31a1染色的乳腺癌阵列的照片。图70g显示了用结合psmgfr n+9/c-9肽的抗muc1*单克隆抗体17h6染色的乳腺癌阵列的照片。
[0099]
图71a-71f显示了通过标准ihc方法用识别全长muc1的抗体或仅识别muc1*的抗体染色的来自乳腺癌阵列的相邻连续切片的照片和病理学家染色评分的图形表示。图71a显示了用结合全长muc1的串联重复结构域中的捕获的o连接聚糖的抗体5e5染色的乳腺癌阵列。图71b显示了阵列中每个样本的病理学家评分。图71c显示了用结合muc1*的psmgfr n+20/c-27肽的抗muc1*抗体29h1染色的乳腺癌阵列。图71d显示了阵列中每个样本的病理学家评分。图71e显示了用结合全长muc1的串联重复结构域中的表位的抗体vu4h5染色的乳腺癌阵列。图71f显示了阵列中每个样本的病理学家评分。如该图所示,抗体5e5识别vu4h5不能识别的一些样本,然而,抗muc1*抗体29h1识别被两种识别全长muc1的抗体识别的样本以及不能被任一种抗muc1抗体识别的其它样本。这些发现表明,结合包括n端延伸超出psmgfr序列的氨基酸的肽的抗muc1*抗体不能识别全长muc1。
[0100]
图72a-72f显示了用各种抗muc1*单克隆抗体染色的乳腺癌组织阵列br1141的相邻连续切片的照片,其中所有抗体都结合psmgfr肽。图72a显示了用mnc2染色的乳腺癌样本。图72b显示了用20a10染色的乳腺癌样本。图72c显示了用25e6染色的乳腺癌样本。图72d显示了用28f9染色的乳腺癌样本。图72e显示了用18g12染色的乳腺癌样本。图72f显示了用18b4染色的乳腺癌样本。所有这些抗体均结合psmgfr肽,并大致产生相同染色模式的这种乳腺癌阵列。然而,这些抗体在阵列中识别各个样本的方式存在一些差异,这可能代表不同酶对muc1至muc1*的剪切。参照图39,mnc2和20a10结合n-10肽而不结合c-10肽,表明10个膜近端氨基酸对于它们的结合是重要的。抗体18b4、18g12和25e6显示出与c-10肽的一定程度结合,而28f9显示出与c-10肽的更多结合。值得注意的是,18b4不结合n-10肽,表明其相比psmgfr中其他结合更加n端的表位。红色圆圈表示用于比较的所关注的样本。
[0101]
图73a-73f显示了用各种抗muc1*单克隆抗体染色的乳腺癌组织阵列br1141的相邻连续切片的照片,其中结合psmgfr n+9/c-9肽的抗体与mnc2及其人源化单链形式humnc2-scfv-fc比较,二者均结合psmgfr n-10,但不结合c-10肽。图73a显示了用mnc2染色的乳腺癌样本。图73b显示了用8a9染色的乳腺癌样本。图73c显示了用17h6染色的乳腺癌样本。图73d显示了用humnc2-scfv-fc染色的乳腺癌样本。图73e显示了用3c5染色的乳腺癌样本。图73f显示了用39h5染色的乳腺癌样本。现在参考用红色圆圈标记的患者样本,容易看出,结合psmgfr n+9/c-9肽的抗体识别mnc2抗psmgfr抗体错过或结合较弱的乳腺癌细胞的群体。
[0102]
除了单克隆抗体mnc2、mne6、mnc3、mnc8和18b4,本申请中公开的18g12、20a10、25e6、1e4、29h1、31a1、32c1、45c11、3c5、8a9、17h6和39h5之外,其他单克隆抗体序列引述于由接种psmgfr肽制成的seq id nos:237-349中。
具体实施方式
[0103]
在本申请中,“一个”和“一种”用于指单个和多个对象。
[0104]
如本文所用,有时简而言之,多肽被指示为被“转导或转染”到细胞中。在这些情况下,应该理解的是,编码多肽序列的核酸被转导或转染到细胞中,因为将多肽转导或转染到细胞中是不可能的。
[0105]
如本文所用,有时在提及注入动物的细胞数,或另外在上下文中提及细胞数时,“m”是指百万,而“k”是指千。
[0106]
如本文所用,使用了各种单克隆抗体的可互换名称,例如“mn-c2”,其可以与“c2”、“min-c2”和“mnc2”互换;“mn-e6”,可以与“e6”、“min-e6”和“mne6”互换;“mn-c3”,可以与“c3”、“min-c3”和“mnc3”互换;而“mn-c8”,可以与“c8”、“min-c8”和“mnc8”互换。
[0107]
如本文所用,置于抗体构建体之前的“h”或“hu”是人或人源化的简写。
[0108]
如本文所用,术语“抗体样”是指可以工程化而使其包含部分抗体但不是自然中天然存在的抗体的分子。实例包括,但不限于,car(嵌合抗原受体)t细胞技术和技术。car技术使用与部分t细胞融合而使身体免疫系统直接攻击特异性靶蛋白或细胞的抗体表位。技术由“抗体样”库构成,该库是合成人fab的集合,然后针对与靶蛋白肽表位的结合进行筛选。然后可以将所选定的fab区工程化成支架或框架,而使其类似于抗体。
[0109]
如本文所用,“psmgfr”是muc1生长因子受体的一级序列的缩写,其由seq id no:4标识,而因此不会与六个氨基酸序列混淆。“psmgfr肽”或“psmgfr区”是指引入muc1生长因子受体的一级序列(seq id no:4)的肽或区。
[0110]
如本文所用,术语“psmgfr”是muc1生长因子受体的一级序列的首字母缩写,其表示为。在此方面,“n-10psmgfr”、“n-15psmgfr”或“n-20psmgfr”中的“n-数字”是指在psmgfr的n端缺失的氨基酸残基的数目。同样地,“n+10psmgfr”、“n+15psmgfr”或“n+20psmgfr”是指在psmgfr的n端添加的氨基酸残基的数目。同样地,“c-10psmgfr”、“c-15psmgfr”或“c-20psmgfr”中的“c-数目”是指在psmgfr的c端缺失的氨基酸残基的数量,而“c+10psmgfr”、“c+15psmgfr”或“c+20psmgfr”是指在psmgfr的c端添加的氨基酸残基数。此外,组合也是可能的,如“n+20/c-27psmgfr”、“psmgfr n+20/c-27”或“n+20/c-27”,它们是指相同的肽,其中psmgfr的n端包含muc1肽的20个其他氨基酸,而在psmgfr的c端缺失了27个氨基酸。
[0111]
如本文所用,当期望指代psmgfr肽的属时,它们称为“psmgfr组”。例如,“n+20psmgfr组”是指在n端具有另外20个氨基酸的肽,而与c端如何修饰,是否删除氨基酸或添加氨基酸等无关。
[0112]
如本文所用,“muc1*的胞外结构域”是指muc1蛋白的缺少串联重复结构域的胞外部分。在大多数情况下,muc1*是剪切产物,其中muc1*部分由没有串联重复的短胞外结构域、跨膜结构域和细胞质尾部组成。muc1剪切的精确位置可能未知,也许是因为其似乎可以被一种以上的酶剪切。muc1*的胞外结构域将包括大多数psmgfr序列,但可能具有额外的10-20个n端氨基酸。
[0113]
如本文所用,“muc1*”胞外结构域主要由psmgfr序列
定义。因为muc1剪切的确切位点取决于剪切它的酶,并且剪切酶根据细胞类型、组织类型或细胞进化的时间而变化,muc1*胞外结构域的确切序列在n端上可能会有所不同。
[0114]
其他剪切的氨基酸序列可以包括其他剪切的氨基酸序列可以包括
[0115]
如本文所用,“序列一致性”是指具体的多肽或核酸的序列与核酸或氨基酸的参考序列的同源性,使得同源肽的功能与参考肽或核酸的功能相同。这样的同源性可以与参考肽非常接近,使得有时两个序列可以是90%,95%或98%相同,还在结合或其他生物学活性中具有相同功能。
[0116]
如本文所用,“muc1阳性”细胞是指表达muc1、muc1-y或muc1-z或其他muc1变体的基因的细胞。
[0117]
如本文所用,“muc1阴性”细胞是指不表达muc1基因的细胞。
[0118]
如本文所用,“muc1*阳性”细胞是指表达muc1基因的细胞,其中该基因的表达蛋白是没有串联重复的跨膜蛋白,这可能是翻译后修饰、剪切、使用不含串联重复的muc1蛋白选择性剪接,转染或转导细胞的结果。
[0119]
如本文所用,“muc1*阴性”细胞是指可能表达或可能不表达muc1的基因但不表达缺乏串联重复的muc1跨膜蛋白的细胞。
[0120]
如本文所用,“muc1阳性”癌细胞是指过表达muc1的基因、以异常模式表达muc1的癌细胞,其中其表达不限于顶端边界和/或表达没有串联重复的muc1。
[0121]
如本文所用,“muc1阴性”癌细胞是指可能表达或可能不表达muc1的基因但不过表达muc1或不过表达没有串联重复的muc1跨膜蛋白的癌细胞。
[0122]
如本文所用,“muc1*阳性”癌细胞是指过表达没有串联重复的muc1跨膜蛋白的癌细胞。
[0123]
如本文所用,“muc1*阴性”癌细胞是指可能表达或可能不表达muc1的基因但不过表达没有串联重复的muc1跨膜蛋白的癌细胞。
[0124]
一般而言,本发明涉及与癌症有关的诊断测试,该癌症的特征在于一类细胞表面受体的异常表达,其特征在于癌组织中的链间结合区或胞外结构域的剪切增加。这样的一组癌症是特征在于粘蛋白家族蛋白如muc1、muc2、muc3、muc4直至并包括muc16的异常表达的那些。本文中本发明的许多描述涉及异常表达muc1的细胞和组织,作为与癌症有关的更大类的蛋白的实例,其具有在癌症中越来越多地剪切的胞外域和/或具有链间结合区(ibr)。应当理解的是,在这些情况下,该描述应被认为是示例性的,并且本发明的原理适用于通过相似机制发挥功能的其他跨膜蛋白。根据本文的公开内容,本领域普通技术人员应容易鉴定通过该机制或类似机制发挥功能的其他跨膜蛋白,并将本发明应用于那些以受体异常表达为特征的癌症。本发明基于涉及具有其自身胞外结构域会自我聚集和/或渐增受切裂的区域的跨膜蛋白的新机制,例如muc1,本发明人已对其进行了阐述。
[0125]
muc1包括在本文中称为以下的几个区。从细胞内部的c端到细胞外部的n端,muc1蛋白由以下结构域组成:1)胞质尾区;2)跨膜段;3)mgfr;4)ibr(链间结合区);5)ur(独特
区);和6)串联重复。
[0126]
我们先前发明的一方面的特征在于发现了muc1受体的特定区域,即ibr,与其他muc1分子的相同区强烈结合。即muc1受体具有经由各自受体的ibr与其他muc1受体聚结的能力(即,自聚结)。进行金纳米颗粒实验表明,ibr与自身聚结,其可以阻碍配体与muc1或其酶切产物muc1*的结合。ibr和mgfr之间的边界取决于muc1在何处被剪切(其由哪种剪切酶对其剪切决定)而变化。
[0127]
在健康细胞中观察到,这种自聚结可能有助于muc1受体簇聚。muc1受体的ibr部分自聚结的发现与以下机制模型一致,本发明人为此提供了支持证据。(1)受体聚结与健康状态有关,因为聚结的ibr部分阻止配体如生长因子、修饰酶等接近充当功能受体的muc1受体的相邻胞外部分;簇聚还阻止细胞内尾区接近胞内修饰酶和信令配体;(2)当muc1受体在释放部分或全部自聚结部分的位置被剪切时,保持受体聚结的临界力消失,且受体在细胞膜内自由迁移或与修饰酶、分泌的配体如活化配体或生长因子或其他细胞表面受体相互作用。这些相互作用涉及触发细胞增殖信令级联的新的诱导性多聚状态,如二聚化。
[0128]
muc1的剪切释放大块胞外结构域,包括串联重复结构域,并留下具有至少包含psmgfr区的截短的胞外结构域的跨膜蛋白。串联重复结构域本体的剪切和释放,暴露出与截短的胞外结构域结合并使其二聚的配体的结合位点,从而激活了生长和存活途径。我们称muc1酶切产物为“muc1*”。
[0129]
muc1*是一种生长因子受体,其通过其截短的胞外结构域的配体诱导二聚而激活。结合psmgfr肽的二价抗体,是muc1的膜近端部分的45个氨基酸序列,使muc1*二聚并刺激生长。抗psmgfr抗体以浓度依赖性方式刺激t47d muc1阳性癌细胞的生长。在类似的实验中,一定浓度的确定为最大化癌细胞增殖的抗psmgfr抗体添加到如上描述的生长的第一组t47d肿瘤细胞中。相同量的抗psmgfr抗体添加到一组对照细胞,k293细胞中。向muc1肿瘤细胞(t47d)添加抗psmgfr抗体24小时内增强增殖180%,但对对照细胞无影响。
[0130]
使muc1*的胞外结构域二聚的配体会诱导细胞的生长和存活。我们确定的muc1*配体有nme1、nme2、nme6、nme7-ab和其他剪接变体nme7-x1。
[0131]
muc1*是驱动癌细胞生长的生长因子受体,而全长muc1则不是。因此,检测到高于正常水平的muc1*的量是癌症的指标,并且muc1*的量越高,癌症越糟。muc1的剪切可能发生于一个以上的位点,具体取决于肿瘤表达的是哪种剪切酶。muc1的剪切会释放包含串联重复序列的胞外域部分,并且取决于剪切位点,可能会包含部分独特区域或部分ibr。通过测量保留于细胞或组织上的全长muc1的量,可以推断已剪切的muc1的量。这可以通过使细胞或组织与结合串联重复体的抗体或独特区域或ibr接触而实现。与串联重复结构域结合的抗体是能够结合具有序列的肽的抗体。结合串联重复结构域的常用抗体包括但不限于vu4h5(santa cruz biotechnology,dallas texas cat.no.sc-7313),hmpv,5e5(sorensen et al.,glycobiology,vol.16,no.2,pp.96-107,2006),pr81和ldq10。在这些情况下,与表达于相同细胞或组织上的muc1*的量相比,测量全长muc1的量最有效。muc1*与全长muc1的比率是癌症和癌症侵袭性的指标,其中muc1*越多,癌症越具有侵袭性。检测muc1*的量或muc1*与全长muc1的比率也可以用于确定治疗药物靶向muc1*或muc1的癌症治疗的适用性。类似地,通过检测治疗前后muc1*的量或muc1*与全长muc1的比率,可以评价这种疗法的有效性,其中表达的muc1*的量
减少或muc1*与全长muc1的比率的变化将是疗效的指标。
[0132]
可能存在不包含ibr或串联重复序列的muc1的可替换剪接亚型,例如,muc1-y或muc1-x。这些可替换剪接亚型仍然具有由psmgfr肽序列组成的胞外结构域,因为这是与生长因子相互作用而促进癌症和生存的部分。因此,检测细胞或组织表达的muc1*的量仍将是癌症和癌症侵袭性的有效指标。
[0133]
乳腺癌组织上的主要muc1种类是跨膜剪切产物muc1*而非全长muc1。乳腺癌微阵列用vu4h5或mnc2探测。vu4h5是单克隆抗体,仅与全长muc1结合,因为其识别全长muc1的串联重复结构域中的表位该表位在全长muc1的串联重复结构域内会重复数百次。因此,抗体vu4h5应该发出抗体结合分子上的单个表位的更强信号。mnc2是我们通过用psmgfr肽(seq id no:4)免疫化动物而产生的单克隆抗体。转染实验表明mnc2不与全长muc1结合。mnc2结合muc1剪切成muc1*形式后暴露的隐性抗原决定基,其至少包含muc1*胞外结构域的前35个膜近端氨基酸,因为其结合psmgfr肽(45个氨基酸)、n-10肽(35个氨基酸)而不结合c-10肽,表明其同源表位至少部分包含于muc1*胞外结构域的10个膜近端氨基酸内。重要的是,mnc2竞争性抑制muc1*激活生长因子nme1和nme7-ab的结合。
[0134]
图1a-1d显示了根据allred评分系统的乳腺癌组织阵列的相邻连续切片的照片和病理学家评分的图形表示。病理学家评分为0-3,其中0表明未染色,3是最大染色。这些图也用颜色编码,其中病理学家评分为零的是黑色,1是黄色,2是橙色,3是红色;用识别全长muc1的抗体探测时评分为零但用识别muc1*的抗体探测时为阳性的组织被着色成绿色。缺失或无法解释的组织评分为-1。图1a显示了乳腺癌组织阵列用vu4h5染色后的照片,vu4h5是结合全长muc1的串联重复结构域的抗体。图1b显示了图1a中所示的组织的病理学家评分的图。图1c显示了乳腺癌组织阵列用mnc2染色后的照片,mnc2是结合muc1*psmgfr区内的表位的抗体。图1d显示了图1c中所示的组织的病理学家评分的图。图2a-2b显示了图1a和图1c所示的阵列的病理学家评分的饼图。图2a表明结合全长muc1的串联重复的抗体错过了30%的乳腺癌。图2b表明抗muc1*抗体mnc2识别95%的乳腺癌。抗muc1全长仅与10%的乳腺肿瘤牢固结合,而抗muc1*抗体mnc2与约50%的乳腺肿瘤牢固结合。这些数据一起表明,muc1*而非全长muc1是癌性组织上的主要muc1种类。抗muc1*抗体检测或诊断几乎所有乳腺癌,而结合全长muc1的抗体无法检测约30%的乳腺癌。此外,由于muc1*是驱动癌症生长的生长因子受体,因此组织或细胞样本的抗muc1*染色的程度将与癌症的程度或阶段成正比,而全长muc1的表达似乎与癌症分期成反比。
[0135]
用抗muc1*抗体mnc2探测广泛的癌细胞和肿瘤样本。mnc2用于多种测试,包括荧光激活细胞分选(facs)、免疫荧光(if)、免疫组织化学(ihc)中检测muc1*阳性癌症。facs和if通常用于研究已经在实验室中繁殖了数十年的永生化细胞的细胞系。在非自然生长溶液中繁殖数十年后,这些细胞系可能甚至表现出与患者原始肿瘤内的单个细胞几乎没有相似之处,并且绝不代表最近被诊断出而寻求治疗的患者的肿瘤。由于这些原因,我们分析了数千个肿瘤微阵列,其中阵列中的每个点是来自单个患者活检的肿瘤样本。在大多数情况下,活检来自最近诊断出的患者,但随附的匿名患者数据给出了患者的年龄、癌症亚型和癌症分期或等级。在某些情况下,我们分析了其中乳腺癌是全部her2+或全部er+/pr+的组织微阵列。在其他情况下,我们分析了将原始活检样本与后来的转移比较的肿瘤微阵列。在这些研
究中,还将mnc2对肿瘤的识别与使用抗全长muc1抗体vu4h5或结合全长muc1串联重复域中的捕获的o连接聚糖的新抗体5e5染色的结果比较。mnc2和其他抗muc1*抗体始终比vu4h5或5e5更好地识别肿瘤组织。正常组织和正常组织微阵列也进行了广泛研究,以确定mnc2或其人源化单链形式humnc2-scfv或humnc2-scfv-fc与正常组织的结合。在正常组织中,mnc2反应性muc1*的表达仅在少数组织中以百分比局限于导管和腺体的顶端边界。在所有情况下,相比于确实表达mnc2反应性muc1*的正常组织0.2%-5%的表达率,mnc2反应性muc1*在癌组织中的表达程度均高于正常组织,并且在癌组织中的表达率超过50%-100%。
[0136]
图3至图19显示单克隆抗muc1*抗体mnc2结合高百分比的乳腺、卵巢、胰腺、肺和食道肿瘤,而与正常组织的任何结合几乎没有。图3a-3b显示了用抗muc1*抗体humnc2-scfv-fc染色后的乳腺癌阵列br1141的病理学家评分饼图和照片。图4a-4c显示了用抗muc1*抗体humnc2-scfv-fc染色后的乳腺癌阵列br1141的各个乳腺癌样本的两个不同放大倍数的照片。图中指示了阵列中的位置、癌症亚型、肿瘤等级,tnm(肿瘤分期,淋巴结转移和远端转移)和病理学家评分。使用了标准的免疫组织化学方法。抗体浓度使用抗体显示正常组织的预期染色而没有染色基质的最高浓度滴定。抗体通过其fc区缀合于生物素,以避免由于抗人二抗染色宿主抗体以及b细胞滤泡而导致假阳性。图4a显示了位置a7上的样品,其对于humnc2反应性细胞为阴性。图4b显示了位置a9上的样本,其是2级癌症,具有淋巴结转移,对于humnc2反应性的评分为+1。图4c显示了b10位置上的样本,该样本是更大的2级肿瘤,具有淋巴结转移,对于humnc2反应性的评分为+2。图5a-5b显示了用抗muc1*抗体humnc2-scfv-fc染色后的来自乳腺癌阵列br1141的单个乳腺癌样本的两个不同放大倍数的照片。图5a显示了位置d7上的样品,其是2级癌症,没有淋巴结转移,humnc2反应性评分为+3。图5b显示了位置f6上的样本,其是2级肿瘤,具有淋巴结转移,humnc2反应性的评分为+4。图6a-6b显示了用抗muc1*抗体humnc2-scfv-fc染色后的卵巢癌阵列bc1115a的病理学家评分饼状图和照片。图7a-7c显示了用抗muc1*抗体humnc2-scfv-fc染色后的不同癌症亚型的放大照片。图7a显示了病理学家评分为+4的2级乳腺肿瘤的照片。图7b显示了病理学家评分为+3的2级卵巢肿瘤的照片。图7c显示了病理学家评分为+3的3级胰腺肿瘤的照片。对超过1,000个肿瘤样本的ihc研究表明,humnc2-scfv识别95%的乳腺癌(90%三阴性)、83%的卵巢癌、78%的胰腺癌和71%的肺癌。图8a-8d显示了用抗muc1*抗体humnc2-scfv-fc染色后的不同癌症亚型的放大照片。图8a显示了病理学家评分为+2的2级乳腺肿瘤的照片。图8b显示了病理学家评分为+3的3级卵巢肿瘤的照片。图8c显示了3级胰腺肿瘤的照片,其中淋巴结转移病理学家评分为+3。图8d显示了病理学家评分为+3的肺癌照片。图9a-9i显示了用抗muc1*抗体humnc2-scfv-fc染色后的各种正常组织的放大照片。使用的条件和浓度与用于研究癌组织的条件和浓度相同。图9a显示了正常的肾上腺组织。图9b显示了正常的脑组织。图9c显示了正常的乳腺组织。图9d显示了正常的胃组织。图9e显示了正常的心脏组织。图9f显示了正常的肾脏组织。图9g显示了正常的睾丸组织。图9h显示了正常的肠组织。图9i显示了正常肝组织。图10a-10f显示了用抗muc1*抗体humnc2-scfv-fc染色后的正常肾脏组织的照片。使用的条件和浓度与用于研究癌组织的条件和浓度相同。图10a显示了具有限制于顶端边界的humnc2反应性的正常肾脏组织,其为正常表达。图10b是更大放大后的相同组织。图10c显示了具有不可检测的humnc2反应性的正常肾脏组织的另一个实例。图10d是更大放大后的相同组织。图10e显示了具有限制于顶端边界的humnc2反应性的正常肾脏组织的另一个实例,
其表达正常。图10f是更大放大后的相同组织。进一步的研究表明,小于10%的正常肾脏组织在远端收集小管中显示出humnc2反应性,其中这种反应性严格限于顶端边界,这是正常表达模式。图11a-11b显示了用抗muc1*抗体humnc2-scfv-fc染色后的食道癌阵列bc001113的病理学家评分饼图和照片。图12a-12f显示了用抗muc1*抗体humnc2-scfv-fc染色之后的来自食道癌阵列bc001113的单个食道癌样本在两个不同的放大倍数下的照片。图中指示了阵列中的位置、癌症亚型、肿瘤等级和病理学家评分。图12a显示了位置a4上的样品,其对于humnc2反应性细胞呈阴性。图12b显示了更大放大倍数的相同样本。图12c显示了位置d2上的样本,病理学家对其评分为对于humnc2反应性微弱。图12d显示了更大放大倍数的相同样本。图12e显示了位置b8上的样本,病理学家对其评分为humnc2反应性+1。图12f显示了更大放大倍数的相同样本。图13a-13d显示了用抗muc1*抗体humnc2-scfv-fc染色之后来自食道癌阵列bc001113的各个食道癌样本的两个不同放大倍数的照片。图中指示了阵列中的位置、癌症亚型、肿瘤等级和病理学家评分。图13a显示了位置d6上的样本,其是4级肿瘤,病理学家评分为+2。图13b显示了更大放大倍数的相同样本。图13c显示了位置d5上的样本,其是3级肿瘤,病理学家评分为+3。图13d显示了更大放大倍数的相同样本。图14a-14b显示了用抗muc1*抗体humnc2-scfv-fc染色后的胰腺癌阵列pa805b的病理学家评分饼图和照片。图15a-15d显示了用抗muc1*抗体humnc2-scfv-fc染色后来自胰腺癌阵列pa805b的各个胰腺癌样本的两个不同放大倍数的照片。图中指示了阵列中的位置、癌症亚型、肿瘤等级和病理学家评分。图15a显示了位置f3上的样本,其是3级肿瘤,病理学家评分为+3。图15b显示了更大放大倍数的相同样本。图15c显示了b1位置上的样本,其是1级肿瘤,病理学家评分为+2。图15d显示了更大放大倍数的相同样本。图16a-16d显示了用抗muc1*抗体humnc2-scfv-fc染色后来自胰腺癌阵列pa805b的各个胰腺癌样本的两个不同放大倍数照片。图中指示了阵列中的位置、癌症亚型、肿瘤等级和病理学家评分。图16a显示了位置a2上的样本,其是1级肿瘤,病理学家评分为+2。图16b显示了更大放大倍数的相同样本。图16c显示了位置c3上的样本,其是2级肿瘤,病理学家评分为+2。图16d显示了更大放大倍数的相同样本。图17a-17d显示了用抗muc1*抗体humnc2-scfv-fc染色后来自胰腺癌阵列pa805b的各个胰腺癌样本的两个不同放大倍数的照片。图中指示了阵列中的位置、癌症亚型、肿瘤等级和病理学家评分。图17a显示了位置c6上的样品,其是2级肿瘤,病理学家评分为+2。图17b显示了更大放大倍数的相同样本。图17c显示了d1位置上的样本,其是更大的3级肿瘤,具有淋巴结转移,病理学家评分为+3。图17d显示了更大放大倍数的相同样本。图18a-18d显示了用抗muc1*抗体humnc2-scfv-fc染色后来自胰腺癌阵列pa805b的单个胰腺癌样本的两个不同放大倍数的照片。图中指示了阵列中的位置、癌症亚型、肿瘤等级和病理学家评分。图18a显示了位置e2上的样本,其是1级肿瘤,病理学家评分为+2。图18b显示了更大放大倍数的相同样本。图18c显示了e10位置上的样本,其是更小的3级肿瘤,具有淋巴结转移,病理学家评分为+3。图18d显示了更大放大倍数的相同样本。图19显示了作为对照的单用二抗染色的胰腺癌阵列pa805b的照片。
[0137]
尽管mnc2识别所有乳腺癌亚型中约95%的乳腺肿瘤,但我们注意到某些癌症亚型表达的mnc2反应性muc1*不如乳腺癌。具体地,胰腺癌、食道癌和前列腺癌的mnc2反应性muc1*水平较低。胰腺癌阵列显示78%的肿瘤具有mnc2反应性,但与肿瘤表达水平成正比的染色强度相对较弱。图14a的饼图表明65%的胰腺肿瘤评分为+1或+2,只有5%的胰腺肿瘤
评分为+3,而评分为+4则没有。图3a的饼形图表明,超过一半的乳腺肿瘤评分为+2至+3,6%的评分为+4,而只有4%的mnc2 muc1*反应性为阴性。两种阵列均使用相同的mnc2抗muc1*抗体染色,并由相同的专业认证的病理学家评分。我们认为,乳腺癌和胰腺癌中muc1*的mnc2染色之间的差异可能是由于在不同位置剪切muc1为muc1*的剪切酶的差异所致,这种酶差异会导致muc1*胞外结构域发生构象或线性变化。为了研究,我们用抗muc1*多克隆抗体sdix对相同胰腺癌阵列染色。尽管mnc2和sdix都是通过用psmgfr肽免疫动物产生的,但它们显示出结合肿瘤组织的不同特性。通常,sdix比mnc2识别更多的胰腺组织并且染色更牢固,但在某些情况下mnc2可以识别sdix不能识别的肿瘤。
[0138]
在癌组织上,muc1*在大多数组织上表达并且有癌的特性,癌组织中的所有解剖学障碍均被破坏。相反,在正常组织上,muc1*的表达仅限于导管和腺体的顶端边界。mnc2反应性muc1*的表达甚至受到进一步限制。例如,图6b显示了卵巢癌微阵列的照片。然而,j列由正常的卵巢组织构成。这可以看出,没有mnc2反应性muc1*的表达。正常肾脏确实表达一些mnc2反应性muc1*。由图10a-10f中可以看出,正常的muc1*表达较弱,并且局限于约10%的正常肾脏远端收集小管的顶端边界。正常胰腺表达的muc1*再次严格限制于腺泡细胞的顶缘(图20)。本领域技术人员可以很容易地识别癌组织,并可以区分正常组织和癌组织上的muc1*表达。通常而言,muc1*在癌组织上完全过表达,其表达不限于顶端表达模式。
[0139]
在图20至图34中,我们证实一系列胰腺肿瘤未显示单克隆抗体mnc2的染色或仅有最低染色,但用sdix多克隆抗体染色的同一组织产生了牢固染色。mnc2和sdix是通过用相同的肽psmgfr免疫动物产生的。然而,mnc2仅识别sdix识别的那些的子集。这些结果强有力地证明,mnc2识别仅在肿瘤子集中产生的表位。数据表明,muc1*的mnc2反应性子集可能是癌症亚型特异性的或患者特异性的,这可能是由于不同剪切酶的剪切所致。
[0140]
图35至图37所示的数据支持抗muc1*抗体特异性依赖于muc1剪切为muc1*的剪切酶的假说。mnc2、mnc3和sdix全部通过用psmgfr肽对动物免疫而产生。然而,单克隆抗体mnc3和多克隆抗体sdix一样识别近100%的造血干细胞,而单克隆抗体mnc2则不能。相反,mnc2结合近95%的乳腺肿瘤,但mnc3则不能。重要的是,我们证实,mnc2可以识别在剪切酶mmp9剪切muc1后的muc1*,该酶在大多数乳腺癌中均过表达,但在造血干细胞中却不表达。mmp9的表达可以预测大多数实体瘤癌症的不良预后(yousef et al.,bmc cancer 2014,14:609;mehner et al.,oncotarget,vol.5,no.9,pp 2736-2749,2014;radisky et al.,front biosci(landmark ed).;20:1144-1163,2015;gong et al.,journal of surgical oncology 2000;73:95-99;latinovic et al.,arch oncol 2013;21(3-4):109-14;sillanpaa et al.,gynecologic oncology 104(2007)296-303)。
[0141]
为了产生能够识别宽范围的muc1*的新抗muc1*单克隆抗体,其可以是癌症亚型特异性的、患者特异性的或为了更好解决肿瘤异质性,我们用衍生自muc1*胞外结构域序列的以下肽对动物进行免疫:
[0142]
(i)psmgfr肽
[0143][0144]
(ii)psmgfr n+20/c-27
[0145]
或
[0146]
(iii)psmgfr n+9/c-9
[0147][0148]
分离出抗体克隆,并首先基于它们结合免疫肽的能力,其次基于它们识别正常组织之上的癌组织的能力,从每次免疫中选择出子集。图38a至图38c显示了根据免疫肽安排的选择的抗体的表格。在表中,-1或-2的标识表示它们是姐妹克隆,测序后显示它们实际上是相同的抗体。在本公开的其余部分中,提及的抗体没有-1或-2标识。
[0149]
图39至图44显示了新抗muc1*抗体的结合特性。所有抗体首先根据它们与免疫肽结合的事实选择。为了与mnc2和mnc3比较,测试了新抗体结合psmgfr、n-10肽和c-10肽的能力。通过facs还测试了新抗muc1*抗体,以确定它们与t47d乳腺癌细胞系结合的能力。由于数十年前从患者体内产生的抗体与单个细胞系的结合分析,我们将新抗体的分析扩展到了多种癌症亚型的数百个肿瘤组织。每个阵列中代表的患者数量各不相同。正常组织也用这些抗体进行探测。
[0150]
图45至图52比较了新抗muc1*抗体与sdix多克隆的结合,以研究与psmgfr序列的n端区结合的抗体。我们从胰腺癌阵列开始,因为我们先前的研究表明,尽管mnc2识别约78%的胰腺癌,但结合较不牢固,而mnc2或sdix多克隆抗体完全无法识别某些非常疑难的肿瘤。
[0151]
一些抗psmgfr抗体,如18b4,似乎会识别与多克隆抗psmgfr抗体sdix相同的胰腺肿瘤组织(图45a-45bc)。在这种小型胰腺癌阵列中,抗psmgfr n+20/c-27抗体1e4似乎会识别与sdix和18b4相同的肿瘤,然而,这些肿瘤样本的放大图表明,抗体1e4与抗psmgfr抗体相比会识别肿瘤中的不同癌细胞群体(图46a-46f),一些肿瘤不能被sdix很好地识别,但可以被单克隆抗体18b4识别(图47a-48d)。抗psmgfr n+20/c-27抗体1e4可以更好地识别其他胰腺肿瘤(图49a-49d)。类似地,抗psmgfr n+20/c-27抗体29h1会识别抗psmgfr抗体sdix和20a10错过的一些胰腺肿瘤(图51a-51c)。
[0152]
这些研究表明,通常结合在n端延伸超出psmgfr的muc1*胞外结构域的抗体识别胰腺癌比sdix多克隆抗体更好。然而,胰腺肿瘤的抗体特异性似乎也是患者特异性的。某些患者样本用sdix抗psmgfr抗体染色比结合psmgfr n+20/c-27或psmgfr n+9/c-9的新抗体好得多。这支持以下想法:患者肿瘤必须用一组muc1*抗体探测以确定最适合消除其肿瘤的治疗方法。在本发明的一方面中,治疗剂引入作为诊断剂的一些或全部抗体,或引入衍生于作为诊断抗体的抗体的一些或全部抗体。
[0153]
图53显示,与结合全长muc1的抗体相比,这些在n端延伸的新抗体识别更多的胰腺肿瘤。该图将29h1与结合全长muc1串联重复的标准抗体vu4h5的结合与结合一些癌细胞上的捕获的o链聚糖的新抗体5e5比较。
[0154]
我们接着研究了食道肿瘤和前列腺肿瘤。这些研究是由我们先前的发现,即与psmgfr肽都结合的单克隆抗体mnc2以及多克隆抗体sdix也显示出对食道肿瘤和前列腺肿瘤的较差识别启发的。实际上,在肿瘤样本的良好区分的部分中表现出某些mnc2反应性的那些肿瘤,在同一样本的较差区分的部分中丧失了这种反应性。这些结果表明,在大多数食道癌和前列腺癌中,除mmp9以外的其他剪切酶占主导地位。这些研究支持了这一想法。
[0155]
结合肽psmgfr n+20/c-27和/或psmgfr n+9/c-9的新抗muc1*抗体,相比于mnc2、sdix和全长muc1抗体5e5和vu4h5时,对食道和前列腺肿瘤表现出更好的识别能力。
[0156]
图54a-54c显示了通过标准ihc方法用各种抗muc1*抗体染色的来自食道癌阵列的相邻连续切片的照片。图54a显示了用sdix多克隆抗muc1*抗体染色的阵列,其中抗体的免
疫原是psmgfr肽。图54b显示了用20a10单克隆抗muc1*抗体染色的阵列,其中该抗体的免疫原是psmgfr肽。图54c显示了用29h1单克隆抗muc1*抗体染色的阵列,其中该抗体的免疫原是psmgfr n+20/c-27肽。图54d显示了用31a1单克隆抗muc1*抗体染色的阵列,其中该抗体的免疫原是psmgfr n+20/c-27肽。该图表明,尽管结合程度不同,但与psmgfr肽结合的sdix和20a10抗体可以识别相同的肿瘤组织样本,而结合psmgfr n+20/c-27肽的抗体可以结合更多的食道肿瘤样本以及大多数被抗psmgfr抗体识别的那些。这些结果与以下观点相吻合:结合psmgfr n+20/c-27肽的抗体通常比结合psmgfr肽的抗体对食道癌更具特异性,但某些患者可能患有被结合psmgfr肽的抗muc1*抗体更好识别的食道癌。
[0157]
图55a-55c显示了通过标准ihc方法用各种抗muc1*抗体染色的来自食道癌阵列的相邻连续切片的照片。图55a显示了用sdix多克隆抗muc1*抗体染色的阵列,其中该抗体的免疫原是psmgfr肽。图55b显示了用17h6单克隆抗muc1*抗体染色的阵列,其中该抗体结合psmgfr n+9/c-9肽。图55c显示了用mnc2单克隆抗muc1*抗体染色的阵列,其中该抗体结合psmgfr肽。图55d显示了用45c11单克隆抗muc1*抗体染色的阵列,其中该抗体结合psmgfr n+20/c-27肽。这些结果与以下想法相符:在大多数食道癌上,muc1被暴露psmgfr序列n端的隐蔽表位的酶剪切。
[0158]
图56a-56f显示了通过标准ihc方法用识别全长muc1的抗体或仅识别muc1*的抗体染色的来自食道癌阵列的相邻连续切片的照片和病理学家染色分数的图形表示。图56a显示了用抗体5e5染色的食道癌阵列,该抗体是结合全长muc1的串联重复结构域中的捕获的o连接聚糖的抗体。图56b显示了阵列中每个样品的病理学家评分。图56c显示了用抗muc1*抗体29h1染色的食道癌阵列,该抗体是结合muc1*的psmgfr n+20/c-27肽的抗体。图56d显示了阵列中每个样本的病理学家评分。图56e显示了用抗体vu4h5染色的食道癌阵列,该抗体是结合全长muc1的串联重复结构域中的表位的抗体。图56f显示了阵列中每个样本的病理学家评分。如该图所示,抗体5e5识别vu4h5不识别的一些样本,然而,抗muc1*抗体29h1识别被识别全长muc1的两种抗体识别的样本以及未被任一种抗muc1抗体识别的其它样本。这些发现表明,结合包含n端超出psmgfr序列的氨基酸的肽的抗muc1*抗体不能识别全长muc1,而结合psmgfr n+20/c-27肽的抗体可以识别食道癌上普遍存在的表位。
[0159]
图57a-57g显示了用均识别全长muc1的抗体5e5或vu4h5或仅识别muc1*并结合psmgfr n+20/c-27肽的29h1染色的前列腺癌阵列的照片。图57a显示了用抗体5e5染色的食道癌阵列。图57b显示了用抗体29h1染色的食道癌阵列。图57b显示了用抗体29h1染色的食道癌阵列。图57c显示了用抗体vu4h5染色的食道癌阵列。图57d显示了作为对照的仅用二抗染色的食道癌阵列。图57e显示了更大放大倍数的图57a中用红色框标记的组织,其中染色用5e5进行。图57f显示了更大放大倍数的图57b中用红色框标记的组织,其中染色用29h1进行。图57g显示了更大放大倍数的图57c中用红色框标记的组织,其中染色用vu4h5进行。红色虚线框表示许多食道肿瘤样本中对识别全长muc1的抗体染色为阴性而在用抗muc1*抗体,尤其结合psmgfr n+20/c-27肽的那些抗体探测时高度阳性的唯一患者样本。
[0160]
图58a-58c显示了通过标准ihc方法用各种抗muc1*抗体染色的前列腺癌阵列的相邻连续切片的照片。图58a显示了用sdix多克隆抗muc1*抗体染色的阵列,其中抗体的免疫原是psmgfr肽。图58b显示了用18b4单克隆抗muc1*抗体染色的阵列,其中该抗体结合psmgfr肽。图58c显示了用1e4单克隆抗muc1*抗体染色的阵列,其中该抗体结合psmgfr n+
20/c-27肽。
[0161]
图59a-59e显示了通过标准的ihc方法用各种抗muc1*抗体染色的来自前列腺癌阵列的相邻系列切片的照片。图59a显示了用结合psmgfr肽而不结合c-10肽的mnc2单克隆抗体染色的阵列。图59b显示了用结合psmgfr肽的18b4抗体染色的阵列。图59c显示了用结合psmgfr n+20/c-27肽的32c1抗体染色的阵列。图59d显示了用sdix多克隆抗muc1*抗体染色的阵列,其中该抗体的免疫原是psmgfr肽。图59e显示了用结合psmgfr n+20/c-27肽的31a1单克隆抗muc1*抗体染色的阵列。
[0162]
图60a-60f显示了通过标准ihc方法用识别全长muc1的抗体或仅识别muc1*的抗体染色的来自前列腺癌阵列的相邻连续切片的照片和病理学家染色评分的图形表示。图60a显示了用抗体5e5染色的前列腺癌阵列,该抗体是结合全长muc1的串联重复结构域中的捕获的o连接聚糖的抗体。图60b显示了阵列中每个样品的病理学家评分。图60c显示了用抗muc1*抗体29h1染色的前列腺癌阵列,该抗体是结合muc1*的psmgfr n+20/c-27肽的抗体。图60d显示了阵列中每个样本的病理学家评分。图60e显示了用抗体vu4h5染色的前列腺癌阵列,该抗体是结合全长muc1的串联重复结构域中的表位的抗体。图60f显示了阵列中每个样本的病理学家评分。如该图所示,抗体5e5可以识别vu4h5不识别的一些样本,然而,抗muc1*抗体29h1识别被两种识别全长muc1的抗体识别的样本以及未被任一种抗muc1抗体识别的样本。这些发现表明,与包含n端延伸超出psmgfr序列的氨基酸的肽结合的抗muc1*抗体不能识别全长muc1,而与psmgfr n+20/c-27结合的抗体肽识别前列腺癌上普遍存在的表位。
[0163]
图61a-61g显示了用均识别全长muc1的抗体5e5或vu4h5或仅识别muc1*并结合psmgfr n+20/c-27肽的29h1染色的前列腺癌阵列的照片。图61a显示了用抗体5e5染色的前列腺癌阵列。图61b显示了用抗体29h1染色的前列腺癌阵列。图61b显示了用抗体29h1染色的前列腺癌阵列。图61c显示了用抗体vu4h5染色的前列腺癌阵列。图61d显示了作为对照的仅用二抗体染色的前列腺癌阵列。图61e显示了更大放大倍数的图61a中用红色框标记的组织,其中染色用5e5进行。图61f显示了更大放大倍数的图61b中用红色框标记的组织,其中染色用29h1进行。图61g显示了更大放大倍数的图61c中用红色框标记的组织,其中染色用vu4h5进行。红色虚线框表示许多前列腺肿瘤样本中对识别全长muc1的抗体染色为阴性而在用抗muc1*抗体,尤其是结合psmgfr n+20/c-27肽的抗体探测时为高度阳性的唯一患者样本。
[0164]
mnc2识别大百分比的乳腺癌中存在的muc1*。然而,通过其中生长因子受体muc1*被不同的剪切酶剪切而由此被不同的抗muc1*抗体识别的细胞群增殖的肿瘤异质性和肿瘤逃逸的潜力表明,用多于一种抗muc1*抗体进行治疗将是有益的。为此,我们更紧密地比较了新抗muc1*抗体对mnc2的识别(图62-图73)。
[0165]
用均结合psmgfr肽、n-10肽而不结合c-10肽的mnc2或20a10对乳腺癌阵列br1141染色。为了一级近似,两种抗体识别乳腺癌中表达的muc1*的相同或非常接近的表位(图62a-62b)。图63a-65b显示了与25e6、18b4和18g12相比相同的乳腺癌阵列。回想到与mnc2不同,该组新抗psmgfr抗体可以结合c-10肽(图41)。如图中可见,mnc2的结合与这些新的抗psmgfr抗体的结合之间存在差异。当将mnc2与抗psmgfr n+9/c-9抗体8a9(图66a-66b)和抗psmgfr抗体28f9(图67a-67b)比较时,对患者之间的乳腺癌群体以及同一肿瘤内的识别差
异更加明显。参考图41,抗体28f9显示出与c-10肽的最高结合程度,而mnc2不结合c-10肽,这是因为这些抗体结合muc1*的截短的胞外结构域上非常不同的表位。在图69a-69b中清楚可见抗psmgfr n+9/c-9抗体3c5和mnc2的结合之间的差异。抗psmgfr抗体20a10和18b4与其它结合肽psmgfr n+20/c-27的抗体如29h1、45c11和32c1、31a1或结合psmgfr n+9/c-9肽的抗体如17h6之间在乳腺癌识别上的差异显示于图70a-70g中。
[0166]
用抗muc1*抗体29h1探测更小的乳腺癌阵列br1007,并与用抗全长muc1抗体5e5和vu4h5探测时的相同阵列的识别比较(图71a-71f)。如图所见,抗体5e5识别vu4h5不识别的一些样本,然而,抗muc1*抗体29h1识别被两种识别全长muc1的抗体识别的样本以及未被任一种抗muc1抗体识别的样本。这些发现表明,与包括n端延伸超出psmgfr序列的氨基酸的肽结合的抗muc1*抗体不能识别全长muc1。
[0167]
在图72a-72f中,mnc2与乳腺癌阵列br1141的结合与一组抗psmgfr抗体进行了比较。所有这些抗体均结合psmgfr肽,并大致产生与该乳腺癌阵列相同的染色模式。然而,这些抗体在阵列中识别各个样本的方式存在一些差异,这可能代表不同酶将muc1剪切为muc1*。参照图39,mnc2和20a10结合n-10肽而不结合c-10肽,表明10个膜近端氨基酸对于它们的结合是重要的。抗体18b4、18g12和25e6表现出与c-10肽一定程度的结合,而28f9表现出与c-10肽的更多结合。值得注意的是,18b4不与n-10肽结合,表明其相比其他与psmgfr中更加n端的表位结合。除了前面提到的例外,抗psmgfr抗体在该阵列内对肿瘤的识别非常相似。
[0168]
相反,结合psmgfr n+9/c-9肽的抗体牢固地识别未被mnc2识别或未被mnc2和其他抗psmgfr抗体微弱识别的肿瘤子集(图73a-73f)。所示的照片是已用各种抗muc1*单克隆抗体染色的乳腺癌组织阵列br1141的相邻连续切片的照片,其中结合psmgfr n+9/c-9肽的抗体与mnc2及其人源化单链形式humnc2-scfv-fc比较,这两者均结合psmgfr n-10,但不结合c-10肽。图73a显示了用mnc2染色的乳腺癌样本。图73b显示了用8a9染色的乳腺癌样本。图73c显示了用17h6染色的乳腺癌样本。图73d显示了用humnc2-scfv-fc染色的乳腺癌样本。图73e显示了用3c5染色的乳腺癌样本。图73f显示了用39h5染色的乳腺癌样本。现在参考通过红色圆圈标记的患者样本,容易看出结合psmgfr n+9/c-9肽的抗体识别mnc2抗psmgfr抗体错过或微弱结合的乳腺癌细胞群体。抗muc1*抗体8a9、17h6、3c5和39h5识别不被抗psmgfr抗体如mnc2、20a10、25e6、28f9、18g12或18b4识别或识别程度较低的癌细胞的独特子集。
[0169]
总体而言,这些数据表明:(i)当用抗muc1*抗体而不是结合全长muc1的抗体探测肿瘤时,即使在癌症亚型如乳腺癌等中,muc1阳性癌症的诊断也更准确;(ii)当用一种以上的抗muc1*探测肿瘤时,即使在癌症亚型如乳腺癌等中,muc1阳性癌症的诊断也更加准确;(iii)当用一种以上的抗muc1*探测肿瘤时,即使在癌症亚型如乳腺癌等中,muc1阳性癌症的诊断更加准确,其中至少两种不同的抗体选自两个不同的组中,其中该组是结合psmgfr肽的抗体、结合psmgfr n+20/c-27肽的抗体和结合psmgfr n+9/c-9肽的抗体。
[0170]
本发明可以用于诊断癌症的抗muc1*抗体包括结合psmgfr肽、psmgfr n+20/c-27肽、psmgfr n+9/c-9肽,或更具体地结合具有以下序列的至少15个连续氨基酸(至多具有四个氨基酸取代)的肽的抗体:
[0171]
(i)muc1的psmgfr区;
[0172]
(ii)seq id no:4所示的psmgfr肽;
[0173]
(iii)psmgfr n+20/c-22,具有的氨基酸序列的肽;
[0174]
(iv)psmgfr n+12/c-22,具有的氨基酸序列的肽;
[0175]
(v)psmgfr n+9/c-30,具有的氨基酸序列的肽;
[0176]
(vi)psmgfr n+20/c-41,具有的氨基酸序列的肽;
[0177]
(vii)psmgfr n+20/c-27,具有的氨基酸序列的肽;或
[0178]
(viii)psmgfr n+9/c-9,具有具有的氨基酸序列的肽。具体地,抗psmgfr抗体mnc2、mne6、18b4、18g12、20a10、25e6、抗psmgfr n+20/c-27抗体1e4、29h1、31a1、32c1、45c11和抗psmgfr n+9/c-9抗体3c5、8a9、17h6和39h5是可以用于诊断癌症的抗体。这些抗体可以是人的、人源化的或非人的。它们可以是完整的抗体或抗体片段。通过用上述序列(i)-(viii)的肽免疫动物可以产生抗体。用muc1*胞外结构域肽免疫而产生抗体的动物可以是人、兔、小鼠、山羊、驴、骆驼科、美洲驼、羊驼或其他非人物种。
[0179]
本发明的抗体可以用于诊断测试中,其中其可以源自显像剂、染料、荧光实体、生色试剂或使抗体可以光学、视觉、电学或放射性检测的任何其他实体或与其连接。本发明的抗体可以以多种诊断形式使用。
[0180]
在另一个实施例中,本发明的抗muc1*抗体可以附连至成像剂,用于活体患者作为全身诊断以确定患者是否患有muc1*阳性肿瘤或确定患者是否将受益于包含抗muc1*抗体的全部或片段的治疗剂,该抗muc1*抗体可以源自诊断中使用的抗体或具有与其相似的结合特性。诊断抗体和治疗性抗体的种类不必相同。在骆驼科中产生的抗体对于体内诊断分析测试特别有用,因为骆驼科会产生在人体内半衰期很短的小单价抗体。
[0181]
在又一个实施例中,本发明的抗muc1*抗体可以附连至成像剂,并且在手术中用于检测或标记癌组织,因此它们可以在手术期间完全切除。
[0182]
在本发明的一个方面中,使来自被诊断患有癌症或怀疑有患癌风险的患者的体液或组织样本与一种或多种本发明的抗muc1*抗体接触;对抗体与样品细胞的结合的分析表明指示癌症的结合水平或结合模式。然后将用于癌症治疗的治疗剂给予患者。在本发明的一方面中,治疗剂包含抗muc1*抗体的全部或片段。
[0183]
在一个实施例中,使用抗muc1*抗体或其片段的诊断测试用于筛选患者以确定他
们从靶向muc1*的治疗中的潜在受益。诊断中使用的抗muc1*抗体和引入治疗剂中的抗体或其片段可以源自同一抗体。诊断抗体和治疗抗体的种类不必相同。诊断测试可以设计一种或多种抗muc1*抗体的使用。与一种或多种抗muc1*抗体反应的患者样本指示该患者可能会受益于含有一种或多种反应性抗体或其片段的治疗药物的给予。
[0184]
一个实施例包括以下步骤:(i)使来自被诊断患有癌症或怀疑患有癌症的患者的可疑细胞或组织样本与抗muc1*抗体接触,该样本可以是活检;(ii)使来自患者或健康供体的正常细胞或组织样本与相同的抗muc1*抗体接触,其可以是存档的参考样本;(iii)检测抗体结合;(iv)将抗体与可疑样本结合的程度和模式与正常样本比较;(v)确定可疑样本过表达muc1*或与局限于顶端边界的表达相反,以统一的模式表达muc1*,指示该患者患有muc1*阳性癌症;(vi)然后向患者给予用于癌症治疗的治疗剂,该治疗剂可以是引入抗muc1*抗体或其片段的治疗剂。
[0185]
在本发明的一个方面中,来自诊断或疑似癌症患者的体液或组织样本与本发明的抗muc1*抗体接触,并且检测高于正常水平的muc1*或检测muc1*异常模式,表明该患者患有muc1*阳性癌症,然后向该患者给予其中引入抗muc1*抗体或抗体片段的治疗药物。在一种情况下,引入抗体或抗体片段的治疗药物是免疫学肿瘤药剂,如car t细胞、工程化nk细胞或树突细胞。在另一种情况下,引入抗体或抗体片段的治疗剂是humnc2-car44 t细胞。在本发明的又一方面中,其中引入抗体或抗体片段的治疗剂是双特异性抗体。在本发明的又一方面中,其中引入抗体或抗体片段的治疗剂是抗体药物偶联物(adc)。在本发明的又一方面中,其中引入抗体或抗体片段的治疗剂是双特异性t细胞衔接剂(bite)。
[0186]
在另一个实施例中,诊断测试可以包含抗muc1*抗体和第二抗体,并且步骤可以包括确定第一抗体与第二抗体的量的比率。第一抗体可以结合muc1*胞外结构域,而第二抗体可以结合剪切位点n端的muc1胞外结构域的部分,如串联重复序列。在接触组织样本的情况下,muc1*与全长muc1的比率越高,癌症进展越多,而患者越有可能受益于靶向muc1*的治疗药物。
[0187]
本发明包括抗体以及抗体样蛋白,包括但不限于,多克隆,单克隆,嵌合体,人源化,单链,抗体片段等。另外,本发明包括蛋白质支架用于产生抗体模拟物而获得可以通过本文的结合测试表征的蛋白质的用途。本发明还包括使用本文的方法鉴定在muc1*胞外结构域内识别在癌细胞上差异表达的特异性表位的抗体。
[0188]
在一方面中,本发明涉及结合缺乏串联重复结构域的muc1亚型或剪切产物的胞外结构域上的区域的人的或人源化的抗muc1*抗体或抗体片段或抗体样蛋白。人的或人源化的抗muc1*抗体或抗体片段或抗体样蛋白可以特异性地结合
[0189]
(i)muc1的psmgfr区;
[0190]
(ii)seq id no:4所示的psmgfr肽;
[0191]
(iii)psmgfr n+20/c-22,具有的氨基酸序列的肽;
[0192]
(iv)psmgfr n+12/c-22,具有的氨基酸序列的肽;
[0193]
(v)psmgfr n+9/c-30,具有的氨基酸序列的肽;
[0194]
(vi)psmgfr n+20/c-41,具有的氨基酸序列的肽;
[0195]
(vii)psmgfr n+20/c-27,具有的氨基酸序列的肽;或
[0196]
(viii)psmgfr n+9/c-9,具有的氨基酸序列的肽。
[0197]
人的或人源化的抗体可以是igg1、igg2、igg3、igg4或igm。人的或人源化的抗体片段或抗体样蛋白可以是scfv或scfv-fc。
[0198]
根据以上的人或人源化的抗体、抗体片段或抗体样蛋白可以包含重链可变区和轻链可变区,其源自小鼠单克隆mn-e6抗体,并具有与小鼠单克隆mn-e6抗体至少80%、90%或95%或98%的序列一致性。
[0199]
根据上述的人或人源化的抗体、抗体片段或抗体样蛋白可以包括与具有psmgfr n+20/c-27反应性的抗体1e4、29h1、31a1、32c1和45c11;具有psmgfr n+9/c-9反应性的17h6、39h5、3c5、8a9;具有psmgfr反应性的18g12、20a10、25e6、28f9、18b4,mnc2和mne6的cdr1,cdr2或cdr3区具有至少90%或95%或98%序列一致性的重链可变区和轻链可变区中的互补决定区(cdr)。
[0200]
在另一方面中,本发明涉及根据上文抑制nme蛋白与muc1*的结合的人或人源化的抗muc1*抗体或抗体片段或抗体样蛋白。nme可以是nme1、nme6、nme7ab、nme7或nme8。
[0201]
在又一方面中,本发明涉及包含结合不含串联重复的muc1的胞外结构域、接头分子、跨膜结构域和胞质结构域的scfv或人源化可变区的嵌合抗原受体(car)。单链抗体片段可以结合
[0202]
(i)muc1的psmgfr区;
[0203]
(ii)seq id no:4所示的psmgfr肽;
[0204]
(iii)psmgfr n+20/c-22,具有的氨基酸序列的肽;
[0205]
(iv)psmgfr n+12/c-22,具有的氨基酸序列的肽;
[0206]
(v)psmgfr n+9/c-30,具有的氨基酸序列的肽;
[0207]
(vi)psmgfr n+20/c-41,具有的氨基酸序列的肽;
[0208]
(vii)psmgfr n+20/c-27,具有的氨基酸序列的肽;或
[0209]
(viii)psmgfr n+9/c-9,具有的氨基酸序列的肽;
[0210]
在这方面中,优选的实施方式是seq id no:236中所示的humnc2-car44。
[0211]
在一个方面中,本发明涉及一种用于治疗被诊断、怀疑患有muc1或muc1*阳性癌症或具有发展为muc1或muc1*阳性癌症的风险的人的方法,该方法涉及向该人给予有效量的癌症特异性抗体如mnc2或mne6,或其片段,其中该抗体可以是人的、人源化的或非人物种的。在本发明的一个具体方面中,muc1*靶向治疗剂是用也称为car t的嵌合抗原受体转导的免疫细胞,其中car的抗体片段衍生自muc1*癌细胞特异性抗体。在一方面中,其源自mnc2。在另一种情况下,其源自mne6。
[0212]
在另一方面中,本发明涉及一种用于鉴定可能受益于用包括选自1e4、29h1、31a1、32c1、45c11、17h6、39h5、3c5、8a9、18g12、20a10、25e6、28f9、18b4,mnc2和mne6抗体的组的抗体或其片段的治疗剂治疗muc1或muc1*阳性癌症的人的诊断测试。在本发明的一方面中,抗muc1*抗体或其片段包含全部或部分治疗剂,并且可以衍生自用于诊断的抗体或其片段,其中治疗剂和诊断剂不必是相同的物种。在另一个实例中,包含全部或部分治疗剂的抗muc1*抗体或其片段不是衍生自用于诊断的抗体或其片段,其中治疗剂和诊断剂不必是同一物种。
[0213]
在本发明的一方面中,治疗剂靶向muc1*。在本发明的另一方面中,包含一些或全部抗muc1*抗体的治疗剂是癌症免疫疗法组合物、car t、bite、抗体或抗体药物偶联物adc。
[0214]
在本发明的一个方面中,诊断剂是用于确定治疗剂的治疗资格的伴随诊断剂。在本发明的另一方面中,诊断剂用于评价疗法治疗的功效。在本发明的又一方面中,分析诊断方法以及治疗剂的临床试验结果,使得诊断结果可以用于预测哪些患者将从该治疗中受益。在本发明的另一方面中,癌细胞抗体或其片段源自成像剂,然后将该组合物给予于患者而使患者体内的反应性肿瘤可视化。以此方式,抗体加上显像剂可以用于诊断癌症,评价疗法治疗的反应或评价对疗法治疗的反应,其中治疗剂靶向muc1*并可以包含用于诊断的一些或全部癌细胞抗体。在本发明的一方面中,附着于成像剂的抗体是骆驼科动物抗体,包括但不限于,美洲驼,羊驼和骆驼。
[0215]
本文中描述的诊断测试可以用于可以是取自测试对象、患者或作为对照的正常人的组织、活检样本、细胞或体液的样品。诊断测试可以在体外或体内进行。在手术中可以使用该诊断测试(例如,在不从受试者去除组织的情况下可以研究手术部位的组织)。以这种方式,该诊断测试会指导外科医生去除所有可检测到的muc1*阳性组织,无论这些组织看起来是否是肿瘤的一部分。在任一这些研究中,肿瘤发生或肿瘤发生潜力的主要指标是抗psmgfr抗体或癌细胞抗体可触及的细胞或组织表面处的muc1*量。通过扩展,暴露的癌细胞抗体结合表位是指muc1*的psmgfr区也可以触及与结合并激活由muc1*生长因子受体介导的生长和存活功能的生长因子。在另一种技术中,muc1*区和串联重复序列ibr或ur的抗体可以暴露于样品中,并确定muc1*的结合与全长muc1的比率。健康的样品几乎不或不表现出
抗体结合muc1*区。指示肿瘤发生的样品将显示出抗muc1*抗体与抗串联重复序列抗体或抗ibr抗体的非零比率,其中随着癌症分期/级别的增加,muc1*与含有串联重复序列的muc1、ibr或ur的比率就会增加。
[0216]
除了在细胞和组织上检测一定量的muc1*或含muc1的串联重复序列之外,还可以检测体液如血液、乳汁或分泌物、尿液、肺外排物等中从组织中脱落的含有串联重复序列的muc1的部分。在这些情况下,通过使用抗体,包括但不限于,结合串联重复序列的抗体,ibr的n端的独特区域或ibr本身测量脱落muc1的量,推断出muc1剪切成跨膜muc1*的水平。
[0217]
测量或推断细胞或组织上的muc1*的量,这会大于正常组织或患者的以前样品,是潜在的肿瘤形成、肿瘤存在或肿瘤进展的指标,而因此可以用作诊断和/或患者癌症治疗效果的评价因子。在一个方面中,通过使组织样品与抗muc1*抗体接触并确定muc1*的量大于正常组织或健康人中表达的量而测定muc1*的量。
[0218]
序列表自由文本
[0219]
在下面的抗体序列中,带下划线的序列是指cdr序列,且带双下划线的区域是指框架区。
[0220]
全长muc1受体(黏蛋白1前体,genbank登录号:p15941
[0221][0222]
截短的muc1受体亚型,在其n端具有nat-psmgfr和psibr,并包括在翻译后和受体在细胞表面表达之前可以被剪切的全长muc1受体的跨膜和细胞质序列:
[0223][0224]
截短的muc1受体亚型,在其n端具有nat-psmgfr+psibr+独特区并包括全长muc1受体的跨膜和细胞质序列:
[0225][0226]
psmgfr
[0227][0228]
psmgfr n+20/c-22
[0229][0230]
psmgfr n+12/c-22
[0231][0232]
psmgfr n+9/c-30
[0233][0234]
psmgfr n+20/c-41
[0235][0236]
psmgfr n+20/c-27
[0237][0238]
psmgfr n+9/c-9
[0239][0240]
抗体17h6重链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0241][0242]
抗体17h6重链-信号肽-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0243][0244]
抗体17h6轻链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0245][0246]
抗体17h6轻链-信号肽-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0247][0248]
抗体17h6重链cdr1
[0249][0250]
抗体17h6重链cdr1
[0251][0252]
抗体17h6重链cdr2
[0253][0254]
抗体17h6重链cdr2
[0255][0256]
抗体17h6重链cdr3
[0257][0258]
抗体17h6重链cdr3
[0259][0260]
抗体17h6轻链cdr1
[0261][0262]
抗体17h6轻链cdr1
[0263][0264]
抗体17h6轻链cdr2
[0265][0266]
抗体17h6轻链cdr2
[0267][0268]
抗体17h6轻链cdr3
[0269][0270]
抗体17h6轻链cdr3
[0271][0272]
抗体39h5重链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0273][0274]
抗体39h5重链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0275][0276]
抗体39h5轻链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0277][0278]
抗体39h5轻链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0279][0280]
抗体39h5重链cdr1
[0281][0282]
抗体39h5重链cdr1
[0283][0284]
抗体39h5重链cdr2
[0285][0286]
抗体39h5重链cdr2
[0287][0288]
抗体39h5重链cdr3
[0289][0290]
抗体39h5重链cdr3
[0291][0292]
抗体39h5轻链cdr1
[0293][0294]
抗体39h5轻链cdr1
[0295][0296]
抗体39h5轻链cdr2
[0297][0298]
抗体39h5轻链cdr2
[0299]
[0300]
抗体39h5轻链cdr3
[0301][0302]
抗体39h5轻链cdr3
[0303][0304]
抗体3c5重链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0305][0306]
抗体3c5重链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0307][0308]
抗体3c5轻链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0309][0310]
抗体3c5轻链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0311][0312]
抗体3c5重链cdr1
[0313][0314]
抗体3c5重链cdr1
[0315][0316]
抗体3c5重链cdr2
[0317][0318]
抗体3c5重链cdr2
[0319][0320]
抗体3c5重链cdr3
[0321][0322]
抗体3c5重链cdr3
[0323][0324]
抗体3c5轻链cdr1
[0325][0326]
抗体3c5轻链cdr1
[0327][0328]
抗体3c5轻链cdr2
[0329][0330]
抗体3c5轻链cdr2
[0331][0332]
抗体3c5轻链cdr3
[0333][0334]
抗体3c5轻链cdr3
[0335][0336]
抗体8a9重链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0337][0338]
抗体8a9重链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0339][0340]
抗体8a9轻链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0341][0342]
抗体8a9轻链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0343][0344]
抗体8a9重链cdr1
[0345][0346]
抗体8a9重链cdr1
[0347][0348]
抗体8a9重链cdr2
[0349][0350]
抗体8a9重链cdr2
[0351][0352]
抗体8a9重链cdr3
[0353]
[0354]
抗体8a9重链cdr3
[0355][0356]
抗体8a9轻链cdr1
[0357][0358]
抗体8a9轻链cdr1
[0359][0360]
抗体8a9轻链cdr2
[0361][0362]
抗体8a9轻链cdr2
[0363][0364]
抗体8a9轻链cdr3
[0365][0366]
抗体8a9轻链cdr3
[0367][0368]
抗体18g12重链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0369][0370]
抗体18g12重链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0371][0372]
抗体18g12轻链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0373][0374]
抗体18g12轻链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0375][0376]
抗体18g12重链cdr1
[0377][0378]
抗体18g12重链cdr1
[0379][0380]
抗体18g12重链cdr2
[0381]
[0382]
抗体18g12重链cdr2
[0383][0384]
抗体18g12重链cdr3
[0385][0386]
抗体18g12重链cdr3
[0387][0388]
抗体18g12轻链cdr1
[0389][0390]
抗体18g12轻链cdr1
[0391][0392]
抗体18g12轻链cdr2
[0393][0394]
抗体18g12轻链cdr2
[0395][0396]
抗体18g12轻链cdr3
[0397][0398]
抗体18g12轻链cdr3
[0399][0400]
抗体20a10重链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0401][0402]
抗体20a10重链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0403][0404]
抗体20a10轻链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0405][0406]
抗体20a10轻链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0407][0408]
抗体20a10重链cdr1
[0409][0410]
抗体20a10重链cdr1
[0411][0412]
抗体20a10重链cdr2
[0413][0414]
抗体20a10重链cdr2
[0415][0416]
抗体20a10重链cdr3
[0417][0418]
抗体20a10重链cdr3
[0419][0420]
抗体20a10轻链cdr1
[0421][0422]
抗体20a10轻链cdr1
[0423][0424]
抗体20a10轻链cdr2
[0425][0426]
抗体20a10轻链cdr2
[0427][0428]
抗体20a10轻链cdr3
[0429][0430]
抗体20a10轻链cdr3
[0431][0432]
抗体25e6重链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0433][0434]
抗体25e6重链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0435][0436]
抗体25e6轻链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0437][0438]
抗体25e6轻链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0439][0440]
抗体25e6重链cdr1
[0441][0442]
抗体25e6重链cdr1
[0443][0444]
抗体25e6重链cdr2
[0445][0446]
抗体25e6重链cdr2
[0447][0448]
抗体25e6重链cdr3
[0449][0450]
抗体25e6重链cdr3
[0451][0452]
抗体25e6轻链cdr1
[0453][0454]
抗体25e6轻链cdr1
[0455][0456]
抗体25e6轻链cdr2
[0457][0458]
抗体25e6轻链cdr2
[0459][0460]
抗体25e6轻链cdr3
[0461][0462]
抗体25e6轻链cdr3
[0463][0464]
抗体28f9重链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0465][0466]
抗体28f9重链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0467][0468]
抗体28f9轻链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0469][0470]
抗体28f9轻链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0471][0472]
抗体28f9重链cdr1
[0473][0474]
抗体28f9重链cdr1
[0475][0476]
抗体28f9重链cdr2
[0477][0478]
抗体28f9重链cdr2
[0479][0480]
抗体28f9重链cdr3
[0481][0482]
抗体28f9重链cdr3
[0483][0484]
抗体28f9轻链cdr1
[0485][0486]
抗体28f9轻链cdr1
[0487][0488]
抗体28f9轻链cdr2
[0489][0490]
抗体28f9轻链cdr2
[0491][0492]
抗体28f9轻链cdr3
[0493][0494]
抗体28f9轻链cdr3
[0495][0496]
抗体18b4重链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0497][0498]
抗体18b4重链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0499][0500]
抗体18b4轻链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0501][0502]
抗体18b4重链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0503][0504]
抗体18b4重链cdr1
[0505][0506]
抗体18b4重链cdr1
[0507][0508]
抗体18b4重链cdr2
[0509][0510]
抗体18b4重链cdr2
[0511][0512]
抗体18b4重链cdr3
[0513][0514]
抗体18b4重链cdr3
[0515][0516]
抗体18b4轻链cdr1
[0517][0518]
抗体18b4轻链cdr1
[0519]
[0520]
抗体18b4轻链cdr2
[0521][0522]
抗体18b4轻链cdr2
[0523][0524]
抗体18b4轻链cdr3
[0525][0526]
抗体18b4轻链cdr3
[0527][0528]
抗体1e4重链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0529][0530]
抗体1e4重链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0531][0532]
抗体1e4轻链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0533][0534]
抗体1e4轻链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0535][0536]
抗体1e4重链cdr1
[0537][0538]
抗体1e4重链cdr1
[0539][0540]
抗体1e4重链cdr2
[0541][0542]
抗体1e4重链cdr2
[0543][0544]
抗体1e4重链cdr3
[0545][0546]
抗体1e4重链cdr3
[0547]
[0548]
抗体1e4轻链cdr1
[0549][0550]
抗体1e4轻链cdr1
[0551][0552]
抗体1e4轻链cdr2
[0553][0554]
抗体1e4轻链cdr2
[0555][0556]
抗体1e4轻链cdr3
[0557][0558]
抗体1e4轻链cdr3
[0559][0560]
抗体29h1重链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0561][0562]
抗体29h1重链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0563][0564]
抗体29h1轻链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0565][0566]
抗体29h1轻链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0567][0568]
抗体29h1重链cdr1
[0569][0570]
抗体29h1重链cdr1
[0571][0572]
抗体29h1重链cdr2
[0573][0574]
抗体29h1重链cdr2
[0575][0576]
抗体29h1重链cdr3
[0577][0578]
抗体29h1重链cdr3
[0579][0580]
抗体29h1轻链cdr1
[0581][0582]
抗体29h1轻链cdr1
[0583][0584]
抗体29h1轻链cdr2
[0585][0586]
抗体29h1轻链cdr2
[0587][0588]
抗体29h1轻链cdr3
[0589][0590]
抗体29h1轻链cdr3
[0591][0592]
抗体31a1重链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0593][0594]
抗体31a1重链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0595][0596]
抗体31a1轻链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0597][0598]
抗体31a1轻链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0599][0600]
抗体31a1重链cdr1
[0601][0602]
抗体31a1重链cdr1
[0603][0604]
抗体31a1重链cdr2
[0605][0606]
抗体31a1重链cdr2
[0607][0608]
抗体31a1重链cdr3
[0609][0610]
抗体31a1重链cdr3
[0611][0612]
抗体31a1轻链cdr1
[0613][0614]
抗体31a1轻链cdr1
[0615][0616]
抗体31a1轻链cdr2
[0617][0618]
抗体31a1轻链cdr2
[0619][0620]
抗体31a1轻链cdr3
[0621][0622]
抗体31a1轻链cdr3
[0623][0624]
抗体32c1重链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0625][0626]
抗体32c1重链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0627][0628]
抗体32c1轻链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0629][0630]
抗体32c1轻链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0631][0632]
抗体32c1重链cdr1
[0633][0634]
抗体32c1重链cdr1
[0635][0636]
抗体32c1重链cdr2
[0637][0638]
抗体32c1重链cdr2
[0639][0640]
抗体32c1重链cdr3
[0641][0642]
抗体32c1重链cdr3
[0643][0644]
抗体32c1轻链cdr1
[0645][0646]
抗体32c1轻链cdr1
[0647][0648]
抗体32c1轻链cdr2
[0649][0650]
抗体32c1轻链cdr2
[0651][0652]
抗体32c1轻链cdr3
[0653][0654]
抗体32c1轻链cdr3
[0655][0656]
抗体45c11重链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0657][0658]
抗体45c11重链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0659]
[0660]
抗体45c11轻链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0661][0662]
抗体45c11轻链-信号序列-fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4
[0663][0664]
抗体45c11重链cdr1
[0665][0666]
抗体45c11重链cdr1
[0667][0668]
抗体45c11重链cdr2
[0669][0670]
抗体45c11重链cdr2
[0671][0672]
抗体45c11重链cdr3
[0673][0674]
抗体45c11重链cdr3
[0675][0676]
抗体45c11轻链cdr1
[0677][0678]
抗体45c11轻链cdr1
[0679][0680]
抗体45c11轻链cdr2
[0681][0682]
抗体45c11轻链cdr2
[0683][0684]
抗体45c11轻链cdr3
[0685][0686]
抗体45c11轻链cdr3
[0687][0688]
串联重复结构域肽
[0689][0690]
car44:cd8/humnc2/cd8/4—1bb/cd3
[0691][0692]
min-a2-1轻链可变区氨基酸序列
[0693][0694]
min-a2-2轻链可变区氨基酸序列
[0695][0696]
min-c9-1轻链可变区氨基酸序列
[0697][0698]
min-c9-2轻链可变区氨基酸序列
[0699][0700]
min-d7-1轻链可变区氨基酸序列
[0701][0702]
min-d7-2轻链可变区氨基酸序列
[0703][0704]
min-f2-1轻链可变区氨基酸序列
[0705][0706]
min-f2-2轻链可变区氨基酸序列
[0707][0708]
min-a2-1重链可变区氨基酸序列
[0709][0710]
min-a2-2重链可变区氨基酸序列
[0711][0712]
min-c9-1重链可变区氨基酸序列
[0713][0714]
min-c9-2重链可变区氨基酸序列
[0715][0716]
min-d7-1重链可变区氨基酸序列
[0717][0718]
min-d7-2重链可变区氨基酸序列
[0719][0720]
min-f2-1重链可变区氨基酸序列
[0721][0722]
min-f2-2重链可变区氨基酸序列
[0723][0724]
min-f2-3重链可变区氨基酸序列
[0725][0726]
min-f2-4重链可变区氨基酸序列
[0727][0728]
min-14轻链可变区氨基酸序列
[0729][0730]
min-17-1轻链可变区氨基酸序列
[0731][0732]
min-17-2轻链可变区氨基酸序列
[0733][0734]
min-29轻链可变区氨基酸序列
[0735][0736]
min-34轻链可变区氨基酸序列
[0737][0738]
min-42轻链可变区氨基酸序列
[0739][0740]
min-45轻链可变区氨基酸序列
[0741][0742]
min-14重链可变区氨基酸序列
[0743][0744]
min-17-2重链可变区氨基酸序列
[0745][0746]
min-17-1重链可变区氨基酸序列
[0747][0748]
min-29重链可变区氨基酸序列
[0749][0750]
min-34重链可变区氨基酸序列
[0751][0752]
min-42重链可变区氨基酸序列
[0753][0754]
min-45重链可变区氨基酸序列
[0755][0756]
描述min-a2-1轻链可变互补决定区1(cdr1)氨基酸序列。
[0757]
描述min-a2-2轻链可变互补决定区1(cdr1)氨基酸序列。
[0758]
描述min-c9-1轻链可变互补决定区1(cdr1)氨基酸序列。
[0759]
描述min-c9-2轻链可变互补决定区1(cdr1)氨基酸序列。
[0760]
描述min-d7-1轻链可变互补决定区1(cdr1)氨基酸序列。
[0761]
描述min-d7-2轻链可变互补决定区1(cdr1)氨基酸序列。
[0762]
描述min-f2-1轻链可变互补决定区1(cdr1)氨基酸序列。
[0763]
描述min-f2-2轻链可变互补决定区1(cdr1)氨基酸序列。
[0764]
描述min-a2-1轻链可变互补决定区2(cdr2)氨基酸序列。
[0765]
描述min-a2-2轻链可变互补决定区2(cdr2)氨基酸序列。
[0766]
描述min-c9-1轻链可变互补决定区2(cdr2)氨基酸序列。
[0767]
描述min-c9-2轻链可变互补决定区2(cdr2)氨基酸序列。
[0768]
描述min-d7-1轻链可变互补决定区2(cdr2)氨基酸序列。
[0769]
描述min-d7-2轻链可变互补决定区2(cdr2)氨基酸序列。
[0770]
描述min-f2-1轻链可变互补决定区2(cdr2)氨基酸序列。
[0771]
描述min-f2-2轻链可变互补决定区2(cdr2)氨基酸序列。
[0772]
描述min-a2-1轻链可变互补决定区3(cdr3)氨基酸序列。
[0773]
描述min-a2-2轻链可变互补决定区3(cdr3)氨基酸序列。
[0774]
描述min-c9-1轻链可变互补决定区3(cdr3)氨基酸序列。
[0775]
描述min-c9-2轻链可变互补决定区3(cdr3)氨基酸序列。
[0776]
描述min-d7-1轻链可变互补决定区3(cdr3)氨基酸序列。
[0777]
描述min-d7-2轻链可变互补决定区3(cdr3)氨基酸序列。
[0778]
描述min-f2-1轻链可变互补决定区3(cdr3)氨基酸序列。
[0779]
描述min-f2-2轻链可变互补决定区3(cdr3)氨基酸序列。
[0780]
描述min-a2-1轻链可变互补决定区1(cdr1)氨基酸序列。
[0781]
描述min-a2-2重链可变互补决定区1(cdr1)氨基酸序列。
[0782]
描述min-c9-1重链可变互补决定区1(cdr1)氨基酸序列。
[0783]
描述min-c9-2重链可变互补决定区1(cdr1)氨基酸序列。
[0784]
描述min-d7-1重链可变互补决定区1(cdr1)氨基酸序列。
[0785]
描述min-d7-2重链可变互补决定区1(cdr1)氨基酸序列。
[0786]
描述min-f2-1重链可变互补决定区1(cdr1)氨基酸序列。
[0787]
描述min-f2-2重链可变互补决定区1(cdr1)氨基酸序列。
[0788]
描述min-f2-3重链可变互补决定区1(cdr1)氨基酸序列。
[0789]
描述min-f2-4重链可变互补决定区1(cdr1)氨基酸序列。
[0790]
描述min-a2-1重链可变互补决定区2(cdr2)氨基酸序列。
[0791]
描述min-a2-2重链可变互补决定区2(cdr2)氨基酸序列。
[0792]
描述min-c9-1重链可变互补决定区2(cdr2)氨基酸序列。
[0793]
描述min-c9-2重链可变互补决定区2(cdr2)氨基酸序列。
[0794]
描述min-d7-1重链可变决定区2(cdr2)氨基酸序列。
[0795]
描述min-d7-2重链可变互补决定区2(cdr2)氨基酸序列。
[0796]
描述min-f2-1重链可变互补决定区2(cdr2)氨基酸序列。
[0797]
描述min-f2-2重链可变互补决定区2(cdr2)氨基酸序列。
[0798]
描述min-f2-3重链可变互补决定区2(cdr2)氨基酸序列。
[0799]
描述min-f2-4重链可变互补决定区2(cdr2)氨基酸序列。
[0800]
描述min-a2-1重链可变互补决定区3(cdr3)氨基酸序列。
[0801]
描述min-a2-2重链可变互补决定区3(cdr3)氨基酸序列。
[0802]
描述min-c9-1重链可变互补决定区3(cdr3)氨基酸序列。
[0803]
描述min-c9-2重链可变互补决定区3(cdr3)氨基酸序列。
[0804]
描述min-d7-1重链可变互补决定区3(cdr3)氨基酸序列。
[0805]
描述min-d7-2重链可变互补决定区3(cdr3)氨基酸序列。
[0806]
描述min-f2-1重链可变互补决定区3(cdr3)氨基酸序列。
[0807]
描述min-f2-2重链可变互补决定区3(cdr3)氨基酸序列。
[0808]
描述min-f2-3重链可变互补决定区3(cdr3)氨基酸序列。
[0809]
描述min-f2-4重链可变互补决定区3(cdr3)氨基酸序列。
[0810]
描述min-14轻链可变互补性确定区域1(cdr1)氨基酸序列。
[0811]
描述min-17-1轻链可变互补决定区1(cdr1)氨基酸序列。
[0812]
描述min-17-2轻链可变互补决定区1(cdr1)氨基酸序列。
[0813]
描述min-29轻链可变互补决定区1(cdr1)氨基酸序列。
[0814]
描述min-34轻链可变互补决定区1(cdr1)氨基酸序列。
[0815]
描述min-42轻链可变互补性确定区域1(cdr1)氨基酸序列。
[0816]
描述min-45轻链可变互补决定区1(cdr1)氨基酸序列。
[0817]
描述min-14轻链可变互补决定区2(cdr2)氨基酸序列。
[0818]
描述min-17-1轻链可变互补决定区2(cdr2)氨基酸序列。
[0819]
描述min-17-2轻链可变互补决定区2(cdr2)氨基酸序列。
[0820]
描述min-29轻链可变互补决定区2(cdr2)氨基酸序列。
[0821]
描述min-34轻链可变互补决定区2(cdr2)氨基酸序列。
[0822]
描述min-42轻链可变互补决定区2(cdr2)氨基酸序列。
[0823]
描述min-45轻链可变互补决定区2(cdr2)氨基酸序列。
[0824]
描述min-14重链互补性确定区域1(cdr1)氨基酸序列。
[0825]
描述min-17-1重链互补决定区1(cdr1)氨基酸序列。
[0826]
描述min-17-2重链互补决定区1(cdr1)氨基酸序列。
[0827]
描述min-34重链互补决定区1(cdr1)氨基酸序列。
[0828]
描述min-42重链互补决定区1(cdr1)氨基酸序列。
[0829]
描述min-45重链互补决定区1(cdr1)氨基酸序列。
[0830]
描述min-14重链互补决定区2(cdr2)氨基酸序列。
[0831]
描述min-17-1重链互补决定区2(cdr2)氨基酸序列。
[0832]
描述min-17-2重链互补决定区2(cdr2)氨基酸序列。
[0833]
描述min-29重链互补决定区2(cdr2)氨基酸序列。
[0834]
描述min-34重链互补决定区2(cdr2)氨基酸序列。
[0835]
描述min-42重链互补决定区2(cdr2)氨基酸序列。
[0836]
描述min-45重链互补决定区2(cdr2)氨基酸序列。
[0837]
人源化e6重链可变区序列:
[0838][0839]
人源化e6重链可变互补决定区1(cdr1)序列:
[0840][0841]
人源化e6重链可变互补决定区2(cdr2)序列:
[0842][0843]
人源化e6重链可变互补决定区3(cdr3)序列:
[0844][0845]
人源化e6轻链可变区序列:
[0846][0847]
人源化e6轻链可变互补决定区1(cdr1)序列:
[0848][0849]
人源化e6轻链可变互补决定区2(cdr2)序列:
[0850][0851]
人源化e6轻链可变互补决定区3(cdr3)序列:
[0852][0853]
人源化c2重链可变区域序列:
[0854][0855]
人源化c2重链可变互补决定区1(cdr1)序列:
[0856][0857]
人源化c2重链可变互补决定区2(cdr2)序列:
[0858][0859]
人源化c2重链可变互补决定区3(cdr3)序列:
[0860][0861]
人源化c2轻链可变区域序列:
[0862][0863]
人源化c2轻链可变互补决定区1(cdr1)序列:
[0864][0865]
人源化c2轻链可变互补决定区2(cdr2)序列:
[0866]
人源化c2轻链可变互补决定区3(cdr3)序列:
[0867][0868]
人源化c2轻链可变互补决定区3(cdr3)序列:
[0869][0870]
psectag2 c2 scfv-fc
[0871][0872]
psectag2 e6 scfv-fc
[0873][0874]
人源化c2 scfv(vh-vl)序列:
[0875][0876]
人源化e6 scfv(vl-vh)序列:
[0877][0878]
人源化c3重链可变区序列:
[0879][0880]
人源化c3重链可变互补决定区1(cdr1)序列:
[0881][0882]
人源化c3重链可变互补决定区2(cdr2)序列:
[0883][0884]
人源化c3重链可变互补决定区3(cdr3)序列:
[0885][0886]
人源化c3轻链可变区域序列:
[0887][0888]
人源化c3轻链可变互补决定区1(cdr1)序列:
[0889][0890]
人源化c3轻链可变互补决定区2(cdr2)序列:
[0891][0892]
人源化c3轻链可变互补决定区3(cdr3)序列:
[0893]
[0894]
人源化c8重链可变区序列:
[0895][0896]
人源化c8重链可变互补决定区1(cdr1)序列:
[0897][0898]
人源化c8重链可变互补决定区2(cdr2)序列:
[0899][0900]
人源化c8重链可变互补决定区3(cdr3)序列:
[0901][0902]
人源化c8轻链可变区序列
[0903][0904]
人源化c8轻链可变互补决定区1(cdr1)序列:
[0905][0906]
人源化c8轻链可变互补决定区2(cdr2)序列:
[0907][0908]
人源化c8轻链可变互补决定区3(cdr3)序列:
[0909][0910]
本文引述的所有参考文献通过引用整体结合于本文中。
[0911]
*****
[0912]
本领域技术人员仅使用常规实验将会认识到或可以确定本文具体描述的本发明的具体实施方式的许多等同方案。
起点商标作为专业知识产权交易平台,可以帮助大家解决很多问题,如果大家想要了解更多知产交易信息请点击 【在线咨询】或添加微信 【19522093243】 与客服一对一沟通,为大家解决相关问题。
与客服一对一沟通,为大家解决相关问题。
此文章来源于网络,如有侵权,请联系删除
相关标签:
 热门咨询
热门咨询

tips


 商标分类
商标分类  商标转让
商标转让 



